Υπολογιστική βιολογία
Η υπολογιστική βιολογία αναφέρεται στη χρήση της ανάλυσης δεδομένων, της μαθηματικής μοντελοποίησης και των υπολογιστικών προσομοιώσεων για την κατανόηση των βιολογικών συστημάτων και σχέσεων.[1]
Στο σταυροδρόμι της επιστήμης των υπολογιστών, της βιολογίας και των μεγάλων δεδομένων, ο τομέας αυτός αντλεί επίσης από τα εφαρμοσμένα μαθηματικά, τη χημεία και τη γενετική[2]. Διαφέρει από τη βιολογική πληροφορική, ένα υποπεδίο της επιστήμης των υπολογιστών και της μηχανικής που χρησιμοποιεί τη βιομηχανική για την κατασκευή υπολογιστών.
Ιστορία[Επεξεργασία | επεξεργασία κώδικα]
Η βιοπληροφορική, η ανάλυση των διαδικασιών πληροφορικής στα βιολογικά συστήματα, ξεκίνησε στις αρχές της δεκαετίας του 1970. Εκείνη την εποχή, η έρευνα στην τεχνητή νοημοσύνη χρησιμοποιούσε δικτυακά μοντέλα του ανθρώπινου εγκεφάλου προκειμένου να δημιουργήσει νέους αλγορίθμους. Αυτή η χρήση βιολογικών δεδομένων ώθησε τους βιολογικούς ερευνητές να χρησιμοποιήσουν υπολογιστές για την αξιολόγηση και τη σύγκριση μεγάλων συνόλων δεδομένων στον τομέα τους[3] .
Μέχρι το 1982, οι ερευνητές μοιράζονταν πληροφορίες μέσω διάτρητων καρτών. Ο όγκος των δεδομένων αυξήθηκε εκθετικά μέχρι το τέλος της δεκαετίας του 1980, απαιτώντας νέες υπολογιστικές μεθόδους για τη γρήγορη ερμηνεία των σχετικών πληροφοριών[3].
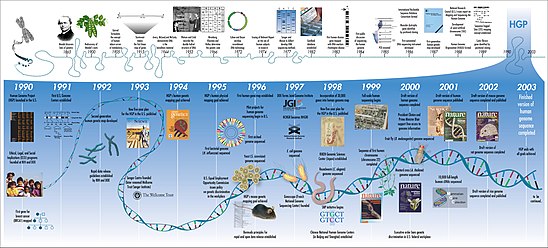
Ισως το πιο γνωστό παράδειγμα υπολογιστικής βιολογίας, το Πρόγραμμα Ανθρώπινου Γονιδιώματος, ξεκίνησε επίσημα το 1990[4]. Μέχρι το 2003, το πρόγραμμα είχε χαρτογραφήσει περίπου το 85% του ανθρώπινου γονιδιώματος, ικανοποιώντας τους αρχικούς του στόχους[5]. Οι εργασίες συνεχίστηκαν, ωστόσο, και μέχρι το 2021 είχε επιτευχθεί επίπεδο "πλήρους γονιδιώματος" με μόνο το 0,3% των υπόλοιπων βάσεων να καλύπτονται από πιθανά ζητήματα.[6][7] Το χρωμόσωμα Υ που έλειπε προστέθηκε τον Ιανουάριο του 2022.
Από τα τέλη της δεκαετίας του 1990, η υπολογιστική βιολογία αποτελεί σημαντικό μέρος της βιολογίας, οδηγώντας σε πολυάριθμα υποπεδία[8]. Σήμερα, η Διεθνής Εταιρεία Υπολογιστικής Βιολογίας αναγνωρίζει 21 διαφορετικές "Κοινότητες Ειδικού Ενδιαφέροντος", καθεμία από τις οποίες αντιπροσωπεύει ένα κομμάτι του ευρύτερου πεδίου[9]. Εκτός από τη βοήθεια στην αλληλουχία του ανθρώπινου γονιδιώματος, η υπολογιστική βιολογία βοήθησε στη δημιουργία αξιόπιστων μοντέλων του ανθρώπινου εγκεφάλου, στη χαρτογράφηση της τρισδιάστατης δομής των γονιδιωμάτων και στη μοντελοποίηση βιολογικών συστημάτων[3].
Εφαρμογές[Επεξεργασία | επεξεργασία κώδικα]
Ανατομία[Επεξεργασία | επεξεργασία κώδικα]
Η υπολογιστική ανατομία είναι η μελέτη του ανατομικού σχήματος και της μορφής στην ορατή ή ακατέργαστη ανατομική κλίμακα μορφολογίας. Περιλαμβάνει την ανάπτυξη υπολογιστικών μαθηματικών και δεδομένων-αναλυτικών μεθόδων για τη διαμόρφωση και προσομοίωση βιολογικών δομών. Επικεντρώνεται στις ανατομικές δομές που απεικονίζονται και όχι στις συσκευές ιατρικής απεικόνισης. Λόγω της διαθεσιμότητας πυκνών τρισδιάστατων μετρήσεων μέσω τεχνολογιών όπως η απεικόνιση μαγνητικού συντονισμού, η υπολογιστική ανατομία έχει αναδειχθεί ως υποπεδίο της ιατρικής απεικόνισης και της βιομηχανικής για την εξαγωγή ανατομικών συστημάτων συντεταγμένων σε κλίμακα μορφής σε 3D.
Η αρχική διατύπωση της υπολογιστικής ανατομίας είναι ως ένα παραγωγικό πρότυπο σχήματος και μορφής από υποδείγματα που επιδρούν μέσω μετασχηματισμών[10]. Η ομάδα του διαφιομορφισμού χρησιμοποιείται για τη μελέτη διαφορετικών συστημάτων συντεταγμένων μέσω μετασχηματισμών συντεταγμένων, όπως δημιουργούνται μέσω των ταχυτήτων Λαγκράνζ και Όιλερ της ροής από μια ανατομική διαμόρφωση στο σε μια άλλη. Σχετίζεται με τη στατιστική του σχήματος και τη μορφομετρία, με τη διαφορά ότι οι διαφιομορφισμοί χρησιμοποιούνται για τη χαρτογράφηση συστημάτων συντεταγμένων, η μελέτη των οποίων είναι γνωστή ως διαφιομορφομετρία.
Δεδομένα και διαμόρφωση προτύπων[Επεξεργασία | επεξεργασία κώδικα]
Κύριο άρθρο: Βιοπληροφορική
Η μαθηματική βιολογία είναι η χρήση μαθηματικών προτύπων ζωντανών οργανισμών για την εξέταση των συστημάτων που διέπουν τη δομή, την ανάπτυξη και τη συμπεριφορά των βιολογικών συστημάτων. Αυτό συνεπάγεται μια πιο θεωρητική προσέγγιση των προβλημάτων, σε αντίθεση με την πειραματική βιολογία, η οποία έχει περισσότερο εμπειρικό πνεύμα [11] . Η μαθηματική βιολογία βασίζεται στα διακριτά μαθηματικά, την τοπολογία (επίσης χρήσιμη για τη διαμόρφωση μοντέλων σε υπολογιστές), τη στατιστική κατά Μπέιζ, τη γραμμική άλγεβρα και την Άλγεβρα Μπουλ[12].
Αυτές οι μαθηματικές προσεγγίσεις επέτρεψαν τη δημιουργία βάσεων δεδομένων και άλλων μεθόδων για την αποθήκευση, την εξαγωγή και την ανάλυση βιολογικών δεδομένων, ένα πεδίο γνωστό ως βιοπληροφορική. Σε γενικές γραμμές, η διαδικασία αυτή περιλαμβάνει τη γενετική και την ανάλυση γονιδίων.
Η συλλογή και ανάλυση μεγάλων συνόλων δεδομένων άνοιξε το δρόμο για την ανάπτυξη ερευνητικών τομέων όπως η εξόρυξη δεδομένων[12] και η υπολογιστική βιομοντελοποίηση, η οποία περιλαμβάνει τη δημιουργία υπολογιστικών προτύπων και οπτικών προσομοιώσεων βιολογικών συστημάτων, επιτρέποντας στους ερευνητές να προβλέψουν πώς αυτά τα συστήματα θα ανταποκριθούν σε διαφορετικά περιβάλλοντα. Αυτό δίνει τη δυνατότητα στους ερευνητές να προβλέψουν πώς αυτά τα συστήματα θα αντιδράσουν σε διαφορετικά περιβάλλοντα, κάτι που είναι χρήσιμο για τον προσδιορισμό του κατά πόσον ένα σύστημα μπορεί "να διατηρήσει την κατάσταση και τη λειτουργία του απέναντι σε εξωτερικές και εσωτερικές διαταραχές"[13]. Ενώ οι τρέχουσες τεχνικές επικεντρώνονται σε μικρά βιολογικά συστήματα, οι ερευνητές εργάζονται πάνω σε προσεγγίσεις που επιτρέπουν την ανάλυση και μοντελοποίηση μεγαλύτερων δικτύων. Η πλειονότητα των ερευνητών πιστεύει ότι αυτό θα είναι απαραίτητο για την ανάπτυξη σύγχρονων ιατρικών προσεγγίσεων για τη δημιουργία νέων φαρμάκων και γονιδιακών θεραπειών [13]. Μια χρήσιμη προσέγγιση μοντελοποίησης είναι η χρήση δικτύων Πέτρι μέσω εργαλείων όπως το esyN[14].
Σε παρόμοιες γραμμές, έως τις τελευταίες δεκαετίες, η θεωρητική οικολογία ασχολήθηκε σε μεγάλο βαθμό με αναλυτικά πρότυπα που ήταν αποκομμένα από τα στατιστικά μοντέλα που χρησιμοποιούσαν οι εμπειρικοί οικολόγοι. Ωστόσο, οι υπολογιστικές μέθοδοι βοήθησαν στην ανάπτυξη της οικολογικής θεωρίας μέσω της προσομοίωσης οικολογικών συστημάτων, εκτός από την αυξανόμενη εφαρμογή μεθόδων από την υπολογιστική στατιστική στις οικολογικές αναλύσεις.
Βιολογία συστημάτων[Επεξεργασία | επεξεργασία κώδικα]
Κύριο άρθρο: Βιολογία συστημάτων
Η Βιολογία συστημάτων συνίσταται στον υπολογισμό των αλληλεπιδράσεων μεταξύ διαφόρων βιολογικών συστημάτων που κυμαίνονται από το κυτταρικό επίπεδο έως ολόκληρους πληθυσμούς με στόχο την ανακάλυψη αναδυόμενων ιδιοτήτων. Η διαδικασία αυτή περιλαμβάνει συνήθως τη δικτύωση κυτταρικών σηματοδοτικών και μεταβολικών μονοπατιών. Η συστημική βιολογία χρησιμοποιεί συχνά υπολογιστικές τεχνικές από τη βιολογική μοντελοποίηση και τη θεωρία γραφημάτων για τη μελέτη αυτών των πολύπλοκων αλληλεπιδράσεων σε κυτταρικό επίπεδο[12].
Εξελικτική βιολογία[Επεξεργασία | επεξεργασία κώδικα]
Κύριο άρθρο: Εξελικτική βιολογία
Η υπολογιστική βιολογία βοήθησε την εξελικτική βιολογία ως εξής:
- Με τη χρήση δεδομένων DNA για την ανακατασκευή του δέντρου της ζωής με την υπολογιστική φυλογενετική
- Με την εφαρμογή μοντέλων πληθυσμιακής γενετικής (είτε προς τα εμπρός[15] είτε προς τα πίσω) σε δεδομένα DNA για την εξαγωγή συμπερασμάτων σχετικά με τη δημογραφική ή επιλεκτική ιστορία.
- Με την κατασκευή πληθυσμιακών γενετικών μοντέλων εξελικτικών συστημάτων από τις πρώτες αρχές προκειμένου να προβλεφθεί τι είναι πιθανό να εξελιχθεί.
Γονιδιωματική[Επεξεργασία | επεξεργασία κώδικα]

Η υπολογιστική γονιδιωματική είναι η μελέτη των γονιδιωμάτων των κυττάρων και των οργανισμών. Το Πρόγραμμα Ανθρώπινου Γονιδιώματος είναι ένα παράδειγμα υπολογιστικής γονιδιωματικής. Το έργο αυτό αποσκοπεί στην αλληλουχία ολόκληρου του ανθρώπινου γονιδιώματος σε ένα σύνολο δεδομένων. Μόλις υλοποιηθεί πλήρως, αυτό θα μπορούσε να επιτρέψει στους γιατρούς να αναλύσουν το γονιδίωμα ενός μεμονωμένου ασθενούς[16]. Αυτό ανοίγει τη δυνατότητα εξατομικευμένης ιατρικής, συνταγογραφώντας θεραπείες με βάση τα προϋπάρχοντα γενετικά πρότυπα ενός ατόμου. Οι ερευνητές επιδιώκουν την αλληλουχία των γονιδιωμάτων των ζώων, των φυτών, των βακτηρίων και όλων των άλλων ειδών ζωής[17].
Ένας από τους κύριους τρόπους με τους οποίους συγκρίνονται τα γονιδιώματα είναι η ομολογία αλληλουχιών. Η ομολογία είναι η μελέτη των βιολογικών δομών και των νουκλεοτιδικών αλληλουχιών σε διαφορετικούς οργανισμούς που προέρχονται από έναν κοινό πρόγονο. Έρευνες δείχνουν ότι μεταξύ 80 και 90% των γονιδίων σε πρόσφατα αλληλουχημένα προκαρυωτικά γονιδιώματα μπορούν να αναγνωριστούν με αυτόν τον τρόπο[17].
Η ευθυγράμμιση ακολουθιών είναι μια άλλη διαδικασία για τη σύγκριση και τον εντοπισμό ομοιοτήτων μεταξύ βιολογικών ακολουθιών ή γονιδίων. Η στοίχιση ακολουθιών είναι χρήσιμη σε πολλές εφαρμογές βιοπληροφορικής, όπως ο υπολογισμός της μεγαλύτερης κοινής υποακολουθίας δύο γονιδίων ή η σύγκριση παραλλαγών ορισμένων ασθενειών[18].
Ένα ανέγγιχτο έργο στην υπολογιστική γονιδιωματική είναι η ανάλυση των διαγονιδιακών περιοχών, οι οποίες αποτελούν περίπου το 97% του ανθρώπινου γονιδιώματος[17]. Οι ερευνητές εργάζονται για την κατανόηση των λειτουργιών των μη κωδικοποιητικών περιοχών του ανθρώπινου γονιδιώματος μέσω της ανάπτυξης υπολογιστικών και στατιστικών μεθόδων και μέσω μεγάλων κοινοπραξιών όπως το ENCODE και το Σχέδιο Επιγονιδιωματικής του Οδικού Χάρτη (Roadmap Epigenomics Project).
Η κατανόηση του τρόπου με τον οποίο τα μεμονωμένα γονίδια συμβάλλουν στη βιολογία ενός οργανισμού σε μοριακό, κυτταρικό και οργανικό επίπεδο είναι γνωστή ως γονιδιακή οντολογία. Η αποστολή της κοινοπραξίας γονιδιακής οντολογίας είναι να αναπτύξει ένα ολοκληρωμένο και ενημερωμένο υπολογιστικό μοντέλο βιολογικών συστημάτων, από το μοριακό επίπεδο μέσω των κυτταρικών και οργανωσιακών συστημάτων έως τα ευρύτερα μονοπάτια. Ο πόρος της Γονιδιακής Οντολογίας παρέχει μια υπολογιστική αναπαράσταση της τρέχουσας επιστημονικής γνώσης σχετικά με τις λειτουργίες των γονιδίων (ή, ακριβέστερα, των πρωτεϊνών και των μη κωδικών μορίων RNA που παράγονται από τα γονίδια) σε πολλούς διαφορετικούς οργανισμούς, από τον άνθρωπο έως τα βακτήρια[19].
Η τρισδιάστατη γονιδιωματική είναι ένα υποτμήμα της υπολογιστικής βιολογίας που επικεντρώνεται στην οργάνωση και την αλληλεπίδραση των γονιδίων μέσα σε ένα ευκαρυωτικό κύτταρο. Η χαρτογράφηση της αρχιτεκτονικής του γονιδιώματος (GAM) είναι μία από τις μεθόδους που χρησιμοποιούνται για τη συλλογή τρισδιάστατων γονιδιωματικών δεδομένων. Η GAM μετρά τις τρισδιάστατες αποστάσεις της χρωματίνης και του DNA στο γονιδίωμα συνδυάζοντας την κρυοτομή, η οποία περιλαμβάνει την αποκοπή μιας λωρίδας του πυρήνα για την εξέταση του DNA, και τη μικροδιατομή με λέιζερ. Ένα πυρηνικό προφίλ είναι απλώς μια λωρίδα ή φέτα που λαμβάνεται από τον πυρήνα. Κάθε πυρηνικό προφίλ περιέχει γονιδιωματικά παράθυρα, δηλαδή ορισμένες αλληλουχίες νουκλεοτιδίων, τη βασική μονάδα του DNA. Το GAM αποτυπώνει ένα γονιδιωματικό δίκτυο πολύπλοκων, πολλαπλών ενισχυτικών επαφών χρωματίνης σε ολόκληρο το κύτταρο[20].
Νευροεπιστήμη[Επεξεργασία | επεξεργασία κώδικα]
Κύριο άρθρο: Υπολογιστική νευροεπιστήμη
Η υπολογιστική νευροεπιστήμη είναι η μελέτη της λειτουργίας του εγκεφάλου από την άποψη των ιδιοτήτων επεξεργασίας πληροφοριών του νευρικού συστήματος. Ως υποσύνολο της νευροεπιστήμης, επιδιώκει να μοντελοποιήσει τον εγκέφαλο για να εξετάσει συγκεκριμένες πτυχές του νευρολογικού συστήματος.[21] Τα μοντέλα του εγκεφάλου περιλαμβάνουν:
- Ρεαλιστικά εγκεφαλικά πρότυπα: Τα πρότυπα αυτά επιδιώκουν να αναπαραστήσουν όλες τις πτυχές του εγκεφάλου, περιλαμβάνοντας όσο το δυνατόν περισσότερες λεπτομέρειες σε κυτταρικό επίπεδο. Τα ρεαλιστικά μοντέλα παρέχουν τις περισσότερες πληροφορίες σχετικά με τον εγκέφαλο, αλλά έχουν επίσης το μεγαλύτερο περιθώριο σφάλματος. Όσο περισσότερες μεταβλητές υπάρχουν σε ένα μοντέλο εγκεφάλου, τόσο μεγαλύτερο είναι το περιθώριο σφάλματος. Αυτά τα μοντέλα δεν λαμβάνουν υπόψη μέρη της κυτταρικής δομής που δεν γνωρίζουν οι επιστήμονες. Τα ρεαλιστικά μοντέλα εγκεφάλου είναι τα πιο εντατικά σε υπολογισμούς και τα πιο ακριβά στην υλοποίησή τους[22].
- Πρότυπα απλοποίησης του εγκεφάλου: Τα πρότυπα αυτά επιδιώκουν να περιορίσουν το πεδίο εφαρμογής ενός μοντέλου προκειμένου να αξιολογήσουν μια συγκεκριμένη φυσική ιδιότητα του νευρολογικού συστήματος. Με τον τρόπο αυτό επιλύονται υπολογιστικά εντατικά προβλήματα και μειώνεται η ποσότητα του δυνητικού σφάλματος σε ένα ρεαλιστικό μοντέλο εγκεφάλου[22].
Το έργο των υπολογιστικών νευροεπιστημόνων έγκειται στη βελτίωση των αλγορίθμων και των δομών δεδομένων που χρησιμοποιούνται σήμερα για την αύξηση της ταχύτητας αυτών των υπολογισμών.
Η υπολογιστική νευροψυχιατρική είναι ένας αναδυόμενος τομέας που χρησιμοποιεί μαθηματική και υπολογιστική μοντελοποίηση των μηχανισμών του εγκεφάλου που εμπλέκονται στις ψυχικές διαταραχές. Αρκετές πρωτοβουλίες υπέδειξαν ότι η υπολογιστική μοντελοποίηση αποτελεί σημαντική συμβολή στην κατανόηση των νευρωνικών κυκλωμάτων που μπορούν να δημιουργήσουν ψυχικές λειτουργίες και δυσλειτουργίες[23][24][25].
Φαρμακολογία[Επεξεργασία | επεξεργασία κώδικα]
Κύριο άρθρο: Φαρμακολογία
Η υπολογιστική φαρμακολογία είναι "η μελέτη των επιπτώσεων των γονιδιωματικών δεδομένων για την εύρεση συνδέσεων μεταξύ συγκεκριμένων γονότυπων και ασθενειών και στη συνέχεια η διαλογή δεδομένων φαρμάκων"[26]. Οι φαρμακολόγοι ήταν σε θέση να χρησιμοποιούν το Microsoft Excel για να συγκρίνουν χημικά και γονιδιωματικά δεδομένα που σχετίζονται με την αποτελεσματικότητα των φαρμάκων. Ωστόσο, η βιομηχανία έχει φτάσει σε αυτό που αναφέρεται ως το οδόφραγμα του Excel. Αυτό προκύπτει από τον περιορισμένο αριθμό κελιών που είναι προσβάσιμα σε ένα υπολογιστικό φύλλο. Αυτή η εξέλιξη οδήγησε στην ανάγκη για υπολογιστική φαρμακολογία. Οι επιστήμονες και οι ερευνητές αναπτύσσουν υπολογιστικές μεθόδους για την ανάλυση αυτών των μαζικών συνόλων δεδομένων. Αυτό επιτρέπει την αποτελεσματική σύγκριση μεταξύ των αξιοσημείωτων σημείων δεδομένων και επιτρέπει την ανάπτυξη ακριβέστερων φαρμάκων[27].
Οι αναλυτές προβλέπουν ότι αν τα κύρια φάρμακα αποτύχουν λόγω πατεντών, η υπολογιστική βιολογία θα είναι απαραίτητη για την αντικατάσταση των σημερινών φαρμάκων στην αγορά. Οι διδακτορικοί φοιτητές στην υπολογιστική βιολογία ενθαρρύνονται να ακολουθήσουν καριέρα στη βιομηχανία αντί να αναλάβουν μεταδιδακτορικές θέσεις. Αυτό είναι άμεσο αποτέλεσμα των μεγάλων φαρμακευτικών εταιρειών που χρειάζονται περισσότερους εξειδικευμένους αναλυτές των μεγάλων συνόλων δεδομένων που απαιτούνται για την παραγωγή νέων φαρμάκων[27].
Αντίστοιχα, η υπολογιστική ογκολογία αποσκοπεί στον προσδιορισμό των μελλοντικών μεταλλάξεων στον καρκίνο μέσω αλγοριθμικών προσεγγίσεων. Η έρευνα σε αυτόν τον τομέα οδήγησε στη χρήση μετρήσεων υψηλής απόδοσης που περιλαμβάνουν εκατομμύρια σημεία δεδομένων με τη χρήση ρομποτικής και άλλων αισθητήρων. Τα δεδομένα αυτά συλλέγονται από το DNA, το RNA και άλλες βιολογικές δομές. Οι τομείς εστίασης περιλαμβάνουν τον προσδιορισμό των χαρακτηριστικών των όγκων, την ανάλυση μορίων που είναι ντετερμινιστικά στην πρόκληση καρκίνου και την κατανόηση του τρόπου με τον οποίο το ανθρώπινο γονιδίωμα σχετίζεται με την πρόκληση όγκων και καρκίνου.[28][29]
Τεχνικές[Επεξεργασία | επεξεργασία κώδικα]
Οι υπολογιστικοί βιολόγοι χρησιμοποιούν ένα ευρύ φάσμα λογισμικού και αλγορίθμων για να διεξάγουν την έρευνά τους.
Μάθηση χωρίς επίβλεψη[Επεξεργασία | επεξεργασία κώδικα]
Η μάθηση χωρίς επίβλεψη είναι ένας τύπος αλγορίθμου που βρίσκει μοτίβα σε μη επισημασμένα δεδομένα. Ένα παράδειγμα είναι η συσταδοποίηση k-μέσων, η οποία αποσκοπεί στην κατανομή n σημείων δεδομένων σε k συστάδες, στις οποίες κάθε σημείο δεδομένων ανήκει στη συστάδα με τον πλησιέστερο μέσο όρο. Μια άλλη εκδοχή είναι ο αλγόριθμος k-medoids, ο οποίος, όταν επιλέγει ένα κέντρο συστάδας ή κεντροειδές συστάδας, επιλέγει ένα από τα σημεία δεδομένων του συνόλου και όχι απλώς έναν μέσο όρο της συστάδας.
Ο αλγόριθμος ακολουθεί τα εξής βήματα:
- Επιλογή k διαφορετικών σημείων δεδομένων με τυχαίο τρόπο. Αυτές είναι οι αρχικές συστάδες.
- Μέτρηση της απόστασης μεταξύ κάθε σημείου και κάθε μιας από τις "k" συστάδες. (Αυτή είναι η απόσταση των σημείων από κάθε σημείο k).
- Ανάθεση κάθε σημείου στην πλησιέστερη συστάδα.
- Εύρεση του κέντρου κάθε συστάδας (medoid).
- Επανάληψη έως ότου οι συστάδες δεν αλλάζουν πλέον.
- Αξιολόγηση της ποιότητας της συσταδοποίησης προσθέτοντας τη διακύμανση εντός κάθε συστάδας.
- Επανάληψη των διαδικασιών με διαφορετικές τιμές του k.
- Επιλογή της καλύτερης τιμής για το "k" με την εύρεση του "αγκώνα" στο διάγραμμα του οποίου η τιμή k έχει τη μικρότερη διακύμανση.
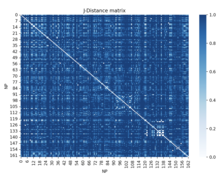
Ένα χαρακτηριστικό παράδειγμα στη βιολογία χρησιμοποιείται στην τρισδιάστατη χαρτογράφηση ενός γονιδιώματος. Πληροφορίες για την περιοχή HIST1 του χρωμοσώματος 13 ενός ποντικού συλλέγονται από το γονιδιακό αρχείο γονιδιακής έκφρασης Gene Expression Omnibus[30]. Οι πληροφορίες αυτές περιέχουν δεδομένα σχετικά με το ποια πυρηνικά προφίλ εμφανίζονται σε ορισμένες γονιδιωματικές περιοχές. Με αυτές τις πληροφορίες, η απόσταση Jaccard μπορεί να χρησιμοποιηθεί για την εύρεση μιας κανονικοποιημένης απόστασης μεταξύ όλων των τόπων.
Αναλυτικά γραφήματα[Επεξεργασία | επεξεργασία κώδικα]
Η ανάλυση γραφημάτων, ή ανάλυση δικτύων, είναι η μελέτη γραφημάτων που αναπαριστούν συνδέσεις μεταξύ διαφορετικών αντικειμένων. Οι γράφοι μπορούν να αναπαραστήσουν όλα τα είδη δικτύων στη βιολογία, όπως δίκτυα πρωτεϊνικών αλληλεπιδράσεων, ρυθμιστικά δίκτυα, μεταβολικά και βιοχημικά δίκτυα και πολλά άλλα. Υπάρχουν πολλοί τρόποι ανάλυσης αυτών των δικτύων. Ένας από αυτούς είναι η μελέτη της κεντρικότητας στους γράφους. Η έρευνα της κεντρικότητας γράφων αποδίδει στους κόμβους μια κατάταξη ανάλογα με τη δημοτικότητά τους ή την κεντρικότητά τους στο γράφο. Αυτό μπορεί να είναι χρήσιμο για τον προσδιορισμό των κόμβων που είναι οι πιο σημαντικοί. Αυτό μπορεί να είναι πολύ χρήσιμο στη βιολογία με πολλούς τρόπους. Για παράδειγμα, αν έχουμε δεδομένα σχετικά με τη γονιδιακή δραστηριότητα για μια δεδομένη περίοδο, μπορούμε να χρησιμοποιήσουμε την κεντρικότητα βαθμού για να δούμε ποια γονίδια είναι πιο ενεργά στο δίκτυο ή ποια γονίδια αλληλεπιδρούν περισσότερο με άλλα στο δίκτυο. Αυτό μπορεί να μας βοηθήσει να κατανοήσουμε το ρόλο που διαδραματίζουν ορισμένα γονίδια στο δίκτυο.
Υπάρχουν πολλοί τρόποι υπολογισμού της κεντρικότητας σε γραφήματα, οι οποίοι μπορούν να δώσουν διαφορετικά είδη πληροφοριών σχετικά με την κεντρικότητα. Η εύρεση κεντρικοτήτων στη βιολογία μπορεί να εφαρμοστεί σε πολλές διαφορετικές περιπτώσεις, μερικές από τις οποίες είναι η γονιδιακή ρύθμιση, η αλληλεπίδραση πρωτεϊνών και τα μεταβολικά δίκτυα[31].
Μάθηση με επίβλεψη[Επεξεργασία | επεξεργασία κώδικα]
Η μάθηση με επίβλεψη είναι ένας τύπος αλγορίθμου που μαθαίνει από δεδομένα με ετικέτες και μαθαίνει πώς να αποδίδει ετικέτες σε μελλοντικά δεδομένα που δεν έχουν ετικέτες. Στη βιολογία η μάθηση με επίβλεψη μπορεί να είναι χρήσιμη όταν έχουμε δεδομένα που γνωρίζουμε πώς να κατηγοριοποιήσουμε και θα θέλαμε να κατηγοριοποιήσουμε περισσότερα δεδομένα σε αυτές τις κατηγορίες.
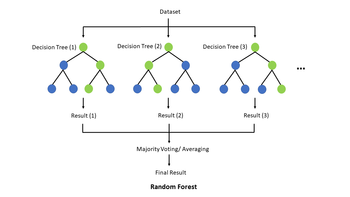
Ένας συνηθισμένος αλγόριθμος μάθησης με επίβλεψη είναι το τυχαίο δάσος, το οποίο χρησιμοποιεί πολυάριθμα δέντρα αποφάσεων για να εκπαιδεύσει ένα μοντέλο για την ταξινόμηση ενός συνόλου δεδομένων. Αποτελώντας τη βάση του τυχαίου δάσους, ένα δέντρο απόφασης είναι μια δομή που αποσκοπεί στην ταξινόμηση ή την επισήμανση κάποιου συνόλου δεδομένων χρησιμοποιώντας ορισμένα γνωστά χαρακτηριστικά των δεδομένων αυτών. Ένα πρακτικό βιολογικό παράδειγμα θα ήταν η λήψη των γενετικών δεδομένων ενός ατόμου και η πρόβλεψη του κατά πόσον το άτομο αυτό έχει προδιάθεση ή όχι να αναπτύξει μια συγκεκριμένη ασθένεια ή καρκίνο. Σε κάθε εσωτερικό κόμβο ο αλγόριθμος ελέγχει το σύνολο δεδομένων για ακριβώς ένα χαρακτηριστικό, ένα συγκεκριμένο γονίδιο στο προηγούμενο παράδειγμα, και στη συνέχεια διακλαδίζεται αριστερά ή δεξιά με βάση το αποτέλεσμα. Στη συνέχεια, σε κάθε κόμβο φύλλου, το δέντρο απόφασης αποδίδει μια ετικέτα κλάσης στο σύνολο δεδομένων. Έτσι, στην πράξη, ο αλγόριθμος διανύει ένα συγκεκριμένο μονοπάτι από τη ρίζα στο φύλλο με βάση το σύνολο δεδομένων εισόδου μέσω του δέντρου απόφασης, το οποίο οδηγεί στην ταξινόμηση του εν λόγω συνόλου δεδομένων. Συνήθως, τα δέντρα αποφάσεων έχουν μεταβλητές-στόχους που λαμβάνουν διακριτές τιμές, όπως ναι/όχι, οπότε αναφέρεται ως δέντρο ταξινόμησης, αλλά αν η μεταβλητή-στόχος είναι συνεχής τότε ονομάζεται δέντρο παλινδρόμησης. Για να κατασκευαστεί ένα δέντρο απόφασης, πρέπει προηγουμένως να εκπαιδευτεί χρησιμοποιώντας ένα σύνολο εκπαίδευσης για να προσδιοριστεί ποια χαρακτηριστικά είναι οι βέλτιστοι παράγοντες πρόβλεψης της μεταβλητής-στόχου.
Λογισμικό open source[Επεξεργασία | επεξεργασία κώδικα]
Το λογισμικό ανοικτού κώδικα παρέχει μια πλατφόρμα για την υπολογιστική βιολογία, όπου όλοι μπορούν να έχουν πρόσβαση και να επωφεληθούν από το λογισμικό που αναπτύσσεται στην έρευνα. Το PLOS αναφέρει τέσσερις κύριους λόγους για τη χρήση λογισμικού ανοικτού κώδικα:
- Αναπαραγωγιμότητα: Αυτό επιτρέπει στους ερευνητές να χρησιμοποιούν τις ακριβείς μεθόδους που χρησιμοποιούνται για τον υπολογισμό των σχέσεων μεταξύ βιολογικών δεδομένων.
- Ταχύτερη ανάπτυξη: Οι προγραμματιστές και οι ερευνητές δεν χρειάζεται να επανεφεύρουν τον υπάρχοντα κώδικα για δευτερεύουσες εργασίες. Αντίθετα, μπορούν να χρησιμοποιούν προϋπάρχοντα προγράμματα για να εξοικονομούν χρόνο στην ανάπτυξη και υλοποίηση μεγαλύτερων έργων.
- Αυξημένη ποιότητα: Η συμβολή πολλών ερευνητών που μελετούν το ίδιο θέμα παρέχει ένα επίπεδο διασφάλισης ότι δεν θα υπάρχουν λάθη στον κώδικα.
- Μακροπρόθεσμη διαθεσιμότητα: Τα προγράμματα open source δεν είναι συνδεδεμένα με επιχειρήσεις ή πατέντες. Αυτό επιτρέπει την ανάρτησή τους σε πολλαπλές ιστοσελίδες και διασφαλίζει ότι θα είναι διαθέσιμα στο μέλλον.[32]
Έρευνα[Επεξεργασία | επεξεργασία κώδικα]
Υπάρχουν πολλά μεγάλα συνέδρια που ασχολούνται με την υπολογιστική βιολογία. Μερικά αξιοσημείωτα παραδείγματα είναι τα Ευφυή Συστήματα για τη Μοριακή Βιολογία, το Ευρωπαϊκό Συνέδριο Υπολογιστικής Βιολογίας και η Έρευνα στην Υπολογιστική Μοριακή Βιολογία.
Υπάρχουν επίσης πολυάριθμα περιοδικά αφιερωμένα στην υπολογιστική βιολογία. Ορισμένα αξιοσημείωτα παραδείγματα περιλαμβάνουν το Journal of Computational Biology και το PLOS Computational Biology, ένα περιοδικό ανοικτής πρόσβασης με κριτές που έχει πολλά αξιοσημείωτα ερευνητικά έργα στον τομέα της υπολογιστικής βιολογίας. Παρέχουν κριτικές για το λογισμικό, σεμινάρια για λογισμικό ανοικτού κώδικα και εμφανίζουν πληροφορίες για επερχόμενα συνέδρια υπολογιστικής βιολογίας.
Σχετικούς τομείς[Επεξεργασία | επεξεργασία κώδικα]
Η υπολογιστική βιολογία, η βιοπληροφορική και η μαθηματική βιολογία είναι διεπιστημονικές προσεγγίσεις των βιοεπιστημών που βασίζονται σε ποσοτικούς κλάδους, όπως τα μαθηματικά και η επιστήμη της πληροφορικής. Το NIH περιγράφει την υπολογιστική/μαθηματική βιολογία ως τη χρήση υπολογιστικών/μαθηματικών προσεγγίσεων για την απάντηση θεωρητικών και πειραματικών ερωτημάτων στη βιολογία και, αντίστροφα, τη βιοπληροφορική ως την εφαρμογή της επιστήμης της πληροφορικής για την κατανόηση πολύπλοκων δεδομένων στις επιστήμες της ζωής [1].
Συγκεκριμένα, το NIH ορίζει
- Υπολογιστική βιολογία: Η ανάπτυξη και εφαρμογή δεδομένων-αναλυτικών και θεωρητικών μεθόδων, μαθηματικών μοντέλων και τεχνικών υπολογιστικής προσομοίωσης στη μελέτη βιολογικών, συμπεριφορικών και κοινωνικών συστημάτων[1].
- Βιοπληροφορική: Έρευνα, ανάπτυξη ή εφαρμογή υπολογιστικών εργαλείων και προσεγγίσεων για την επέκταση της χρήσης βιολογικών, ιατρικών, συμπεριφορικών ή υγειονομικών δεδομένων, συμπεριλαμβανομένων εκείνων για την απόκτηση, αποθήκευση, οργάνωση, αρχειοθέτηση, ανάλυση ή οπτικοποίηση τέτοιων δεδομένων[1].
Μολονότι κάθε τομέας είναι διακριτός, μπορεί να υπάρχει σημαντική επικάλυψη στη διεπαφή τους[1], σε τέτοιο βαθμό που για πολλούς η βιοπληροφορική και η υπολογιστική βιολογία είναι όροι που χρησιμοποιούνται εναλλακτικά.
Οι όροι υπολογιστική βιολογία και εξελικτικός υπολογισμός έχουν παρόμοιο όνομα, αλλά δεν πρέπει να συγχέονται. Σε αντίθεση με την υπολογιστική βιολογία, ο εξελικτικός υπολογισμός δεν ασχολείται με τη μοντελοποίηση και την ανάλυση βιολογικών δεδομένων. Αντίθετα, δημιουργεί αλγορίθμους με βάση τις ιδέες της εξέλιξης μεταξύ των ειδών. Μερικές φορές αναφέρεται ως γενετικοί αλγόριθμοι, η έρευνα αυτού του τομέα μπορεί να εφαρμοστεί στην υπολογιστική βιολογία. Ενώ ο εξελικτικός υπολογισμός δεν αποτελεί εγγενώς μέρος της υπολογιστικής βιολογίας, η υπολογιστική εξελικτική βιολογία είναι ένα υποπεδίο της[33].
Βιβλιογραφία[Επεξεργασία | επεξεργασία κώδικα]
- Introduction to Computational Biology: An Evolutionary Approach
- Bioinformatics and Computational Biology: A Primer for Biologists
- Encyclopedia of Bioinformatics and Computational Biology: ABC of Bioinformatics
- An Introduction to Computational Systems Biology
Δείτε επίσης[Επεξεργασία | επεξεργασία κώδικα]
- Βιοπληροφορική
- In silico
- Βιοστατιστική
- Υπολογιστική χημεία
- Μέθοδος Μόντε Κάρλο
- Πολύπλοκα βιολογικά δίκτυα
- Συνθετική βιολογία
- Βιολογία συστημάτων
Παραπομπές[Επεξεργασία | επεξεργασία κώδικα]
- ↑ 1,0 1,1 1,2 1,3 1,4 «Wayback Machine» (PDF). web.archive.org. Αρχειοθετήθηκε από το πρωτότυπο στις 5 Σεπτεμβρίου 2012. Ανακτήθηκε στις 21 Ιανουαρίου 2024.
- ↑ <«CCMB | Brown University». ccmb.brown.edu (στα Αγγλικά). Ανακτήθηκε στις 21 Ιανουαρίου 2024.
- ↑ 3,0 3,1 3,2 Hogeweg, Paulien (7 March 2011). «The Roots of Bioinformatics in Theoretical Biology». PLOS Computational Biology. 3 7 (3): e1002021. doi:. PMID 21483479. Bibcode: 2011PLSCB...7E2021H.
- ↑ «The Human Genome Project». The Human Genome Project. 22 Δεκεμβρίου 2020. Ανακτήθηκε στις 13 Απριλίου 2022.
- ↑ «Human Genome Project FAQ». web.archive.org. 23 Απριλίου 2022. Αρχειοθετήθηκε από το πρωτότυπο στις 23 Απριλίου 2022. Ανακτήθηκε στις 19 Ιανουαρίου 2024.
- ↑ «T2T-CHM13v1.1 - Genome - Assembly». NCBI. Αρχειοθετήθηκε από το πρωτότυπο στις 29 Ιουνίου 2023. Ανακτήθηκε στις 20 Απριλίου 2022.
- ↑ «Genome List - Genome». NCBI. Ανακτήθηκε στις 20 Απριλίου 2022.
- ↑ Bourne, Philip (2012). «Rise and Demise of Bioinformatics? Promise and Progress». PLOS Computational Biology 8 (4): e1002487. doi:. PMID 22570600. Bibcode: 2012PLSCB...8E2487O.
- ↑ «COSI Information». www.iscb.org. Αρχειοθετήθηκε από το πρωτότυπο στις 21 Απριλίου 2022. Ανακτήθηκε στις 21 Απριλίου 2022.
- ↑ Grenander, Ulf; Miller, Michael I. (1998-12-01). «Computational Anatomy: An Emerging Discipline». Q. Appl. Math. 56 (4): 617–694. doi:. https://archive.org/details/sim_quarterly-of-applied-mathematics_1998-12_56_4/page/617.
- ↑ «Mathematical Biology | Faculty of Science». www.ualberta.ca. Ανακτήθηκε στις 18 Απριλίου 2022.
- ↑ 12,0 12,1 12,2 «The Sub-fields of Computational Biology». Ninh Laboratory of Computational Biology (στα Αγγλικά). 18 Φεβρουαρίου 2013. Ανακτήθηκε στις 18 Απριλίου 2022.
- ↑ 13,0 13,1 Kitano, Hiroaki (14 November 2002). «Computational systems biology». Nature 420 (6912): 206–10. doi:. PMID 12432404. Bibcode: 2002Natur.420..206K. https://archive.org/details/sim_nature-uk_2002-11-14_420_6912/page/206.
- ↑ Favrin, Bean (2 September 2014). «esyN: Network Building, Sharing and Publishing.». PLOS ONE 9 (9): e106035. doi:. PMID 25181461. Bibcode: 2014PLoSO...9j6035B.
- ↑ Antonio Carvajal-Rodríguez (2012). «Simulation of Genes and Genomes Forward in Time». Current Genomics 11 (1): 58–61. doi:. PMID 20808525.
- ↑ «Genome Sequencing to the Rest of Us». Scientific American. http://www.scientificamerican.com/article.cfm?id=personal-genome-sequencing.
- ↑ 17,0 17,1 17,2 Koonin, Eugene (6 March 2001). «Computational Genomics». Curr. Biol. 11 (5): 155–158. doi:. PMID 11267880.
- ↑ «Sequence Alignment - an overview | ScienceDirect Topics». www.sciencedirect.com. Ανακτήθηκε στις 18 Απριλίου 2022.
- ↑ «Gene Ontology Resource». Gene Ontology Resource. Ανακτήθηκε στις 18 Απριλίου 2022.
- ↑ Beagrie, Robert A.; Scialdone, Antonio; Schueler, Markus; Kraemer, Dorothee C. A.; Chotalia, Mita; Xie, Sheila Q.; Barbieri, Mariano; de Santiago, Inês και άλλοι. (March 2017). «Complex multi-enhancer contacts captured by genome architecture mapping» (στα αγγλικά). Nature 543 (7646): 519–524. doi:. ISSN 1476-4687. PMID 28273065. Bibcode: 2017Natur.543..519B.
- ↑ «Computational Neuroscience | Neuroscience». www.bu.edu.
- ↑ 22,0 22,1 Sejnowski, Terrence; Christof Koch; Patricia S. Churchland (9 September 1988). «Computational Neuroscience». Science. 4871 241 (4871): 1299–306. doi:. PMID 3045969. Bibcode: 1988Sci...241.1299S. https://archive.org/details/sim_science_1988-09-09_241_4871/page/1299.
- ↑ Dauvermann, Maria R.; Whalley, Heather C.; Schmidt, Andrã©; Lee, Graham L.; Romaniuk, Liana; Roberts, Neil; Johnstone, Eve C.; Lawrie, Stephen M. και άλλοι. (2014). «Computational Neuropsychiatry – Schizophrenia as a Cognitive Brain Network Disorder». Frontiers in Psychiatry 5: 30. doi:. PMID 24723894.
- ↑ Tretter, F.; Albus, M. (December 2007). «'Computational Neuropsychiatry' of Working Memory Disorders in Schizophrenia: The Network Connectivity in Prefrontal Cortex - Data and Models». Pharmacopsychiatry 40 (S 1): S2–S16. doi:.
- ↑ Marin-Sanguino, A.; Mendoza, E. (2008). «Hybrid Modeling in Computational Neuropsychiatry». Pharmacopsychiatry 41: S85–S88. doi:. PMID 18756425.
- ↑ Price, Michael (13 Απριλίου 2012). «Computational Biologists: The Next Pharma Scientists?».
- ↑ 27,0 27,1 Jessen, Walter (15 Απριλίου 2012). «Pharma's shifting strategy means more jobs for computational biologists».
- ↑ Barbolosi, Dominique; Ciccolini, Joseph; Lacarelle, Bruno; Barlesi, Fabrice; Andre, Nicolas (2016). «Computational oncology--mathematical modelling of drug regimens for precision medicine». Nature Reviews Clinical Oncology 13 (4): 242–254. doi:. PMID 26598946.
- ↑ Yakhini, Zohar (2011). «Cancer Computational Biology». BMC Bioinformatics 12: 120. doi:. PMID 21521513.
- ↑ «GEO Accession viewer».
- ↑ Koschützki, Dirk; Schreiber, Falk (2008-05-15). «Centrality Analysis Methods for Biological Networks and Their Application to Gene Regulatory Networks». Gene Regulation and Systems Biology 2: 193–201. doi:. ISSN 1177-6250. PMID 19787083.
- ↑ Prlić, Andreas; Lapp, Hilmar (2012). «The PLOS Computational Biology Software Section». PLOS Computational Biology 8 (11): e1002799. doi:. Bibcode: 2012PLSCB...8E2799P.
- ↑ Foster, James (June 2001). «Evolutionary Computation». Nature Reviews Genetics 2 (6): 428–436. doi:. PMID 11389459.
|


