Παράγοντας μεταγραφής

Στη μοριακή βιολογία, ένας παράγοντας μεταγραφής ή μεταγραφικός παράγοντας (transcription factor, TF) (ή αλληλουχιοειδικός παράγοντας δέσμευσης DNA) είναι μια πρωτεΐνη που ελέγχει τον ρυθμό μεταγραφής της γενετικής πληροφορίας από DNA σε αγγελιοφόρο RNA, δεσμεύοντας σε συγκεκριμένη αλληλουχία DNA.[1][2] Η λειτουργία των TFs είναι να ρυθμίζουν —ενεργοποιούν και απενεργοποιούν— γονίδια προκειμένου να διασφαλίσουν ότι εκφράζονται στα επιθυμητά κύτταρα την κατάλληλη στιγμή και στη σωστή ποσότητα σε όλη τη διάρκεια της ζωής του κυττάρου και του οργανισμού. Ομάδες TF λειτουργούν με συντονισμένο τρόπο για να κατευθύνουν την κυτταρική διαίρεση, την κυτταρική ανάπτυξη και τον κυτταρικό θάνατο καθ' όλη τη διάρκεια της ζωής. Η κυτταρική μετανάστευση και οργάνωση (σωματικού σχεδίου) κατά την εμβρυϊκή ανάπτυξη και κατά διαστήματα ως απόκριση σε σήματα έξω από το κύτταρο, όπως μια ορμόνη. Υπάρχουν 1500-1600 TF στο ανθρώπινο γονιδίωμα.[3][4]. Οι μεταγραφικοί παράγοντες είναι μέλη του πρωτεώματος καθώς και του regulome (ρυθμίωμα).
Τα TF λειτουργούν μόνα τους ή με άλλες πρωτεΐνες σε ένα σύμπλεγμα, προάγοντας (ως ενεργοποιητές), ή μπλοκάροντας (ως καταστολείς) τη στρατολόγηση της RNA πολυμεράσης (το ένζυμο που εκτελεί τη μεταγραφή της γενετικής πληροφορίας από το DNA στο RNA) σε συγκεκριμένα γονίδια.[5][6][7]
Ένα καθοριστικό χαρακτηριστικό των TFs είναι ότι περιέχουν τουλάχιστον μία τουλάχιστον περιοχή σύνδεσης (πρόσδεσης) στο DNA (DNA-binding domain, DBD), η οποία προσκολλάται σε μια συγκεκριμένη αλληλουχία DNA δίπλα στα γονίδια που ρυθμίζουν.[8][9] Τα TF ομαδοποιούνται σε κλάσεις (τάξεις) με βάση τα DBD τους.[10][11] Άλλες πρωτεΐνες όπως συνενεργοποιητές (coactivators), αναδιαμορφωτές χρωματίνης (chromatin remodelers), σύμπλοκο αναδιαμόρφωσης δομής χρωματίνης, ακετυλομεταφοράσες ιστόνης, αποακετυλάσες ιστόνης, κινάσες και μεθυλάσες είναι επίσης απαραίτητες για τη γονιδιακή ρύθμιση, αλλά στερούνται περιοχών δέσμευσης DNA και επομένως δεν είναι TF.[12]
Οι TF παρουσιάζουν ενδιαφέρον για την ιατρική, επειδή οι μεταλλάξεις των TF μπορούν να προκαλέσουν συγκεκριμένες ασθένειες και τα φάρμακα μπορεί να στοχεύουν δυνητικά σε αυτές.
Αριθμός
[Επεξεργασία | επεξεργασία κώδικα]Οι μεταγραφικοί παράγοντες είναι απαραίτητοι για τη ρύθμιση της γονιδιακής έκφρασης και, κατά συνέπεια, βρίσκονται σε όλους τους ζωντανούς οργανισμούς. Ο αριθμός των μεταγραφικών παραγόντων που βρίσκονται μέσα σε έναν οργανισμό αυξάνεται με το μέγεθος του γονιδιώματος και τα μεγαλύτερα γονιδιώματα τείνουν να έχουν περισσότερους μεταγραφικούς παράγοντες ανά γονίδιο.[13]
Υπάρχουν περίπου 2800 πρωτεΐνες στο ανθρώπινο γονιδίωμα που περιέχουν περιοχές δέσμευσης στο DNA και 1600 από αυτές υποτίθεται ότι λειτουργούν ως παράγοντες μεταγραφής,[3] αν και άλλες μελέτες δείχνουν ότι είναι μικρότερος ο αριθμός.[14]. Επομένως, περίπου το 10% των γονιδίων στον κώδικα γονιδιώματος είναι για παράγοντες μεταγραφής, γεγονός που καθιστά αυτή την οικογένεια τη μεγαλύτερη οικογένεια ανθρώπινων πρωτεϊνών. Επιπλέον, τα γονίδια συχνά πλαισιώνονται από διάφορες θέσεις δέσμευσης για διαφορετικούς παράγοντες μεταγραφής και η αποτελεσματική έκφραση καθενός από αυτά τα γονίδια απαιτεί τη συνεργατική δράση πολλών διαφορετικών παραγόντων μεταγραφής (βλέπε, για παράδειγμα πυρηνικούς παράγοντες ηπατοκυττάρων). Ως εκ τούτου, η συνδυαστική χρήση ενός υποσυνόλου των περίπου 2000 ανθρώπινων μεταγραφικών παραγόντων αντιπροσωπεύει εύκολα τη μοναδική ρύθμιση κάθε γονιδίου στο ανθρώπινο γονιδίωμα κατά τη διάρκεια της ανάπτυξης.[12]
Μηχανισμός
[Επεξεργασία | επεξεργασία κώδικα]Οι μεταγραφικοί παράγοντες συνδέονται είτε σε περιοχές ενισχυτή, είτε σε περιοχές προαγωγέα του DNA που γειτνιάζουν με τα γονίδια που ρυθμίζουν. Ανάλογα με τον παράγοντα μεταγραφής, η μεταγραφή του παρακείμενου γονιδίου ρυθμίζεται είτε προς τα πάνω, είτε προς τα κάτω. Οι μεταγραφικοί παράγοντες χρησιμοποιούν διάφορους μηχανισμούς για τη ρύθμιση της γονιδιακής έκφρασης.[15] Αυτοί οι μηχανισμοί:
- σταθεροποιούν ή αποκλείουν τη σύνδεση της RNA πολυμεράσης με το DNA
- καταλύουν την ακετυλίωση ή την αποακετυλίωση των πρωτεϊνών ιστόνης. Ο παράγοντας μεταγραφής μπορεί είτε να το κάνει άμεσα είτε να προσλάβει άλλες πρωτεΐνες με αυτή την καταλυτική δραστικότητα. Πολλοί παράγοντες μεταγραφής χρησιμοποιούν τον ένα ή τον άλλο από δύο αντιτιθέμενους μηχανισμούς για να ρυθμίσουν τη μεταγραφή.[16]
- Η ενεργότητα της ακετυλομεταφοράσης ιστόνης (histone acetyltransferase, HAT) – ακετυλιώνει τις πρωτεΐνες ιστόνης, που αποδυναμώνει τη συσχέτιση του DNA με τις ιστόνες, που καθιστούν το DNA πιο προσιτό στη μεταγραφή, ρυθμίζοντας έτσι τη μεταγραφή προς τα πάνω.
- Η ενεργότητα της αποακετυλάσης ιστόνης (histone deacetylase HDAC) – αποακετυλιώνει τις πρωτεΐνες ιστόνης, η οποία ενισχύει τη συσχέτιση του DNA με τις ιστόνες, που καθιστά το DNA λιγότερο προσβάσιμο στη μεταγραφή, μειώνοντας έτσι τη μεταγραφή
- προσλαμβάνουν πρωτεϊνών συνενεργοποιητή ή συνκαταστολέα στο σύμπλεγμα του παράγοντα μεταγραφής DNA [17]
Λειτουργία
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής είναι μία από τις ομάδες πρωτεϊνών που διαβάζουν και ερμηνεύουν το γενετικό «σχεδιασμό» στο DNA. Συνδέονται με το DNA και βοηθούν στην έναρξη ενός προγράμματος αυξημένης ή μειωμένης γονιδιακής μεταγραφής. Ως εκ τούτου, είναι ζωτικής σημασίας για πολλές σημαντικές κυτταρικές διεργασίες. Παρακάτω είναι μερικές από τις σημαντικές λειτουργίες και τους βιολογικούς ρόλους στους οποίους εμπλέκονται οι παράγοντες μεταγραφής:
Βασική μεταγραφική ρύθμιση
[Επεξεργασία | επεξεργασία κώδικα]Στα ευκαρυωτικά κύτταρα, μια σημαντική κατηγορία μεταγραφικών παραγόντων που ονομάζονται γενικοί παράγοντες μεταγραφής (general transcription factors, GTF), είναι απαραίτητοι για να συμβεί η μεταγραφή.[18][19][20] Πολλοί από αυτoύς τους GTF στην πραγματικότητα δεν δεσμεύουν το DNA, αλλά μάλλον αποτελούν μέρος του μεγάλου συμπλέγματος προεκκίνησης μεταγραφής που αλληλεπιδρά με την RNA πολυμεράση άμεσα. Οι πιο κοινοί GTF είναι οι TFIIA, TFIIB, TFIID, TFIIE, TFIIF και TFIIH.[21] Το σύμπλεγμα προεκκίνησης συνδέεται με περιοχές του DNA του προαγωγέα ανάντη προς το γονίδιο που ρυθμίζουν.
Διαφοροποιημένη ενίσχυση της μεταγραφής
[Επεξεργασία | επεξεργασία κώδικα]Άλλοι μεταγραφικοί παράγοντες ρυθμίζουν διαφορετικά την έκφραση ποικίλων γονιδίων δεσμεύοντας σε περιοχές ενισχυτών του DNA που γειτνιάζουν με ρυθμιζόμενα γονίδια. Αυτοί οι παράγοντες μεταγραφής είναι κρίσιμοι για να διασφαλιστεί ότι τα γονίδια εκφράζονται στο σωστό κύτταρο, τη σωστή στιγμή και στη σωστή ποσότητα, ανάλογα με τις μεταβαλλόμενες απαιτήσεις του οργανισμού.
Ανάπτυξη
[Επεξεργασία | επεξεργασία κώδικα]Πολλοί μεταγραφικοί παράγοντες σε πολυκύτταρους οργανισμούς εμπλέκονται στην ανάπτυξη.[22] Ανταποκρινόμενοι σε ερεθίσματα, αυτοί οι μεταγραφικοί παράγοντες ενεργοποιούν/απενεργοποιούν τη μεταγραφή των κατάλληλων γονιδίων, τα οποία, με τη σειρά τους, επιτρέπουν αλλαγές στη μορφολογία του κυττάρου ή σε δραστηριότητες που απαιτούνται για τον προσδιορισμό της μοίρας των κυττάρων και την κυτταρική διαφοροποίηση. Η οικογένεια μεταγραφικών παραγόντων Hox, για παράδειγμα, είναι σημαντική για τον σωστό σχηματισμό σωματικού μοτίβου σε οργανισμούς τόσο διαφορετικούς όσο οι μύγες των φρούτων και οι άνθρωποι.[23][24] Ένα άλλο παράδειγμα είναι ο παράγοντας μεταγραφής που κωδικοποιείται από το γονίδιο περιοχής φυλοκαθορισμού του Υ (sex-determining region Y, SRY), το οποίο παίζει σημαντικό ρόλο στον προσδιορισμό του φύλου στους ανθρώπους.[25]
Απόκριση σε μεσοκυττάρια σήματα
[Επεξεργασία | επεξεργασία κώδικα]Τα κύτταρα μπορούν να επικοινωνούν μεταξύ τους απελευθερώνοντας μόρια που παράγουν καταρράκτες σηματοδότησης μέσα σε ένα άλλο δεκτικό κύτταρο. Εάν το σήμα απαιτεί ρύθμιση προς τα πάνω ή προς τα κάτω των γονιδίων στο κύτταρο δέκτη, συχνά οι παράγοντες μεταγραφής θα είναι κατάντη στον καταρράκτη σηματοδότησης.[26] Η σηματοδότηση οιστρογόνων είναι ένα παράδειγμα ενός αρκετά σύντομου καταρράκτη σηματοδότησης που περιλαμβάνει τον μεταγραφικό παράγοντα υποδοχέα οιστρογόνου: Τα οιστρογόνα εκκρίνονται από ιστούς όπως οι ωοθήκες και ο πλακούντας, διασταυρώνονται με την κυτταρική μεμβράνη του υποδοχέα κυττάρου και δεσμεύονται από τον υποδοχέα οιστρογόνου στο κυτταρόπλασμα του κυττάρου. Στη συνέχεια, ο υποδοχέας οιστρογόνου πηγαίνει στον πυρήνα του κυττάρου και συνδέεται με τις θέσεις δέσμευσης DNA του κυττάρου, αλλάζοντας τη μεταγραφική ρύθμιση των σχετικών γονιδίων.[27]
Απόκριση στο περιβάλλον
[Επεξεργασία | επεξεργασία κώδικα]Όχι μόνο οι παράγοντες μεταγραφής δρουν κατάντη των καταρρακτών σηματοδότησης που σχετίζονται με βιολογικά ερεθίσματα, αλλά μπορούν επίσης να είναι και κατάντη των καταρρακτών σηματοδότησης που εμπλέκονται σε περιβαλλοντικά ερεθίσματα. Τα παραδείγματα περιλαμβάνουν τον παράγοντα θερμικού πλήγματος (heat shock factor, HSF), ο οποίος ρυθμίζει προς τα πάνω τα γονίδια που είναι απαραίτητα για την επιβίωση σε υψηλότερες θερμοκρασίες,[28] [[]] (HIF), τον επαγώγιμο παράγοντα υποξίας (hypoxia inducible factor, HIF), ο οποίος ρυθμίζει προς τα πάνω τα γονίδια που είναι απαραίτητα για την επιβίωση των κυττάρων σε περιβάλλοντα με χαμηλή περιεκτικότητα σε οξυγόνο,[29] και την πρωτεΐνη δέσμευσης ρυθμιστικού στοιχείου στερόλης (SREBP), η οποία βοηθά στη διατήρηση των σωστών επιπέδων λιπιδίων στο κύτταρο.[30]
Έλεγχος κυτταρικού κύκλου
[Επεξεργασία | επεξεργασία κώδικα]Πολλοί μεταγραφικοί παράγοντες, ειδικά κάποιοι που είναι πρωτοογκογονίδια ή ογκοκαταστολείς, βοηθούν στη ρύθμιση του κυτταρικού κύκλου και ως εκ τούτου καθορίζουν πόσο μεγάλο θα γίνει ένα κύτταρο και πότε μπορεί να χωριστεί σε δύο θυγατρικά κύτταρα.[31][32] Ένα παράδειγμα είναι το ογκογονίδιο Myc, το οποίο έχει σημαντικούς ρόλους στην κυτταρική ανάπτυξη και την απόπτωση.[33]
Παθογένεση
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής μπορούν επίσης να χρησιμοποιηθούν για την αλλαγή της γονιδιακής έκφρασης σε ένα κύτταρο ξενιστή για την προώθηση της παθογένεσης. Ένα καλά μελετημένο παράδειγμα αυτού είναι οι τελεστές που μοιάζουν με ενεργοποιητές μεταγραφής (transcription-activator like effectors, TAL ή TALE) που εκκρίνονται από τα βακτήρια Xanthomonas. Όταν εγχέονται στα φυτά, αυτές οι πρωτεΐνες μπορούν να εισέλθουν στον πυρήνα του φυτικού κυττάρου, να δεσμεύσουν τις αλληλουχίες των φυτικών προαγωγέων και να ενεργοποιήσουν τη μεταγραφή των φυτικών γονιδίων που βοηθούν στη βακτηριακή μόλυνση.[34] Οι τελεστές TAL περιέχουν μια κεντρική περιοχή επανάληψης στην οποία υπάρχει μια απλή σχέση μεταξύ της ταυτότητας δύο κρίσιμων υπολειμμάτων σε διαδοχικές επαναλήψεις και διαδοχικές βάσεις DNA στη θέση στόχου του τελεστή TAL.[35][36] Αυτή η ιδιότητα πιθανότατα διευκολύνει την εξέλιξη αυτών των πρωτεϊνών προκειμένου να ανταγωνιστούν καλύτερα τους αμυντικούς μηχανισμούς του κυττάρου ξενιστή.[37]
Ρύθμιση
[Επεξεργασία | επεξεργασία κώδικα]Είναι σύνηθες στη βιολογία οι σημαντικές διεργασίες να έχουν πολλαπλές στρώσεις ρύθμισης και ελέγχου. Αυτό ισχύει και με τους μεταγραφικούς παράγοντες: Όχι μόνο οι μεταγραφικοί παράγοντες ελέγχουν τους ρυθμούς μεταγραφής για να ρυθμίσουν τις ποσότητες γονιδιακών προϊόντων (RNA και πρωτεΐνη) που είναι διαθέσιμα στο κύτταρο, αλλά και οι ίδιοι οι μεταγραφικοί παράγοντες ρυθμίζονται (συχνά από άλλους μεταγραφικούς παράγοντες). Ακολουθεί μια σύντομη περίληψη ορισμένων από τους τρόπους με τους οποίους μπορεί να ρυθμιστεί η δραστηριότητα των παραγόντων μεταγραφής:
Σύνθεση
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής (όπως όλες οι πρωτεΐνες) μεταγράφονται από ένα γονίδιο σε ένα χρωμόσωμα σε RNA και στη συνέχεια το RNA μεταφράζεται σε πρωτεΐνη. Οποιοδήποτε από αυτά τα βήματα μπορεί να ρυθμιστεί για να επηρεάσει την παραγωγή (και συνεπώς την ενεργότητα) ενός μεταγραφικού παράγοντα. Μια συνέπεια αυτού είναι ότι οι παράγοντες μεταγραφής μπορούν να αυτορυθμιστούν. Για παράδειγμα, σε έναν βρόχο αρνητικής ανάδρασης, ο παράγοντας μεταγραφής λειτουργεί ως ο δικός του καταστολέας: Εάν η πρωτεΐνη του μεταγραφικού παράγοντα δεσμεύει το DNA του δικού της γονιδίου, ρυθμίζει προς τα κάτω την παραγωγή περισσότερων από τον εαυτό της. Αυτός είναι ένας μηχανισμός για τη διατήρηση χαμηλών επιπέδων ενός μεταγραφικού παράγοντα σε ένα κύτταρο.[38]
Πυρηνικός εντοπισμός
[Επεξεργασία | επεξεργασία κώδικα]Στα ευκαρυωτικά κύτταρα, οι παράγοντες μεταγραφής (όπως οι περισσότερες πρωτεΐνες) μεταγράφονται στον πυρήνα αλλά στη συνέχεια μεταφράζονται στο κυτταρόπλασμα του κυττάρου. Πολλές πρωτεΐνες που είναι ενεργές στον πυρήνα περιέχουν σήματα εντοπισμού πυρήνων που τις κατευθύνουν στον πυρήνα. Όμως, για πολλούς μεταγραφικούς παράγοντες, αυτό είναι ένα βασικό σημείο στη ρύθμισή τους.[39] Σημαντικές κατηγορίες παραγόντων μεταγραφής, όπως ορισμένοι πυρηνικοί υποδοχείς πρέπει πρώτα να δεσμεύσουν έναν συνδέτη, ενώ βρίσκονται στο κυτταρόπλασμα προτού μπορέσουν να μετακινηθούν στον πυρήνα.[39]
Ενεργοποίηση
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής μπορούν να ενεργοποιηθούν (ή να απενεργοποιηθούν) μέσω της περιοχής ανίχνευσης σήματος με διάφορους μηχανισμούς όπως:
- Δέσμευση συνδέτη (ligand) – Η δέσμευση συνδέτη μπορεί να επηρεάσει πού βρίσκεται ένας μεταγραφικός παράγοντας μέσα σε ένα κύτταρο, αλλά η δέσμευση συνδέτη μπορεί επίσης να επηρεάσει εάν ο μεταγραφικός παράγοντας είναι σε ενεργή κατάσταση και ικανός να δεσμεύει το DNA ή άλλους συμπαράγοντες.
- Φωσφορυλίωση[40][41] –Πολλοί μεταγραφικοί παράγοντες όπως οι πρωτεΐνες STAT πρέπει να φωσφορυλιωθούν για να μπορέσουν να δεσμεύσουν το DNA.
- Αλληλεπίδραση με άλλους μεταγραφικούς παράγοντες (π.χ., ομο- ή ετερο-διμερισμός) ή συνρυθμιστικές πρωτεΐνες
Προσβασιμότητα θέσης δέσμευσης DNA
[Επεξεργασία | επεξεργασία κώδικα]Στους ευκαρυώτες, το DNA οργανώνεται με τη βοήθεια ιστονών σε συμπαγή σωματίδια που ονομάζονται νουκλεοσώματα, όπου αλληλουχίες περίπου 147 ζευγών βάσεων DNA κάνουν ~1,65 στροφές γύρω από τα οκταμερή πρωτεΐνης ιστόνης. Το DNA μέσα στα νουκλεοσώματα είναι απρόσιτο σε πολλούς μεταγραφικούς παράγοντες. Ορισμένοι παράγοντες μεταγραφής, οι λεγόμενοι πρωτοπόροι παράγοντες (pioneer factors) εξακολουθούν να είναι σε θέση να δεσμεύουν τις θέσεις δέσμευσης DNA τους στο νουκλεοσωμικό DNA. Για τους περισσότερους άλλους μεταγραφικούς παράγοντες, το νουκλεόσωμα θα πρέπει να ξετυλιχθεί ενεργά από μοριακούς κινητήρες όπως τους αναδιαμορφωτές χρωματίνης (chromatin remodelers). [42] Εναλλακτικά, το νουκλεόσωμα μπορεί να ξετυλιχθεί μερικώς από θερμικές διακυμάνσεις, επιτρέποντας την προσωρινή πρόσβαση στη θέση δέσμευσης του μεταγραφικού παράγοντα. Σε πολλές περιπτώσεις, ένας παράγοντας μεταγραφής χρειάζεται να ανταγωνίζεται για τη δέσμευσή του στη θέση πρόσδεσής του στο DNA με άλλους μεταγραφικούς παράγοντες και ιστόνες ή μη ιστονικές πρωτεΐνες χρωματίνης.[43] Ζεύγη μεταγραφικών παραγόντων και άλλων πρωτεϊνών μπορούν να παίξουν ανταγωνιστικούς ρόλους (ενεργοποιητής έναντι καταστολέα) στη ρύθμιση του ίδιου γονιδίου.
Διαθεσιμότητα άλλων συμπαραγόντων/παραγόντων μεταγραφής
[Επεξεργασία | επεξεργασία κώδικα]Οι περισσότεροι παράγοντες μεταγραφής δεν λειτουργούν μόνοι τους. Πολλές μεγάλες οικογένειες TF σχηματίζουν σύνθετες ομοτυπικές ή ετεροτυπικές αλληλεπιδράσεις μέσω διμερισμού.[44] Για να συμβεί γονιδιακή μεταγραφή, ένας αριθμός μεταγραφικών παραγόντων πρέπει να συνδεθεί με τις ρυθμιστικές αλληλουχίες του DNA. Αυτή η συλλογή παραγόντων μεταγραφής, με τη σειρά της, στρατολογεί ενδιάμεσες πρωτεΐνες όπως συμπαράγοντες που επιτρέπουν αποτελεσματική στρατολόγηση του συμπλόκου προεκκίνησης και της RNA πολυμεράσης. Έτσι, για να ξεκινήσει η μεταγραφή ένας μεμονωμένος παράγοντας μεταγραφής, όλες αυτές οι άλλες πρωτεΐνες πρέπει επίσης να είναι παρούσες και ο μεταγραφικός παράγοντας πρέπει να βρίσκεται σε μια κατάσταση όπου μπορεί να συνδεθεί με αυτές εάν είναι απαραίτητο. Οι συμπαράγοντες είναι πρωτεΐνες που ρυθμίζουν τις επιδράσεις των παραγόντων μεταγραφής. Οι συμπαράγοντες είναι εναλλάξιμοι μεταξύ συγκεκριμένων γονιδιακών προαγωγέων. Το πρωτεϊνικό σύμπλεγμα που καταλαμβάνει το DNA του προαγωγέα και η αλληλουχία αμινοξέων του συμπαράγοντα καθορίζουν τη χωρική του διαμόρφωση. Για παράδειγμα, ορισμένοι υποδοχείς στεροειδών μπορούν να ανταλλάσσουν συμπαράγοντες με NF-κB, που είναι ένας διακόπτης μεταξύ της φλεγμονής και της κυτταρικής διαφοροποίησης. Ως εκ τούτου, τα στεροειδή μπορούν να επηρεάσουν τη φλεγμονώδη απόκριση και τη λειτουργία ορισμένων ιστών.[45]
Αλληλεπίδραση με μεθυλιωμένη κυτοσίνη
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής και οι μεθυλιωμένες κυτοσίνες στο DNA έχουν και οι δύο κύριους ρόλους στη ρύθμιση της γονιδιακής έκφρασης. (Η μεθυλίωση της κυτοσίνης στο DNA συμβαίνει κυρίως όπου η κυτοσίνη ακολουθείται από γουανίνη στην αλληλουχία DNA 5' έως 3', μια θέση CpG.) Η μεθυλίωση των θέσεων CpG σε μια περιοχή προαγωγέα ενός γονιδίου συνήθως καταστέλλει τη γονιδιακή μεταγραφή,[46] ενώ η μεθυλίωση των CpG στο σώμα ενός γονιδίου αυξάνει την έκφραση.[47] Τα ένζυμα TET παίζουν κεντρικό ρόλο στην απομεθυλίωση των μεθυλιωμένων κυτοσινών. Η απομεθυλίωση των CpG σε έναν προαγωγέα γονιδίου από τη δραστικότητα του ενζύμου ΤΕΤ αυξάνει τη μεταγραφή του γονιδίου.[48]
Αξιολογήθηκαν οι θέσεις δέσμευσης του DNA 519 μεταγραφικών παραγόντων.[49] Από αυτούς, 169 μεταγραφικοί παράγοντες (33%) δεν είχαν δινουκλεοτίδια CpG στις θέσεις πρόσδεσής τους και 33 μεταγραφικοί παράγοντες (6%) μπορούσαν να συνδεθούν σε ένα μοτίβο που περιέχει CpG, αλλά δεν εμφάνιζαν προτίμηση για μια θέση δέσμευσης είτε με μεθυλιωμένο ή μη μεθυλιωμένο CpG. Υπήρχαν 117 μεταγραφικοί παράγοντες (23%) που παρεμποδίστηκαν από τη δέσμευση στην αλληλουχία δέσμευσής τους εάν περιείχε μεθυλιωμένη θέση CpG, 175 μεταγραφικοί παράγοντες (34%) που είχαν ενισχυμένη δέσμευση εάν η αλληλουχία δέσμευσής τους είχε μεθυλιωμένη θέση CpG και σε 25 μεταγραφικούς παράγοντες (5%) είτε αναστέλλονταν, είτε είχαν ενισχυμένη δέσμευση ανάλογα με το πού στην αλληλουχία δέσμευσης βρισκόταν το μεθυλιωμένο CpG.
Τα ένζυμα ΤΕΤ δεν δεσμεύονται ειδικά με μεθυλοκυτοσίνη εκτός από την πρόσληψη. Πολλοί μεταγραφικοί παράγοντες σημαντικοί για τη διαφοροποίηση των κυττάρων και την προδιαγραφή γενεαλογίας, συμπεριλαμβανομένων των NANOG, SALL4A, WT1, EBF1, PU.1 και E2A, έχει αποδειχθεί ότι στρατολογούν ένζυμα TET σε συγκεκριμένους γονιδιωματικούς τόπους (κυρίως ενισχυτές) για να δράσουν στη μεθυλκυτοσίνη (mC) και να τη μετατρέψουν σε υδροξυμεθυλκυτοσίνη hmC (και στις περισσότερες περιπτώσεις να τα σημαδέψουν για επακόλουθη πλήρη απομεθυλίωση σε κυτοσίνη).[50]. Ο μεταγραφικός παράγοντας EGR1 στρατολογεί την πρωτεΐνη TET1 που ξεκινά μια οδό απομεθυλίωσης DNA. Η μετατροπή του mC σε hmC με τη μεσολάβηση TET φαίνεται να διαταράσσει τη δέσμευση πρωτεϊνών που δεσμεύουν τη 5 mC, συμπεριλαμβανομένων των πρωτεϊνών MECP2 και MBD (Methyl-CpG-binding domain), διευκολύνοντας την αναδιαμόρφωση των νουκλεοσωμάτων και τη δέσμευση των παραγόντων μεταγραφής,. ενεργοποιώντας έτσι τη μεταγραφή αυτών των γονιδίων. Το EGR1 είναι ένας σημαντικός μεταγραφικός παράγοντας στον σχηματισμό μνήμης. Έχει ουσιαστικό ρόλο στον επαναπρογραμματισμό επιγενετικά του νευρώνα του εγκεφάλου. Ο μεταγραφικός παράγοντας EGR1 στρατολογεί την πρωτεΐνη TET1 που ξεκινά μια οδό απομεθυλίωσης του DNA.[51] Το EGR1, μαζί με το TET1, χρησιμοποιείται στον προγραμματισμό της κατανομής των θέσεων μεθυλίωσης στο DNA του εγκεφάλου κατά την ανάπτυξη του εγκεφάλου και στη μάθηση.
Δομή
[Επεξεργασία | επεξεργασία κώδικα]

Οι παράγοντες μεταγραφής είναι αρθρωτοί στη δομή και περιέχουν τις ακόλουθες περιοχές:[1]
- Περιοχή πρόσδεσης στο DNA ( DNA-binding domain, DBD), που συνδέεται σε συγκεκριμένες αλληλουχίες DNA ενισχυτή ή προαγωγέα. Απαραίτητο συστατικό για όλους τους φορείς Χρησιμοποιείται για την καθοδήγηση της μεταγραφής των αλληλουχιών του διαγονιδίου προαγωγέα του φορέα δίπλα σε ρυθμιζόμενα γονίδια. Οι αλληλουχίες DNA που δεσμεύουν παράγοντες μεταγραφής αναφέρονται συχνά ως στοιχεία απόκρισης.
- Η περιοχή ενεργοποίησης (Activation domain, AD), που περιέχει θέσεις δέσμευσης για άλλες πρωτεΐνες όπως συνρυθμιστές μεταγραφής. Αυτές οι τοποθεσίες δέσμευσης αναφέρονται συχνά ως συναρτήσεις ενεργοποίησης ( activation functions, AFs), πεδίο μετενεργοποίησης (Transactivation domain ή Trans-activating domain, TAD). Ο TAD, δεν πρέπει να συγχέεται με τον τοπολογικά συσχετιζόμενο τομέα (Topologically associating domain, TAD).[52]
- Μια προαιρετική περιοχή ανίχνευσης σήματος ( signal-sensing domain, SSD) (π.χ., μια περιοχή δέσμευσης συνδέτη), ο οποίος ανιχνεύει εξωτερικά σήματα και, ως απόκριση, μεταδίδει αυτά τα σήματα στους υπόλοιπους του συμπλέγματος μεταγραφής, με αποτέλεσμα τη ρύθμιση προς τα πάνω ή προς τα κάτω της γονιδιακής έκφρασης. Επίσης, οι περιοχές DBD και ανίχνευσης σήματος μπορεί να βρίσκονται σε ξεχωριστές πρωτεΐνες που συνδέονται εντός του συμπλέγματος μεταγραφής για να ρυθμίσουν την έκφραση του γονιδίου.
Περιοχή δέσμευσης DNA
[Επεξεργασία | επεξεργασία κώδικα]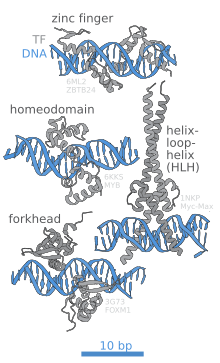
Το τμήμα της περιοχής του μεταγραφικού παράγοντα που δεσμεύει το DNA ονομάζεται περιοχή σύνδεσης του DNA. Παρακάτω είναι μια μερική λίστα μερικών από τις κύριες οικογένειες περιοχών σύνδεσης DNA/παραγόντων μεταγραφής:
| Οικογένεια | InterPro | Pfam | SCOP |
|---|---|---|---|
| βασικός έλικας-βρόχος-έλικας[53] | IPR001092 | PF00010 | 47460 |
| φερμουάρ βασικής λευκίνης (bZIP)[54] | IPR004827 | PF00170 | 57959 |
| Η τελική τελεστική περιοχή C των διμερών ρυθμιστών απόκρισης | IPR001789 | PF00072 | 46894 |
| AP2/ERF/GCC box | IPR001471 | PF00847 | 54176 |
| έλικας-στροφή-έλικας[55] | |||
| Οι πρωτεΐνες με ομοιοτικές περιοχές (homeodomain proteins), οι οποίες κωδικοποιούνται από τα γονίδια ομοιοπλαισίων (homeobox), είναι παράγοντες μεταγραφής. Οι πρωτεΐνες της ομοιοτικής περιοχής παίζουν κρίσιμους ρόλους στη ρύθμιση της ανάπτυξης.[56][57] | IPR009057 | PF00046 | 46689 |
| παρόμοιος καταστολέας λ | IPR010982 | 47413 | |
| παρόμοιος παράγοντας απόκρισης ορού (srf-like) | IPR002100 | PF00319 | 55455 |
| Συζευγμένο πλαίσιο (paired box)[58] | |||
| φτερωτός έλικας | IPR013196 | PF08279 | 46785 |
| Δάκτυλοι ψευδάργυρου [59] | |||
| * πολλαπλή περιοχή Cys2His2 δακτύλων ψευδαργύρου [60] | IPR007087 | PF00096 | 57667 |
| * Zn2/Cys6 | 57701 | ||
| * Zn2/Cys8 δάκτυλος ψευδαργύρου πυρηνικού υποδοχέα | IPR001628 | PF00105 | 57716 |
Στοιχεία απόκρισης
[Επεξεργασία | επεξεργασία κώδικα]Η αλληλουχία DNA στην οποία συνδέεται ένας παράγοντας μεταγραφής ονομάζεται θέση δέσμευσης του παράγοντα μεταγραφής, ή στοιχείο απόκρισης.[61]
Οι μεταγραφικοί παράγοντες αλληλεπιδρούν με τις θέσεις δέσμευσής τους χρησιμοποιώντας έναν συνδυασμό ηλεκτροστατικών δυνάμεων (που οι δεσμοί υδρογόνου είναι μια ειδική περίπτωση) και δυνάμεων Van der Waals. Λόγω της φύσης αυτών των χημικών αλληλεπιδράσεων, οι περισσότεροι μεταγραφικοί παράγοντες δεσμεύουν το DNA με έναν συγκεκριμένο τρόπο αλληλουχίας. Ωστόσο, δεν μπορεί πραγματικά να αλληλεπιδράσουν όλες οι βάσεις στη θέση δέσμευσης του μεταγραφικού παράγοντα με τον παράγοντα μεταγραφής. Επιπλέον, ορισμένες από αυτές τις αλληλεπιδράσεις μπορεί να είναι πιο αδύναμες από άλλες. Έτσι, οι παράγοντες μεταγραφής δεν δεσμεύουν μόνο μία αλληλουχία, αλλά είναι ικανοί να δεσμεύουν ένα υποσύνολο στενά συγγενών αλληλουχιών, καθεμία με διαφορετική ισχύ αλληλεπίδρασης.
Για παράδειγμα, αν και η συναινετική θέση δέσμευσης συναίνεσης για την πρωτεΐνη δέσμευσης ΤΑΤΑ (TATA-binding protein, TBP) είναι ΤΑΤΑΑΑΑ, ο παράγοντας μεταγραφής ΤΒΡ μπορεί επίσης να δεσμεύσει παρόμοιες αλληλουχίες όπως ΤΑΤΑΤΑΤ ή ΤΑΤΑΤΑΑ.
Επειδή οι παράγοντες μεταγραφής μπορούν να δεσμεύσουν ένα σύνολο σχετικών αλληλουχιών και αυτές οι αλληλουχίες τείνουν να είναι σύντομες, πιθανές θέσεις δέσμευσης μεταγραφικού παράγοντα μπορεί να προκύψουν τυχαία εάν η αλληλουχία DNA είναι αρκετά μεγάλη. Είναι απίθανο, ωστόσο, ένας παράγοντας μεταγραφής να δεσμεύσει όλες τις συμβατές αλληλουχίες στο γονιδίωμα του κυττάρου. Άλλοι περιορισμοί, όπως η προσβασιμότητα του DNA στο κύτταρο, ή η διαθεσιμότητα συμπαραγόντων μπορεί επίσης να βοηθήσουν στον καθορισμό της πραγματικής θέσης δέσμευσης ενός παράγοντα μεταγραφής. Έτσι, δεδομένης της αλληλουχίας του γονιδιώματος, είναι ακόμα δύσκολο να προβλεφθεί πού θα δεσμευτεί πραγματικά ένας μεταγραφικός παράγοντας σε ένα ζωντανό κύτταρο.
Επιπρόσθετη εξειδίκευση αναγνώρισης, ωστόσο, μπορεί να ληφθεί μέσω της χρήσης περισσότερων από μία περιοχών δέσμευσης DNA (για παράδειγμα διαδοχικά DBD στον ίδιο μεταγραφικό παράγοντα, ή μέσω διμερισμού δύο παραγόντων μεταγραφής) που συνδέονται με δύο ή περισσότερες γειτονικές αλληλουχίες DNA.
Κλινική σημασία
[Επεξεργασία | επεξεργασία κώδικα]Οι μεταγραφικοί παράγοντες έχουν κλινική σημασία για τουλάχιστον δύο λόγους: (1) οι μεταλλάξεις μπορεί να σχετίζονται με συγκεκριμένες ασθένειες και (2) μπορεί να είναι στόχοι φαρμάκων.
Διαταραχές
[Επεξεργασία | επεξεργασία κώδικα]Λόγω των σημαντικών ρόλων τους στην ανάπτυξη, τη διακυτταρική σηματοδότηση και τον κυτταρικό κύκλο, ορισμένες ανθρώπινες ασθένειες έχουν συσχετιστεί με μεταλλάξεις σε παράγοντες μεταγραφής .[62]
Πολλοί μεταγραφικοί παράγοντες είναι είτε ογκοκατασταλτικοί, είτε ογκογονίδια και, ως εκ τούτου, οι μεταλλάξεις, ή η ανώμαλη ρύθμιση τους συνδέονται με τον καρκίνο. Τρεις ομάδες μεταγραφικών παραγόντων είναι γνωστό ότι είναι σημαντικές στον ανθρώπινο καρκίνο: (1) οι οικογένειες NF-kappaB και AP-1, (2) η οικογένεια STAT και (3) οι υποδοχείς στεροειδών.[63]
Παρακάτω είναι μερικά από τα καλύτερα μελετημένα παραδείγματα:
| Κατάσταση | Περιγραφή | Θέση |
|---|---|---|
| Σύνδρομο Ρετ | Οι μεταλλάξεις στον μεταγραφικό παράγοντα MECP2 σχετίζονται με το σύνδρομο Ρετ, μια νευροαναπτυξιακή διαταραχή.[64][65] | Xq28 |
| Διαβήτης (ασθένεια) | Μια σπάνια μορφή διαβήτη που ονομάζεται (νεανικός διαβήτης όψιμης έναρξης) (Maturity onset diabetes of the young, MODY) μπορεί να προκληθεί από μεταλλάξεις στους πυρηνικούς παράγοντες ηπατοκυττάρων (hepatocyte nuclear factors, HNFs)[66], ή παράγοντας προαγωγέα ινσουλίνης-1 (IPF1/Pdx1).[67] | πολλαπλό |
| Αναπτυξιακή λεκτική δυσπραξία | Οι μεταλλάξεις στον μεταγραφικό παράγοντα FOXP2 σχετίζονται με αναπτυξιακή λεκτική δυσπραξία, μια ασθένεια στην οποία τα άτομα δεν είναι σε θέση να παράγουν τις λεπτώς συντονισμένες κινήσεις που απαιτούνται για την ομιλία.[68] | 7q31 |
| Αυτοάνοσες ασθένειες | Οι μεταλλάξεις στον μεταγραφικό παράγοντα FOXP3 προκαλούν μια σπάνια μορφή [[
Αυτοάνοσες ασθένειες|αυτοάνοσης ασθένειας]] το σύνδρομο ανοσοαπορύθμισης πολυενδοκρινοπάθειας – εντεροπάθειας που εξαρτάται από το χρωμόσωμα Χ (Immunodysregulation polyendocrinopathy enteropathy X-linked syndrome, IPEX).[69] |
Xp11.23-q13.3 |
| Σύνδρομο Λι-Φραουμένι | Caused by mutations in the tumor suppressor p53. Προκαλείται από μεταλλάξεις στην ογκοκατασταλτική πρωτεΐνη p53.[70] | 17p13.1 |
| Καρκίνος του μαστού | Η οικογένεια STAT σχετίζεται με τον καρκίνο του μαστού.[71] | πολλαπλός |
| Πολλαπλοί καρκίνοι | Η οικογένεια γονιδίων HOX εμπλέκεται σε μια ποικιλία καρκίνων.[72] | πολλαπλός |
| Οστεοαρθρίτιδα | Μετάλλαξη ή μειωμένη δραστηριότητα του SOX9 [73] |
Πιθανοί στόχοι φαρμάκων
[Επεξεργασία | επεξεργασία κώδικα]Περίπου το 10% των επί του παρόντος συνταγογραφούμενων φαρμάκων στοχεύουν άμεσα την κατηγορία μεταγραφικών παραγόντων πυρηνικοί υποδοχείς.[74] Τα παραδείγματα περιλαμβάνουν ταμοξιφαίνη και μπικαλουταμίδη για τη θεραπεία του καρκίνου του μαστού και του καρκίνου του προστάτη, αντίστοιχα, και διάφορων τύπων αντιφλεγμονωδών γλυκοκορτικοειδών και αναβολικών στεροειδών.[75]. Επιπλέον, οι παράγοντες μεταγραφής συχνά ρυθμίζονται έμμεσα από φάρμακα μέσω καταρρακτών σηματοδότησης. Ίσως είναι δυνατό να στοχευθούν άμεσα άλλοι λιγότερο εξερευνημένοι μεταγραφικοί παράγοντες, όπως NF-kB με φάρμακα.[76][77][78][79] Οι μεταγραφικοί παράγοντες εκτός της οικογένειας των πυρηνικών υποδοχέων πιστεύεται ότι είναι πιο δύσκολο να στοχευτούν με θεραπευτικά μικρά μόρια, καθώς δεν είναι σαφές ότι προσβάλλονται από φάρμακα, αλλά έχει σημειωθεί πρόοδος στο Pax2[80][81] και η οδός σηματοδότησης εντομής.[82]
Ρόλος στην εξέλιξη
[Επεξεργασία | επεξεργασία κώδικα]Οι διπλασιασμοί γονιδίων έχουν παίξει καθοριστικό ρόλο στην εξέλιξη των ειδών. Αυτό ισχύει ιδιαίτερα για τους παράγοντες μεταγραφής. Μόλις εμφανιστούν ως διπλότυπα, οι συσσωρευμένες μεταλλάξεις που κωδικοποιούν ένα αντίγραφο μπορούν να πραγματοποιηθούν χωρίς να επηρεαστεί αρνητικά η ρύθμιση των στόχων κατάντη. Ωστόσο, οι αλλαγές στις εξειδικεύσεις δέσμευσης DNA του παράγοντα μεταγραφής ενός αντιγράφου Leafy, ο οποίος εμφανίζεται στα περισσότερα φυτά της γης, έχουν πρόσφατα αποσαφηνιστεί. Από την άποψη αυτή, ένας παράγοντας μεταγραφής ενός αντιγράφου μπορεί να υποστεί αλλαγή εξειδίκευσης μέσω ενός ακατάστατου ενδιάμεσου χωρίς να χάσει τη λειτουργία του. Παρόμοιοι μηχανισμοί έχουν προταθεί στο πλαίσιο όλων των εναλλακτικών φυλογενετικών υποθέσεων και του ρόλου των μεταγραφικών παραγόντων στην εξέλιξη όλων των ειδών.[83][84]
Ρόλος στη δραστηριότητα βιοελέγχου
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής παίζουν ρόλο στη δραστηριότητα αντίστασης, η οποία είναι σημαντική για την επιτυχή δραστηριότητα βιοελέγχου. Η ανθεκτικότητα στην οξειδωτική καταπόνηση και η ανίχνευση του αλκαλικού pH συνεισφέρθηκαν από τους μεταγραφικούς παράγοντες Yap1 και Rim101 του Papiliotrema terrestris LS28 ως μοριακά εργαλεία και αποκάλυψαν την κατανόηση των γενετικών μηχανισμών που διέπουν την δραστηριότητα του βιοελέγχου που υποστηρίζει προγράμματα διαχείρισης ασθενειών που βασίζονται σε βιολογικό και ολοκληρωμένο έλεγχο.[85]
Ανάλυση
[Επεξεργασία | επεξεργασία κώδικα]Υπάρχουν διάφορες διαθέσιμες τεχνολογίες για την ανάλυση των παραγόντων μεταγραφής. Χρησιμοποιούνται συνήθως σε γονιδιωματικό επίπεδο, αλληλουχία DNA[86] και έρευνα βάσεων δεδομένων.[87] Η πρωτεϊνική έκδοση του παράγοντα μεταγραφής είναι ανιχνεύσιμη με τη χρήση ειδικών αντισωμάτων. Το δείγμα ανιχνεύεται σε στύπωμα western (western blot). Με τη χρήση δοκιμής μεταβολής ηλεκτροφορητικής κινητικότητας (electrophoretic mobility shift assay, EMSA),[88] μπορεί να ανιχνευθεί η κατατομή (προφίλ) ενεργοποίησης των παραγόντων μεταγραφής. Μια πολλαπλή προσέγγιση για τη δημιουργία κατατομής (προφίλ) ενεργοποίησης είναι ένα σύστημα τσιπ TF όπου μπορούν να ανιχνευθούν παράλληλα πολλοί διαφορετικοί παράγοντες μεταγραφής.
Η πιο συχνά χρησιμοποιούμενη μέθοδος για τον εντοπισμό θέσεων δέσμευσης μεταγραφικού παράγοντα είναι η ανοσοκαθίζηση χρωματίνης (chromatin immunoprecipitation, ChIP).[89] Αυτή η τεχνική βασίζεται στη χημική δέσμευση της χρωματίνης με φορμαλδεΰδη, ακολουθούμενη από ταυτόχρονη καθίζηση του DNA και του μεταγραφικού παράγοντα ενδιαφέροντος χρησιμοποιώντας ένα αντίσωμα που στοχεύει ειδικά αυτή την πρωτεΐνη. Οι αλληλουχίες DNA μπορούν στη συνέχεια να αναγνωριστούν με προσδιορισμό αλληλουχίας μικροσυστοιχίας ή υψηλής απόδοσης για τον προσδιορισμό των θέσεων δέσμευσης του παράγοντα μεταγραφής. Εάν δεν υπάρχει διαθέσιμο αντίσωμα για την πρωτεΐνη που μας ενδιαφέρει, η DamID μπορεί να είναι μια βολική εναλλακτική.[90]
Κατηγορίες
[Επεξεργασία | επεξεργασία κώδικα]Όπως περιγράφεται λεπτομερέστερα παρακάτω, οι παράγοντες μεταγραφής μπορούν να ταξινομηθούν με βάση τον (1) μηχανισμό δράσης τους, (2) τη ρυθμιστική τους λειτουργία ή (3) την ομολογία αλληλουχίας (και συνεπώς τη δομική ομοιότητα) στις περιοχές δέσμευσης DNA τους.
Μηχανιστικός
[Επεξεργασία | επεξεργασία κώδικα]Υπάρχουν δύο μηχανιστικές κατηγορίες παραγόντων μεταγραφής:
- Οι Γενικοί μεταγραφικοί παράγοντες εμπλέκονται στον σχηματισμό συμπλέγματος προεκκίνησης. Οι πιο κοινοί συντομεύονται ως TFIIA, TFIIB, TFIID, TFIIE, TFIIF και TFIIH. Είναι πανταχού παρόντες και αλληλεπιδρούν με την περιοχή του πυρήνα του προαγωγέα που περιβάλλει τις θέσεις έναρξης μεταγραφής όλων των γονιδίων κατηγορίας II.[91]
- Οι Ανοδικοί μεταγραφικοί παράγοντες είναι πρωτεΐνες που δεσμεύονται κάπου ανάντη της θέσης έναρξης για να διεγείρουν ή να καταστείλουν τη μεταγραφή. Αυτά είναι περίπου συνώνυμα με τους ειδικούς μεταγραφικούς παράγοντες, επειδή ποικίλλουν σημαντικά ανάλογα με το τι αλληλουχίες αναγνώρισης υπάρχουν στην εγγύτητα του γονιδίου.[92]
| Παραδείγματα συγκεκριμένων παραγόντων μεταγραφής [92] | |||
|---|---|---|---|
| Παράγοντας | Δομικός τύπος | Αλληλουχία αναγνώρισης | Δεσμεύει ως |
| SP1 | Δάκτυλος ψευδαργύρου | άκρο 5'-GGGCGG άκρο 3' | Μονομερές |
| AP-1 | Βασικό φερμουάρ | 5'-TGA(G/C)TCA-3' | Διμερές |
| C/EBP | Βασικό φερμουάρ | 5'-ATTGCGCAAT-3' | Διμερές |
| Παράγοντας θερμικού πλήγματος | Βασικό φερμουάρ | 5'-XGAAX-3' | Τριμερές |
| CREB | Βασικό φερμουάρ | 5'-TGACGTCA-3' | Διμερές |
| c-Myc | Βασικός έλικας-βρόχος-έλικας | 5'-CACGTG-3' | Διμερές |
| Oct-1 | Έλικας-στροφή-έλικας | 5'-ATGCAAAT-3' | Μονομερές |
| Πυρηνικός παράγοντας 1 | Πρωτότυπος | 5'-TTGGCXXXXXGCCAA-3' | Διμερές |
| (G/C) = G ή C X = A, T, G or C | |||
Λειτουργία
[Επεξεργασία | επεξεργασία κώδικα]Οι παράγοντες μεταγραφής έχουν ταξινομηθεί σύμφωνα με τη ρυθμιστική τους λειτουργία:[12]
- I. Συστατικώς ενεργοί – υπάρχουν σε όλα τα κύτταρα ανά πάσα στιγμή –γενικοί παράγοντες μεταγραφής, παράγοντας μεταγραφής Sp1, Πυρηνικός παράγοντας 1 (NF1), πρωτεΐνες που δεσμεύουν τον ενισχυτή Ccaat
- II. 'Υπό όρους ενεργοί - απαιτούν ενεργοποίηση
- II.A αναπτυξιακοί (ειδικοί για τα κύτταρα) – η έκφραση ελέγχεται αυστηρά, αλλά, αφού εκφραστεί, δεν απαιτεί πρόσθετη ενεργοποίηση – παράγοντας μεταγραφής GATA, πυρηνικοί παράγοντες ηπατοκυττάρων (HNF), PIT-1, MyoD, Myf5, γονίδιο Hox, παράγοντες μεταγραφής φτερωτού έλικα
- II.B εξαρτώμενοι από το σήμα – απαιτεί εξωτερικό σήμα για ενεργοποίηση
- II.B.1 εξωκυτταρικοί συνδέτες που εξαρτώνται από ενδοκρινικούς ή παρακρινείς - πυρηνικοί υποδοχείς
- II.B.2 ενδοκυτταρικοί συνδέτες εξαρτώμενοι από αυτοκρινείς – ενεργοποιούνται από μικρά ενδοκυτταρικά μόρια –SREBP, p53 , ορφανούς πυρηνικούς υποδοχείς
- II.B.3 εξαρτώμενοι από τον υποδοχέα της κυτταρικής μεμβράνης – καταρράκτες σηματοδότησης δεύτερου αγγελιοφόρου που καταλήγουν στη φωσφορυλίωση του μεταγραφικού παράγοντα
- II.B.3.a μόνιμοι πυρηνικοί παράγοντες – βρίσκονται στον πυρήνα ανεξάρτητα από την κατάσταση ενεργοποίησης – CREB, AP-1, Mef2
- II.B.3.b λανθάνοντες κυτταροπλασματικοί παράγοντες – η ανενεργή μορφή βρίσκεται στο κυτταρόπλασμα, αλλά, όταν ενεργοποιηθεί, μετατοπίζεται στον πυρήνα –STAT, R-SMAD, NF-κB, Notch, TUBBY, NFAT
Δομικοί
[Επεξεργασία | επεξεργασία κώδικα]Οι μεταγραφικοί παράγοντες ταξινομούνται συχνά με βάση την ομοιότητα αλληλουχίας και ως εκ τούτου την τριτοταγή δομή των περιοχών δέσμευσης του DNA τους:[93][11][94][10]
- 1 Υπερκατηγορία: Βασικές περιοχές
- 1.1 Κατηγορία: bZIP παράγοντες φερμουάρ λευκίνης
- 1.1.1 Οικογένεια: συστατικά παρόμοια με AP-1. Περιλαμβάνει c-Jun)
- 1.1.2 Οικογένεια: CREB
- 1.1.3 Οικογένεια: παράγοντες παρόμοιοι με C/EBP
- 1.1.4 Οικογένεια: bZIP / PAR
- 1.1.5 Οικογένεια: Φυτικοί συνδετικοί παράγοντες G-box
- 1.1.6 Οικογένεια: Μόνο ZIP
- 1.2 Κατηγορία: Παράγοντες έλικας-βρόχου-έλικας (bHLH)
- 1.2.1 Οικογένεια: Πανταχού παρόντες παράγοντες (κατηγορίας Α).
- 1.2.2 Οικογένεια: Μυογονικοί μεταγραφικοί παράγοντες (MyoD)
- 1.2.3 Οικογένεια: Achaete-Scute
- 1.2.4 Οικογένεια: Tal/Twist/Atonal/Hen
- 1.3 Κατηγορία: Έλικας-βρόχος-έλικας / συντελεστές φερμουάρ λευκίνης bHLH-ZIP
- 1.3.1 Οικογένεια: Πανταχού παρόντες παράγοντες bHLH-ZIP. Περιλαμβάνει USF (USF1, USF2); SREBP.
- 1.3.2 Οικογένεια: Παράγοντες ελέγχου του κυτταρικού κύκλου. Περιλαμβάνει c-Myc
- 1.4 Κατηγορία: NF-1
- 1.4.1 Οικογένεια: NF-1 (A, B, C, X)
- 1.5 Κατηγορία: RF-X
- 1.5.1 Οικογένεια: RF-X (1, 2, 3, 4, 5, ANK)
- 1.6 Κατηγορία: bHSH
- 1.1 Κατηγορία: bZIP παράγοντες φερμουάρ λευκίνης
- 2 Υπερκατηγορία: περιοχές σύνδεσης DNA που συντονίζουν τον ψευδάργυρο
- 2.1 Κατηγορία: Δάκτυλος ψευδάργυρου Cys4 τύπου πυρηνικού υποδοχέα
- 2.1.1 Οικογένεια: Υποδοχείς στεροειδών ορμονών
- 2.1.2 Οικογένεια: Παράγοντες που μοιάζουν με υποδοχείς θυρεοειδικών ορμονών
- 2.2 Κατηγορία: Ποικίλα δάκτυλα ψευδαργύρου Cys4
- 2.2.1 Οικογένεια: Παράγοντες GATA
- 2.3 Κατηγορία: Περιοχή δακτύλου ψευδαργύρου Cys2His2
- 2.3.1 Οικογένεια: Πανταχού παρόντες παράγοντες, περιλαμβάνει TFIIIA, Sp1
- 2.3.2 Οικογένεια: Ρυθμιστές αναπτυξιακού / κυτταρικού κύκλου. περιλαμβάνει Krüppel
- 2.3.4 Οικογένεια: Μεγάλοι παράγοντες με ιδιότητες δέσμευσης που μοιάζουν με NF-6B
- 2.4 Κατηγορία: Σύμπλεγμα κυστεΐνης-ψευδάργυρου Cys6
- 2.5 Κατηγορία: Δάχτυλα ψευδαργύρου εναλλασσόμενης σύνθεσης
- 2.1 Κατηγορία: Δάκτυλος ψευδάργυρου Cys4 τύπου πυρηνικού υποδοχέα
- 3 Υπερκατηγορία: Έλικας-στροφή-έλικας
- 3.1 Κατηγορία: Ομοιωτική περιοχή
- 3.1.1 Οικογένεια: Μόνο ομοιωτική περιοχή. Περιλαμβάνει Ubx
- 3.1.2 Οικογένεια: Παράγοντες περιοχής POU. Περιλαμβάνει Oct
- 3.1.3 Οικογένεια: Ομοιωτική περιοχή με περιοχή LIM
- 3.1.4 Οικογένεια: ομοιωτική περιοχή συν μοτίβα δακτύλων ψευδαργύρου
- 3.2 Κατηγορία: Συζευγμένο πλαίσιο
- 3.2.1 Οικογένεια: Σύζευξη και ομοιωτική περιοχή
- 3.2.2 Οικογένεια: Μόνο συζευγμένη περιοχή
- 3.3 Κατηγορία: Κεφαλή διχάλας / φτερωτός έλικας
- 3.3.1 Οικογένεια: Ρυθμιστές ανάπτυξης. περιλαμβάνει κεφαλές έλικα (forkhead)
- 3.3.2 Οικογένεια: Ειδικοί ρυθμιστές ιστών
- 3.3.3 Οικογένεια: Παράγοντες ελέγχου του κυτταρικού κύκλου
- 3.3.0 Οικογένεια: Άλλοι ρυθμιστές
- 3.4 Κατηγορία: Παράγοντες θερμικής καταπληξίας
- 3.4.1 Οικογένεια: HSF
- 3.5 Κατηγορία: Συστάδες τρυπτοφάνης
- 3.5.1 Οικογένεια: Myb
- 3.5.2 Οικογένεια: Τύπος Ets
- 3.5.3 Οικογένεια: Ρυθμιστικοί παράγοντες ιντερφερόνης
- 3.6 Κατηγορία: Περιοχή μεταγραφικών παραγόντων ενισχυτή (transcriptional enhancer factor, TEA)
- 3.6.1 Οικογένεια: TEA (TEAD1, TEAD2, TEAD3, TEAD4)
- 3.1 Κατηγορία: Ομοιωτική περιοχή
- 4 Υπερκατηγορία: Παράγοντες βήτα-ικριώματος με επαφές μικρής αύλακας
- 4.1 Κατηγορία: περιοχή ομολογίας Rel (Rel homology region, RHR)
- 4.1.1 Οικογένεια: Rel/αγκυρίνη. NF-kappaB
- 4.1.2 Οικογένεια: μόνο αγκυρίνη
- 4.1.3 Οικογένεια: Πυρηνικοί παράγοντες των ενεργοποιημένων Τ κυττάρων (NFAT, Nuclear Factor of Activated T-cells) ( NFATC1, NFATC2, NFATC3)
- 4.2 Κατηγορία: STAT
- 4.2.1 Οικογένεια: STAT
- 4.3 Κατηγορία: p53
- 4.3.1 Οικογένεις: p53
- 4.4 Κατηγορία: MADS box
- 4.4.1 Οικογένεια: Ρυθμιστές διαφοροποίησης. περιλαμβάνει (Mef2)
- 4.4.2 Οικογένεια: Αποκριτές σε εξωτερικά σήματα, παράγοντες απόκρισης ορού (serum response factor, SRF)
- 4.4.3 Οικογένεια: Μεταβολικοί ρυθμιστές (ARG80)
- 4.5 Κατηγορία: Μεταγραφικοί παράγοντες άλφα-έλικα βήτα-βαρελιού
- 4.6 Κατηγορία: Πρωτεΐνες σύνδεσης TATA
- 4.6.1 Οικογένεια: TBP
- 4.7 Κατηγορία: HMG-box
- 4.7.1 Οικογένεια: γονίδια SOX, SRY
- 4.7.2 Οικογένεια: TCF-1
- 4.7.3 Οικογένεια: σχετικές με HMG2, Ειδικές πρωτεΐνες αναγνώρισης δομής (Structure specific recognition protein 1, SSRP1)
- 4.7.4 Οικογένεια: UBF
- 4.7.5 Οικογένεια: MATA
- 4.8 Κατηγορία: Ετερομερείς παράγοντες CCAAT
- 4.8.1 Οικογένεια: Ετερομερείς παράγοντες CCAAT
- 4.9 Κατηγορία: Grainyhead
- 4.9.1 Οικογένεια: Grainyhead
- 4.10 Κατηγορία: Παράγοντες περιοχής κρυοπληξίας
- 4.10.1 Οικογένεια: csd
- 4.11 Κατηγορία: Runt
- 4.11.1 Οικογένεια: Runt
- 4.1 Κατηγορία: περιοχή ομολογίας Rel (Rel homology region, RHR)
- 0 Υπερκατηγορία: Άλλοι παράγοντες μεταγραφής
- 0.1 Κατηγορία: Πρωτεΐνες Copper fist
- 0.2 Κατηγορία: HMGI(Y)
- 0.2.1 Οικογένεια: HMGI(Y)
- 0.3 Κατηγορία: Περιοχή θύλακα (Pocket domain)
- 0.4 Κατηγορία: Παράγοντες που μοιάζουν με E1A
- 0.5 Κατηγορία: AP2/παράγοντες σχετικοί με EREBP
- 0.5.1 Οικογένεια: AP2
- 0.5.2 Οικογένεια: EREBP
- 0.5.3 Υπεροικογένεια: AP2/B3
- 0.5.3.1 Οικογένεια: ARF
- 0.5.3.2 Οικογένεια: ABI
- 0.5.3.3 Οικογένεια: RAV
Βάσεις δεδομένων παραγόντων μεταγραφής
[Επεξεργασία | επεξεργασία κώδικα]Υπάρχουν πολυάριθμες βάσεις δεδομένων που καταλογίζουν πληροφορίες σχετικά με τους παράγοντες μεταγραφής, αλλά το εύρος και η χρησιμότητά τους ποικίλλουν δραματικά. Ορισμένες μπορεί να περιέχουν μόνο πληροφορίες για τις πραγματικές πρωτεΐνες, κάποιες για τις θέσεις δέσμευσής τους ή για τα γονίδια-στόχους τους. Τα παραδείγματα περιλαμβάνουν τα ακόλουθα:
- http://floresta.eead.csic.es/footprintdb footprintDB -- μια βάση μεταδεδομένων πολλών βάσεων δεδομένων, συμπεριλαμβανομένης της JASPAR και άλλων
- JASPAR: βάση δεδομένων θέσεων δέσμευσης παραγόντων μεταγραφής για ευκαρυώτες
- http://planttfdb.gao-lab.org PlantTFD: Βάση δεδομένων παραγόντων μεταγραφής φυτών [95]
- TcoF-DB: Βάση δεδομένων συμπαραγόντων μεταγραφής και αλληλεπιδράσεων μεταγραφικών παραγόντων [96]
- http://tfcheckpoint.org TFcheckpoint: βάση δεδομένων υποψηφίων TF ανθρώπων, ποντικών και αρουραίων
- http://www.transcriptionfactor.org transcriptionfactor.org (τώρα εμπορική, πώληση αντιδραστηρίων)
- https://methmotif.org MethMotif.org: Μια ολοκληρωμένη βάση δεδομένων κυττάρου με μοτίβα δέσμευσης παραγόντων μεταγραφής σε συνδυασμό με κατατομές (προφίλ) μεθυλίωσης DNA.[97]
Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ 1,0 1,1 «Transcription factors: an overview». The International Journal of Biochemistry & Cell Biology 29 (12): 1305–12. December 1997. doi:. PMID 9570129. PMC 2002184. https://archive.org/details/sim_international-journal-of-biochemistry-cell-biology_1997-12_29_12/page/1305.
- ↑ «Too many transcription factors: positive and negative interactions». The New Biologist 2 (2): 126–31. February 1990. PMID 2128034.
- ↑ 3,0 3,1 «Structure and evolution of transcriptional regulatory networks». Current Opinion in Structural Biology 14 (3): 283–91. June 2004. doi:. PMID 15193307. Αρχειοθετήθηκε από το πρωτότυπο στις 2019-08-30. https://web.archive.org/web/20190830175950/https://www.mrc-lmb.cam.ac.uk/genomes/madanm/chalancon_chapter.pdf. Ανακτήθηκε στις 2024-02-15.
- ↑ How Genes are Regulated: Transcription Factors στο YouTube
- ↑ «The role of general initiation factors in transcription by RNA polymerase II». Trends in Biochemical Sciences 21 (9): 327–35. September 1996. doi:. PMID 8870495. https://archive.org/details/sim_trends-in-biochemical-sciences_1996-09_21_9/page/327.
- ↑ «RNA polymerase II transcription initiation: a structural view». Proceedings of the National Academy of Sciences of the United States of America 94 (1): 15–22. January 1997. doi:. PMID 8990153. Bibcode: 1997PNAS...94...15N.
- ↑ «Transcription of eukaryotic protein-coding genes». Annual Review of Genetics 34: 77–137. 2000. doi:. PMID 11092823.
- ↑ «Transcriptional regulation in mammalian cells by sequence-specific DNA binding proteins». Science 245 (4916): 371–8. July 1989. doi:. PMID 2667136. Bibcode: 1989Sci...245..371M.
- ↑ «Transcriptional activation by recruitment». Nature 386 (6625): 569–77. April 1997. doi:. PMID 9121580. Bibcode: 1997Natur.386..569P.
- ↑ 10,0 10,1 «PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors». Nucleic Acids Research 42 (Database issue): D1182-7. January 2014. doi:. PMID 24174544.
- ↑ 11,0 11,1 «TRANSFAC and its module TRANSCompel: transcriptional gene regulation in eukaryotes». Nucleic Acids Research 34 (Database issue): D108-10. January 2006. doi:. PMID 16381825.
- ↑ 12,0 12,1 12,2 «Signal transduction and the control of gene expression». Science 295 (5556): 813–8. February 2002. doi:. PMID 11823631. Bibcode: 2002Sci...295..813B.
- ↑ «Scaling laws in the functional content of genomes». Trends in Genetics 19 (9): 479–84. September 2003. doi:. PMID 12957540. https://archive.org/details/sim_trends-in-genetics_2003-09_19_9/page/479.
- ↑ «List Of All Transcription Factors In Human». biostars.org.
- ↑ «Regulation of the initiation of eukaryotic transcription». Essays in Biochemistry 37: 33–43. 2001. doi:. PMID 11758455.
- ↑ «Cooperation between complexes that regulate chromatin structure and transcription». Cell 108 (4): 475–87. February 2002. doi:. PMID 11909519.
- ↑ «Coactivator and corepressor complexes in nuclear receptor function». Current Opinion in Genetics & Development 9 (2): 140–7. April 1999. doi:. PMID 10322133.
- ↑ Robert O. J. Weinzierl (1999). Mechanisms of Gene Expression: Structure, Function and Evolution of the Basal Transcriptional Machinery
 . World Scientific Publishing Company. ISBN 1-86094-126-5.
. World Scientific Publishing Company. ISBN 1-86094-126-5.
- ↑ «Basal transcription factors». Current Opinion in Genetics & Development 13 (2): 114–8. April 2003. doi:. PMID 12672487.
- ↑ «The RNA polymerase II elongation complex». Annual Review of Biochemistry 72: 693–715. 2003. doi:. PMID 12676794. https://archive.org/details/sim_annual-review-of-biochemistry_2003_72/page/693.
- ↑ «The general transcription machinery and general cofactors». Critical Reviews in Biochemistry and Molecular Biology 41 (3): 105–78. 2006. doi:. PMID 16858867.
- ↑ Lobe CG (1992). Transcription factors and mammalian development. Current Topics in Developmental Biology. 27. σελίδες 351–83. doi:10.1016/S0070-2153(08)60539-6. ISBN 978-0-12-153127-0. PMID 1424766.
- ↑ «Genomic evolution of Hox gene clusters». Science 313 (5795): 1918–22. September 2006. doi:. PMID 17008523. Bibcode: 2006Sci...313.1918L.
- ↑ «Hox cofactors in vertebrate development». Developmental Biology 291 (2): 193–206. March 2006. doi:. PMID 16515781.
- ↑ «Determination and stability of sex». BioEssays 29 (1): 15–25. January 2007. doi:. PMID 17187356.
- ↑ «Signal transduction--a conserved pathway from the membrane to the nucleus». Developmental Genetics 14 (5): 333–8. 1993. doi:. PMID 8293575.
- ↑ «Estrogen receptor: current understanding of its activation and modulation». Clinical Cancer Research 7 (12 Suppl): 4338s–4342s; discussion 4411s–4412s. December 2001. PMID 11916222.
- ↑ «New insights into the mechanism of heat shock response activation». Cellular and Molecular Life Sciences 65 (6): 855–61. March 2008. doi:. PMID 18239856. https://archive.org/details/sim_cellular-and-molecular-life-sciences_2008-03_65_6/page/855.
- ↑ «The magic of the hypoxia-signaling cascade». Cellular and Molecular Life Sciences 65 (7–8): 1133–49. April 2008. doi:. PMID 18202826.
- ↑ «Maintaining cholesterol homeostasis: sterol regulatory element-binding proteins». World Journal of Gastroenterology 10 (21): 3081–7. November 2004. doi:. PMID 15457548.
- ↑ «Regulation of transcription factor activity during cellular aging». Biochemistry and Cell Biology 74 (4): 523–34. 1996. doi:. PMID 8960358. https://archive.org/details/sim_biochemistry-and-cell-biology_1996_74_4/page/523.
- ↑ «Regulation of gene expression and transcription factor binding activity during cellular aging». Biological Signals 5 (3): 130–8. 1996. doi:. PMID 8864058.
- ↑ «Integrated control of cell proliferation and cell death by the c-myc oncogene». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 345 (1313): 269–75. August 1994. doi:. PMID 7846125. Bibcode: 1994RSPTB.345..269E.
- ↑ «Xanthomonas AvrBs3 family-type III effectors: discovery and function». Annual Review of Phytopathology 48: 419–36. 2010. doi:. PMID 19400638. https://archive.org/details/sim_annual-review-of-phytopathology_2010_48/page/419.
- ↑ «A simple cipher governs DNA recognition by TAL effectors». Science 326 (5959): 1501. December 2009. doi:. PMID 19933106. Bibcode: 2009Sci...326.1501M.
- ↑ «Breaking the code of DNA binding specificity of TAL-type III effectors». Science 326 (5959): 1509–12. December 2009. doi:. PMID 19933107. Bibcode: 2009Sci...326.1509B.
- ↑ «Plant science. DNA binding made easy». Science 326 (5959): 1491–2. December 2009. doi:. PMID 20007890. Bibcode: 2009Sci...326.1491V.
- ↑ «A negative feedback loop of transcription factors that controls stem cell pluripotency and self-renewal». FASEB Journal 20 (10): 1730–2. August 2006. doi:. PMID 16790525. https://archive.org/details/sim_faseb-journal_2006-08_20_10/page/1730.
- ↑ 39,0 39,1 «Signal transduction and nuclear targeting: regulation of transcription factor activity by subcellular localisation». Journal of Cell Science 104 (4): 949–55. April 1993. doi:. PMID 8314906.
- ↑ «Transcription factor phosphorylation: a link between signal transduction and the regulation of gene expression». Cancer Cells 2 (11): 337–44. November 1990. PMID 2149275.
- ↑ «Steroid receptor phosphorylation: a key modulator of multiple receptor functions». Molecular Endocrinology 21 (10): 2311–9. October 2007. doi:. PMID 17536004.
- ↑ «Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities». Nucleic Acids Research 37 (17): 5641–55. September 2009. doi:. PMID 19625488.
- ↑ «Statistical-mechanical lattice models for protein-DNA binding in chromatin». Journal of Physics: Condensed Matter 22 (41): 414105. October 2010. doi:. PMID 21386588. Bibcode: 2010JPCM...22O4105T.
- ↑ «Choose your partners: dimerization in eukaryotic transcription factors». Trends in Biochemical Sciences 33 (5): 220–9. May 2008. doi:. PMID 18406148. https://archive.org/details/sim_trends-in-biochemical-sciences_2008-05_33_5/page/220.
- ↑ «Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?». BioEssays 31 (6): 629–41. June 2009. doi:. PMID 19382224.
- ↑ «Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome». Nat. Genet. 39 (4): 457–66. April 2007. doi:. PMID 17334365.
- ↑ «Gene body methylation can alter gene expression and is a therapeutic target in cancer». Cancer Cell 26 (4): 577–90. October 2014. doi:. PMID 25263941.
- ↑ «Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins». Nat. Biotechnol. 31 (12): 1137–42. December 2013. doi:. PMID 24108092. PMC 3858462. https://archive.org/details/sim_nature-biotechnology_2013-12_31_12/page/1137.
- ↑ «Impact of cytosine methylation on DNA binding specificities of human transcription factors». Science 356 (6337): eaaj2239. May 2017. doi:. PMID 28473536.
- ↑ «TET Enzymes and 5hmC in Adaptive and Innate Immune Systems». Front Immunol 10: 210. 2019. doi:. PMID 30809228.
- ↑ Sun Z, Xu X, He J, Murray A, Sun MA, Wei X, Wang X, McCoig E, Xie E, Jiang X, Li L, Zhu J, Chen J, Morozov A, Pickrell AM, Theus MH, Xie H. EGR1 recruits TET1 to shape the brain methylome during development and upon neuronal activity. Nat Commun. 2019 Aug 29;10(1):3892. doi: 10.1038/s41467-019-11905-3. PMID 31467272
- ↑ «Activation functions 1 and 2 of nuclear receptors: molecular strategies for transcriptional activation». Molecular Endocrinology 17 (10): 1901–9. October 2003. doi:. PMID 12893880.
- ↑ «Transcription factors 2: helix-loop-helix». Protein Profile 2 (6): 621–702. 1995. PMID 7553065.
- ↑ «Classification of human B-ZIP proteins based on dimerization properties». Molecular and Cellular Biology 22 (18): 6321–35. September 2002. doi:. PMID 12192032.
- ↑ «Structural classification of HTH DNA-binding domains and protein-DNA interaction modes». Journal of Molecular Biology 262 (2): 294–313. September 1996. doi:. PMID 8831795.
- ↑ «Homeodomain proteins». Annual Review of Biochemistry 63: 487–526. 1994. doi:. PMID 7979246. https://archive.org/details/sim_annual-review-of-biochemistry_1994_63/page/487.
- ↑ «Homeodomain proteins: an update». Chromosoma 125 (3): 497–521. June 2016. doi:. PMID 26464018.
- ↑ «Pax genes and organogenesis». BioEssays 19 (9): 755–65. September 1997. doi:. PMID 9297966.
- ↑ «Zinc finger proteins: new insights into structural and functional diversity». Current Opinion in Structural Biology 11 (1): 39–46. February 2001. doi:. PMID 11179890.
- ↑ «DNA recognition by Cys2His2 zinc finger proteins». Annual Review of Biophysics and Biomolecular Structure 29: 183–212. 2000. doi:. PMID 10940247.
- ↑ «Finding primary targets of transcriptional regulators». Cell Cycle 4 (3): 356–8. March 2005. doi:. PMID 15711128. http://www.landesbioscience.com/journals/cc/abstract.php?id=1521.
- ↑ Semenza, Gregg L. (1999). Transcription factors and human disease
 . Oxford [Oxfordshire]: Oxford University Press. ISBN 978-0-19-511239-9.
. Oxford [Oxfordshire]: Oxford University Press. ISBN 978-0-19-511239-9.
- ↑ «Targeting transcription factors for cancer gene therapy». Current Gene Therapy 6 (1): 17–33. February 2006. doi:. PMID 16475943.
- ↑ «MeCP2 dysfunction in Rett syndrome and related disorders». Current Opinion in Genetics & Development 16 (3): 276–81. June 2006. doi:. PMID 16647848.
- ↑ «MeCP2 in Rett syndrome: transcriptional repressor or chromatin architectural protein?». Current Opinion in Genetics & Development 17 (2): 121–5. April 2007. doi:. PMID 17317146. https://zenodo.org/record/1258987.
- ↑ Maestro MA, Cardalda C, Boj SF, Luco RF, Servitja JM, Ferrer J (2007). «Distinct Roles of HNF1 Β , HNF1 α , and HNF4 α in Regulating Pancreas Development, Β -Cell Function and Growth». Distinct roles of HNF1beta, HNF1alpha, and HNF4alpha in regulating pancreas development, beta-cell function and growth. Endocrine Development. 12. σελίδες 33–45. doi:10.1159/000109603. ISBN 978-3-8055-8385-5. PMID 17923767.
- ↑ «Pancreatic duodenal homeobox factor-1 and diabetes mellitus type 2 (review)». International Journal of Molecular Medicine 21 (4): 399–404. April 2008. doi:. PMID 18360684. http://www.spandidos-publications.com/ijmm/article.jsp?article_id=ijmm_21_4_399.
- ↑ «Deletion of 7q31.1 supports involvement of FOXP2 in language impairment: clinical report and review». American Journal of Medical Genetics. Part A 143A (8): 791–8. April 2007. doi:. PMID 17330859.
- ↑ «IPEX as a result of mutations in FOXP3». Clinical & Developmental Immunology 2007: 1–5. 2007. doi:. PMID 18317533.
- ↑ «Li-Fraumeni syndrome: a p53 family affair». Cell Cycle 4 (7): 865–7. July 2005. doi:. PMID 15917654.
- ↑ «Roles and Regulation of Stat Family Transcription Factors in Human Breast Cancer». American Journal of Pathology 165 (5): 1449–1460. November 2004. doi:. PMID 15509516.
- ↑ «"Transcription factors as targets and markers in cancer" Workshop 2007». Αρχειοθετήθηκε από το πρωτότυπο στις 25 Μαΐου 2012. Ανακτήθηκε στις 14 Δεκεμβρίου 2009.
- ↑ «Changes in Fluorescence Recovery After Photobleaching (FRAP) as an indicator of SOX9 transcription factor activity». Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms 1862 (1): 107–117. January 2019. doi:. PMID 30465885.
- ↑ «How many drug targets are there?». Nature Reviews. Drug Discovery 5 (12): 993–6. December 2006. doi:. PMID 17139284.
- ↑ «Principles for modulation of the nuclear receptor superfamily». Nature Reviews. Drug Discovery 3 (11): 950–64. November 2004. doi:. PMID 15520817.
- ↑ «Transcription factors: targets for new designer drugs». British Journal of Biomedical Science 51 (2): 147–57. June 1994. PMID 8049612. https://archive.org/details/sim_british-journal-of-biomedical-science_1994-06_51_2/page/147.
- ↑ «Transcription factors as drug targets: opportunities for therapeutic selectivity». Gene Expression 4 (6): 319–36. 1995. PMID 7549464.
- ↑ «Transcription-factor-modulating agents: precision and selectivity in drug design». Molecular Medicine Today 4 (8): 358–66. August 1998. doi:. PMID 9755455.
- ↑ «Transcription factor therapeutics: long-shot or lodestone». Current Medicinal Chemistry 12 (6): 691–701. 2005. doi:. PMID 15790306.
- ↑ «Inhibition of Pax2 Transcription Activation with a Small Molecule that Targets the DNA Binding Domain». ACS Chemical Biology 12 (3): 724–734. 2017. doi:. PMID 28094913.
- ↑ «Are Pax proteins potential therapeutic targets in kidney disease and cancer?». Kidney International 94 (2): 259–267. 2018. doi:. PMID 29685496.
- ↑ «Direct inhibition of the NOTCH transcription factor complex». Nature 462 (7270): 182–8. November 2009. doi:. PMID 19907488. Bibcode: 2009Natur.462..182M.
- ↑ «A promiscuous intermediate underlies the evolution of LEAFY DNA binding specificity». Science 343 (6171): 645–8. February 2014. doi:. PMID 24436181. Bibcode: 2014Sci...343..645S. https://zenodo.org/record/3437650.[νεκρός σύνδεσμος]
- ↑ «An Arabidopsis Transcriptional Regulatory Map Reveals Distinct Functional and Evolutionary Features of Novel Transcription Factors». Molecular Biology and Evolution 32 (7): 1767–73. July 2015. doi:. PMID 25750178.
- ↑ «Molecular Tools for the Yeast Papiliotrema terrestris LS28 and Identification of Yap1 as a Transcription Factor Involved in Biocontrol Activity». Applied and Environmental Microbiology 87 (7). March 2021. doi:. PMID 33452020. Bibcode: 2021ApEnM..87E2910C.
- ↑ «PubMed». PubMed.
- ↑ «VOMBAT: prediction of transcription factor binding sites using variable order Bayesian trees». Nucleic Acids Research 34 (Web Server issue): W529-33. July 2006. doi:. PMID 16845064. PMC 1538886. Αρχειοθετήθηκε από το πρωτότυπο στις 2018-09-30. https://web.archive.org/web/20180930084306/http://www.eng.tau.ac.il/~bengal/VOMBAT.pdf. Ανακτήθηκε στις 2024-02-15.
- ↑ «Tyrosine phosphorylation regulates the partitioning of STAT1 between different dimer conformations». Proceedings of the National Academy of Sciences of the United States of America 105 (27): 9238–43. July 2008. doi:. PMID 18591661. Bibcode: 2008PNAS..105.9238W.
- ↑ «ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions». Nature Reviews. Genetics 13 (12): 840–52. December 2012. doi:. PMID 23090257.
- ↑ «Dam it's good! DamID profiling of protein-DNA interactions». Wiley Interdisciplinary Reviews: Developmental Biology 5 (1): 25–37. January 2016. doi:. PMID 26383089.
- ↑ «The general transcription factors of RNA polymerase II». Genes & Development 10 (21): 2657–83. November 1996. doi:. PMID 8946909.
- ↑ 92,0 92,1 Boron WF (2003). Medical Physiology: A Cellular And Molecular Approaoch. Elsevier/Saunders. σελίδες 125–126. ISBN 1-4160-2328-3.
- ↑ «Systematic DNA-binding domain classification of transcription factors». Genome Informatics. International Conference on Genome Informatics 15 (2): 276–86. 2004. PMID 15706513. Αρχειοθετήθηκε από το πρωτότυπο στις 2013-06-19. https://web.archive.org/web/20130619202726/http://www.jsbi.org/journal/GIW04/GIW04F028.html. Ανακτήθηκε στις 2024-02-15.
- ↑ «TRANSFAC database». Ανακτήθηκε στις 5 Αυγούστου 2007.
- ↑ «PlantTFDB 4.0: toward a central hub for transcription factors and regulatory interactions in plants». Nucleic Acids Research 45 (D1): D1040–D1045. January 2017. doi:. PMID 27924042.
- ↑ «TcoF-DB v2: update of the database of human and mouse transcription co-factors and transcription factor interactions». Nucleic Acids Research 45 (D1): D145–D150. January 2017. doi:. PMID 27789689.
- ↑ «MethMotif: an integrative cell specific database of transcription factor binding motifs coupled with DNA methylation profiles». Nucleic Acids Research 47 (D1): D145–D154. January 2019. doi:. PMID 30380113.
Παραπέρα μελέτη
[Επεξεργασία | επεξεργασία κώδικα]- Carretero-Paulet, Lorenzo; Galstyan, Anahit; Roig-Villanova, Irma; Martínez-García, Jaime F.; Bilbao-Castro, Jose R. «Genome-Wide Classification and Evolutionary Analysis of the bHLH Family of Transcription Factors in Arabidopsis, Poplar, Rice, Moss, and Algae». Plant Physiology, 153, 3, 2010-07, pàg. 1398–1412. doi:10.1104/pp.110.153593. ISSN 0032-0889
- «An Arabidopsis Transcriptional Regulatory Map Reveals Distinct Functional and Evolutionary Features of Novel Transcription Factors». Molecular Biology and Evolution 32 (7): 1767–73. 2015. doi:. PMID 25750178.
- «The Human Transcription Factors». Cell 172 (4): 650–665. 2018. doi:. PMID 29425488.
Εξωτερικοί σύνδεσμοι
[Επεξεργασία | επεξεργασία κώδικα]- Transcription factor database Αρχειοθετήθηκε 4 December 2008 στο Wayback Machine.
