Ιστόνη

Στη βιολογία, οι ιστόνες είναι πολύ βασικές πρωτεΐνες πλούσιες σε υπολείμματα λυσίνης και αργινίνης που βρίσκονται σε ευκαρυωτικούς κυτταρικούς πυρήνες και στις περισσότερες συνομοταξίες αρχαίων. Λειτουργούν ως καρούλια γύρω από τα οποία τυλίγεται το DNA για να δημιουργήσει δομικές μονάδες που ονομάζονται νουκλεοσώματα.[1][2] Τα νουκλεοσώματα με τη σειρά τους τυλίγονται σε ίνες 30-νανομέτρων που σχηματίζουν σφιχτά στοιβαγμένη χρωματίνη. Οι ιστόνες εμποδίζουν το DNA να μπλέξει και το προστατεύουν από βλάβη. Επιπλέον, οι ιστόνες παίζουν σημαντικό ρόλο στη γονιδιακή ρύθμιση και την αντιγραφή του DNA. Χωρίς ιστόνες, το ξετυλιγμένο DNA στα χρωμοσώματα θα ήταν πολύ μακρύ. Για παράδειγμα, κάθε ανθρώπινο κύτταρο έχει περίπου 1,8 μέτρα DNA εάν τεντωθεί τελείως. Ωστόσο, όταν τυλίγεται γύρω από ιστόνες, αυτό το μήκος μειώνεται σε περίπου 90 μικρόμετρα (0,09 mm) ινών χρωματίνης διαμέτρου 30 nm.[3]
Υπάρχουν πέντε οικογένειες ιστονών που ονομάζονται Η1/Η5 (συνδετικές ιστόνες), Η2, Η3 και Η4 (πυρηνικές ιστόνες). Ο πυρήνας του νουκλεοσώματος σχηματίζεται από δύο Η2Α-Η2Β διμερή και ένα Η3-Η4 τετραμερές. Το σφιχτό τύλιγμα του DNA γύρω από τις ιστόνες είναι σε μεγάλο βαθμό αποτέλεσμα της ηλεκτροστατικής έλξης μεταξύ των θετικά φορτισμένων ιστονών και του αρνητικά φορτισμένου φωσφορικού σκελετού του DNA.
Οι ιστόνες μπορεί να τροποποιηθούν χημικά μέσω της δράσης ενζύμων για τη ρύθμιση της γονιδιακής μεταγραφής. Η πιο κοινή τροποποίηση είναι η μεθυλίωση υπολειμμάτων αργινίνης ή λυσίνης, ή η ακετυλίωση λυσίνης. Η μεθυλίωση μπορεί να επηρεάσει τον τρόπο με τον οποίο άλλες πρωτεΐνες όπως οι παράγοντες μεταγραφής αλληλεπιδρούν με τα νουκλεοσώματα. Η ακετυλίωση της λυσίνης εξαλείφει ένα θετικό φορτίο στη λυσίνη, αποδυναμώνοντας έτσι την ηλεκτροστατική έλξη μεταξύ ιστόνης και DNA με αποτέλεσμα μερικό ξετύλιγμα του DNA καθιστώντας το πιο προσιτό για γονιδιακή έκφραση.
Τάξεις και παραλλαγές[Επεξεργασία | επεξεργασία κώδικα]

Υπάρχουν πέντε μεγάλες οικογένειες πρωτεϊνών ιστόνης: H1/H5, H2A, H2B, H3, και H4.[2][4][5][6] Οι ιστόνες H2A, H2B, H3 και H4 είναι γνωστές ως ιστόνες πυρήνα ή νουκλεοσωμικές, ενώ οι ιστόνες H1/H5 είναι γνωστές ως ιστόνες σύνδεσης.
Οι ιστόνες του πυρήνα υπάρχουν όλες ως διμερείς, που είναι παρόμοιες στο ότι διαθέτουν όλες τον τομέα πτυχής ιστόνης: τρεις άλφα έλικες που συνδέονται με δύο βρόχους. Είναι αυτή η ελικοειδής δομή που επιτρέπει την αλληλεπίδραση μεταξύ διαφορετικών διμερών, ιδιαίτερα με τον τρόπο της κεφαλής-ουράς (ονομάζεται επίσης μοτίβο χειραψίας).[7] Τα προκύπτοντα τέσσερα διακριτά διμερή ενώνονται στη συνέχεια για να σχηματίσουν έναν οκταμερή πυρήνα νουκλεοσώματος, περίπου 63 Angstroms σε διάμετρο (ένα σωματίδιο που μοιάζει με σωληνοειδές DNA). Περίπου 146 ζεύγη βάσεων (base pairs, bp) DNA τυλίγονται γύρω από αυτό το σωματίδιο του πυρήνα 1,65 φορές σε μια αριστερόστροφη υπερελικοειδή στροφή για να δώσουν ένα σωματίδιο διαμέτρου περίπου 100 Angstroms.[8] Η συνδετική ιστόνη Η1 δεσμεύει το νουκλεόσωμα στις θέσεις εισόδου και εξόδου του DNA, κλειδώνοντας έτσι το DNA στη θέση του [9] και επιτρέποντας το σχηματισμό δομής ανώτερης τάξης. Ο πιο βασικός τέτοιος σχηματισμός είναι η ίνα των 10&nm ή οι χάντρες σε μια διαμόρφωση χορδής. Αυτό περιλαμβάνει το τύλιγμα του DNA γύρω από τα νουκλεοσώματα με περίπου 50 ζεύγη βάσεων DNA που διαχωρίζουν κάθε ζεύγος νουκλεοσωμάτων (αναφέρεται επίσης ως συνδέτης DNA). Οι δομές υψηλότερης τάξης περιλαμβάνουν την ίνα των 30 nm (που σχηματίζει ένα ακανόνιστο ζιγκ-ζαγκ) και την ίνα των 100 nm, αυτές είναι οι δομές που βρίσκονται στα φυσιολογικά κύτταρα. Κατά τη διάρκεια της μίτωσης και της μείωσης, τα συμπυκνωμένα χρωμοσώματα συγκροτούνται μέσω αλληλεπιδράσεων μεταξύ νουκλεοσωμάτων και άλλων ρυθμιστικών πρωτεϊνών.
Οι ιστόνες υποδιαιρούνται σε κανονικές ιστόνες εξαρτώμενες από την αντιγραφή, των οποίων τα γονίδια εκφράζονται κατά τη φάση S του κυτταρικού κύκλου και ανεξάρτητες από την αντιγραφή παραλλαγές ιστονών, που εκφράζονται κατά τη διάρκεια ολόκληρου του κυτταρικού κύκλου. Στα θηλαστικά, τα γονίδια που κωδικοποιούν κανονικές ιστόνες συγκεντρώνονται συνήθως κατά μήκος των χρωμοσωμάτων σε 4 διαφορετικούς τόπους υψηλά διατηρημένους, στερούνται εσωνίων και χρησιμοποιούν μια δομή βρόχου στελέχους στο άκρο 3'. αντί ουράς πολυΑ. Τα γονίδια που κωδικοποιούν παραλλαγές ιστονών δεν είναι συνήθως συγκεντρωμένα, έχουν εσώνια και τα mRNA τους ρυθμίζονται με ουρές πολυΑ.[10] Οι σύνθετοι πολυκύτταροι οργανισμοί έχουν συνήθως μεγαλύτερο αριθμό παραλλαγών ιστονών που παρέχουν μια ποικιλία διαφορετικών λειτουργιών. Πρόσφατα δεδομένα συσσωρεύονται σχετικά με τους ρόλους διαφορετικών παραλλαγών ιστονών που υπογραμμίζουν τους λειτουργικούς δεσμούς μεταξύ των παραλλαγών και τη λεπτή ρύθμιση της ανάπτυξης του οργανισμού.[11] Οι πρωτεΐνες παραλλαγών ιστονών από διαφορετικούς οργανισμούς, η ταξινόμησή τους και τα ειδικά χαρακτηριστικά της παραλλαγής βρίσκονται στη βάση δεδομένων "HistoneDB 2.0 - Variants".[12][13] Αρκετά ψευδογονίδια έχουν επίσης ανακαλυφθεί και ταυτοποιηθεί σε πολύ στενές αλληλουχίες των αντίστοιχων λειτουργικών ορθολογικών γονιδίων τους.[14][15]
The following is a list of human histone proteins, genes and pseudogenes: Ακολουθεί ένας κατάλογος με πρωτεΐνες, γονίδια και ψευδογονίδια ανθρώπινης ιστόνης:[10]
| Υπεροικογένεια | Οικογένεια | Γονίδια που εξαρτώνται από την αντιγραφή | Γονίδια ανεξάρτητα από την αντιγραφή | Ψευδογονίδια |
|---|---|---|---|---|
| Συνδέτης | H1 | H1-1, H1-2, H1-3, H1-4, H1-5, H1-6 | H1-0, H1-7, H1-8, H1-10 | H1-9P, H1-12P |
| Πυρήνας | H2A | H2AC1, H2AC4, H2AC6, H2AC7, H2AC8, H2AC11, H2AC12, H2AC13, H2AC14, H2AC15, H2AC16, H2AC17, H2AC18, H2AC19, H2AC20, H2AC21, H2AC25 | H2AZ1, H2AZ2, MACROH2A1, MACROH2A2, H2AX, H2AJ, H2AB1, H2AB2, H2AB3, H2AP, H2AL1Q, H2AL3 | H2AC2P, H2AC3P, H2AC5P, H2AC9P, H2AC10P, H2AQ1P, H2AL1MP |
| H2B | H2BC1, H2BC3, H2BC4, H2BC5, H2BC6, H2BC7, H2BC8, H2BC9, H2BC10, H2BC11, H2BC12, H2BC13, H2BC14, H2BC15, H2BC17, H2BC18, H2BC21, H2BC26, H2BC12L | H2BK1, H2BW1, H2BW2, H2BW3P, H2BN1 | H2BC2P, H2BC16P, H2BC19P, H2BC20P, H2BC27P, H2BL1P, H2BW3P, H2BW4P | |
| H3 | H3C1, H3C2, H3C3, H3C4, H3C6, H3C7, H3C8, H3C10, H3C11, H3C12, H3C13, H3C14, H3C15, H3-4 | H3-3A, H3-3B, H3-5, H3-7, H3Y1, H3Y2, CENPA | H3C5P, H3C9P, H3P16, H3P44 | |
| H4 | H4C1, H4C2, H4C3, H4C4, H4C5, H4C6, H4C7, H4C8, H4C9, H4C11, H4C12, H4C13, H4C14, H4C15 | H4C16 | H4C10P |
Δομή[Επεξεργασία | επεξεργασία κώδικα]

Ο πυρήνας νουκλεοσώματος σχηματίζεται από δύο διμερή H2A-H2B και ένα τετραμερές H3-H4, σχηματίζοντας δύο σχεδόν συμμετρικά μισά από τριτογενή δομή ]] συμμετρίας C2. Το ένα μακρομόριο είναι η κατοπτρική εικόνα του άλλου).[8] Τα διμερή Η2Α-Η2Β και το τετραμερές Η3-Η4 παρουσιάζουν επίσης ψευδοδυαδική συμμετρία. Οι 4 ιστόνες 'πυρήνα' (H2A, H2B, H3 και H4) είναι σχετικά παρόμοιες στη δομή και διατηρούνται σε μεγάλο βαθμό μέσω εξέλιξης, ενώ όλες διαθέτουν ένα μοτίβο 'έλικα στροφή έλικα στροφή έλικα (πρωτεϊνικό μοτίβο δέσμευσης DNA που αναγνωρίζει τη συγκεκριμένη αλληλουχία DNA). Μοιράζονται επίσης το χαρακτηριστικό των μακριών 'ουρών' στο ένα άκρο της δομής αμινοξέ4ος - αυτή είναι η θέση της μετα-μεταφραστικής τροποποίησης (βλ. παρακάτω).[16]
Η αρχαϊκή ιστόνη περιέχει μόνο διμερή δομή παρόμοια με H3-H4 που αποτελείται από έναν μόνο τύπο μονάδας. Τέτοιες διμερείς δομές μπορούν να στοιβάζονται σε μια ψηλή υπερέλικα ("υπερνουκλεόσωμα") πάνω στην οποία το DNA περιελίσσεται με τρόπο παρόμοιο με τα καρούλια του νουκλεοσώματος.[17] Μόνο μερικές αρχαϊκές ιστόνες έχουν ουρές.[18]
Η απόσταση μεταξύ των καρουλιών γύρω από τα οποία τα ευκαρυωτικά κύτταρα τυλίγουν το DNA τους έχει προσδιοριστεί ότι κυμαίνεται από 59 έως 70 Å.[19]
Συνολικά, οι ιστόνες κάνουν πέντε τύπους αλληλεπιδράσεων με το DNA:
- Γέφυρες άλατος και δεσμοί υδρογόνου μεταξύ πλευρικών αλυσίδων βασικών αμινοξέων (ιδιαίτερα λυσίνης και αργινίνης) και φωσφορικών οξυγόνων στο DNA
- Τα δίπολα έλικας σχηματίζουν άλφα-έλικες στα Η2Β, Η3 και Η4 που προκαλούν τη συσσώρευση καθαρού θετικού φορτίου στο σημείο αλληλεπίδρασης με αρνητικά φορτισμένες φωσφορικές ομάδες στο DNA
- Δεσμοί υδρογόνου μεταξύ της σκελετού του DNA και της αμιδικής ομάδας στην κύρια αλυσίδα των πρωτεϊνικών ιστονών
- Μη πολικές αλληλεπιδράσεις μεταξύ των σακχάρων ιστονών και δεοξυριβόζης στο DNA
- Μη ειδικές δευτερεύουσες αυλακώσεις των Ν-τερματικών ουρών H3 και H2B σε δύο δευτερεύουσες αυλακώσεις καθεμιάς στο μόριο DNA
Η εξαιρετικά βασική φύση των ιστονών, εκτός από τη διευκόλυνση των αλληλεπιδράσεων DNA-ιστονών, συμβάλλει στη διαλυτότητά τους στο νερό.
Οι ιστόνες υπόκεινται σε μετα-μεταφραστική τροποποίηση από ένζυμα κυρίως στις Ν-τελικές ουρές τους, αλλά και στις σφαιρικές περιοχές τους.[20][21] Τέτοιες τροποποιήσεις περιλαμβάνουν μεθυλίωση, κιτρουλλινοποίηση, ακετυλίωση, φωσφορυλίωση, SUMOποίηση, ουβικιτινοποίηση και ριβοζυλίωση ADP. Αυτό επηρεάζει τη γονιδιακή τους ρύθμιση.
Γενικά, τα γονίδια που είναι ενεργά έχουν λιγότερο δεσμευμένη ιστόνη, ενώ τα ανενεργά γονίδια συνδέονται σε μεγάλο βαθμό με τις ιστόνες κατά τη μεσόφαση.[22] Φαίνεται επίσης ότι η δομή των ιστονών έχει εξελικτικά διατηρηθεί, καθώς οποιεσδήποτε επιβλαβείς μεταλλάξεις θα ήταν σοβαρά δυσπροσαρμοστικές. Όλες οι ιστόνες έχουν ένα πολύ θετικά φορτισμένο Ν-άκρο με πολλά υπολείμματα λυσίνης και αργινίνης.
Εξέλιξη και κατανομή ειδών[Επεξεργασία | επεξεργασία κώδικα]
Οι ιστόνες του πυρήνα βρίσκονται στον πυρήνα των ευκαρυωτικών κυττάρων και στα περισσότερα αρχαϊκά φύλα, αλλά όχι σε βακτήρια.[18] Τα μονοκύτταρα φύκια που είναι γνωστά ως δινοφύκη θεωρούνταν προηγουμένως ότι είναι οι μόνοι ευκαρυώτες που στερούνται εντελώς ιστόνων,[23] αλλά μεταγενέστερες μελέτες έδειξαν ότι το DNA τους εξακολουθεί να κωδικοποιεί γονίδια ιστόνης.[24] Σε αντίθεση με τις ιστόνες του πυρήνα, ομόλογα των πρωτεϊνών συνδέτη ιστόνης (H1) που είναι πλούσια σε λυσίνη βρίσκονται σε βακτήρια, γνωστά αλλιώς ως νουκλεοπρωτεΐνες HC1/HC2.[25]
Έχει προταθεί ότι οι πρωτεΐνες του πυρήνα των ιστονών σχετίζονται εξελικτικά με το ελικοειδή τμήμα της εκτεταμένης περιοχής ΑΑΑ+ ΑΤΡάσης, την περιοχή C και με την περιοχή αναγνώρισης του Ν-τερματικού υποστρώματος των πρωτεϊνών Clp/Hsp100. Παρά τις διαφορές στην τοπολογία τους, αυτές οι τρεις πτυχές μοιράζονται ένα ομόλογο μοτίβο έλικας-κλώνου-έλικας (helix-strand-helix, HSH).[16] Προτείνεται επίσης ότι μπορεί να έχουν εξελιχθεί από τις ριβοσωματικές πρωτεΐνες (RPS6/RPS15), που και οι δυο είναι βραχείες και βασικές πρωτεΐνες.[26]
Οι αρχαϊκές ιστόνες μπορεί κάλλιστα να μοιάζουν με τους εξελικτικούς πρόδρομους των ευκαρυωτικών ιστόνων.[18] Οι πρωτεΐνες ιστονών είναι από τις πιο συντηρημένες πρωτεΐνες στους ευκαρυώτες, τονίζοντας τον σημαντικό ρόλο τους στη βιολογία του πυρήνα.[2] Αντίθετα, τα ώριμα σπερματοζωάρια χρησιμοποιούν σε μεγάλο βαθμό πρωταμίνες για να συσκευάσουν το γονιδιωματικό τους DNA, πιθανότατα επειδή αυτό τους επιτρέπει να επιτύχουν ακόμη υψηλότερό λόγο συσκευασίας.[27]
Υπάρχουν ορισμένες μορφές παραλλαγής σε ορισμένες από τις κύριες τάξεις. Μοιράζονται την ομολογία της αλληλουχίας αμινοξέων και τη δομική ομοιότητα του πυρήνα με μια συγκεκριμένη τάξη κύριων ιστονών, αλλά έχουν επίσης το δικό τους χαρακτηριστικό που διαφέρει από τις κύριες ιστόνες. Αυτές οι δευτερεύουσες ιστόνες συνήθως εκτελούν συγκεκριμένες λειτουργίες του μεταβολισμού της χρωματίνης. Για παράδειγμα, η ιστόνη τύπου H3 CENPA σχετίζεται μόνο με την κεντρομεριδιακή περιοχή του χρωμοσώματος. Η παραλλαγή της ιστόνης H2A H2A.Z σχετίζεται με τους προαγωγείς ενεργά μεταγραφόμενων γονιδίων και επίσης εμπλέκεται στην πρόληψη της εξάπλωσης της σιωπηλής ετεροχρωματίνης.[28] Επιπλέον, το H2A.Z έχει ρόλους στη χρωματίνη για τη σταθερότητα του γονιδιώματος.[29] Μια άλλη παραλλαγή της H2A η H2A.X φωσφορυλιώνεται στο S139 σε περιοχές γύρω από τα σπασίματα του διπλού κλώνου και επισημαίνει την περιοχή που υφίσταται επιδιόρθωση του DNA.[30] Η ιστόνη H3.3 σχετίζεται με το σώμα των ενεργά μεταγραφόμενων γονιδίων.[31]
Τροποποιήσεις ιστόνης[Επεξεργασία | επεξεργασία κώδικα]

Συμπίεση κλώνων DNA[Επεξεργασία | επεξεργασία κώδικα]
Οι ιστόνες λειτουργούν ως καρούλια γύρω από τα οποία τυλίγεται το DNA. Αυτό επιτρέπει τη συμπίεση που είναι απαραίτητη για να χωρέσουν τα μεγάλα γονιδιώματα των ευκαρυωτών μέσα στους κυτταρικούς πυρήνες: το συμπιεσμένο μόριο είναι 40.000 φορές μικρότερο από ένα μη συσκευασμένο μόριο.
Ρύθμιση χρωματίνης[Επεξεργασία | επεξεργασία κώδικα]

Οι ιστόνες υφίστανται μετα-μεταφραστικές τροποποιήσεις που αλλάζουν την αλληλεπίδρασή τους με το DNA και τις πυρηνικές πρωτεΐνες. Οι ιστόνες Η3 και Η4 έχουν μακριές ουρές που προεξέχουν από το νουκλεόσωμα, οι οποίες μπορούν να τροποποιηθούν ομοιοπολικά σε πολλά σημεία. Οι τροποποιήσεις της ουράς περιλαμβάνουν μεθυλίωση, ακετυλίωση, φωσφορυλίωση, ουβικιτινοποίηση, SUMOποίηση, κιτρουλλινοποίηση και ριβοζυλίωση ADP . Ο πυρήνας των ιστονών H2A και H2B μπορεί επίσης να τροποποιηθεί. Συνδυασμοί τροποποιήσεων, γνωστοί ως σημάδια ιστόνης, θεωρούνται ότι αποτελούν έναν κώδικα, τον λεγόμενο "κώδικα ιστόνης".[32][33] Οι τροποποιήσεις των ιστονών δρουν σε διάφορες βιολογικές διεργασίες όπως γονιδιακή ρύθμιση, επιδιόρθωση του DNA, συμπύκνωση χρωμοσωμάτων (μίτωση) και σπερματογένεση (μείωση).[34]
The common nomenclature of histone modifications is: Η κοινή ονοματολογία των τροποποιήσεων των ιστονών είναι:
- Το όνομα της ιστόνης (π.χ. H3)
- Η συντομογραφία ενός γράμματος αμινοξύ (π.χ. K για λυσίνη) και η θέση του αμινοξέος στην πρωτεΐνη
- Ο τύπος τροποποίησης (Me: μεθυλ, P: φωσφορικό, Ac: ακετύλιο, Ub: ουβικιτίνη)
- Ο αριθμός των τροποποιήσεων (μόνο το Me είναι γνωστό ότι συμβαίνει σε περισσότερα από ένα αντίγραφα ανά υπόλειμμα. 1, 2 ή 3 είναι μονο-, δι- ή τρι-μεθυλίωση)
Άρα το H3K4me1 υποδηλώνει τη μονομεθυλίωση του 4ου υπολείμματος (λυσίνης) από την αρχή (δηλαδή, το Ν-τελικό) της πρωτεΐνης Η3.
| Τύπος της τροποποίησης |
Ιστόνη | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H3K36 | H4K20 | H2BK5 | H2BK20 | |
| μόνο-μεθυλίωση | H3K4me1-ενεργοποίηση]][35] | ενεργοποίηση [36] | ενεργοποίηση [36] | ενεργοποίηση [36][37] | H4K20me- ενεργοποίηση [36] | ενεργοποίηση [36] | |||
| δι-μεθυλίωση | H3K9me2-καταστολή[38] | καταστολή[38] | H3K79me2-καταστολή[37] | ||||||
| τρι-μεθυλίωση | H3K4me3-ενεργοποίηση[39] | H3K9me3-καταστολή[36] | H3K27me3-καταστολή[36] | ενεργοποίηση,[37] repression[36] |
H3K36me3-ενεργοποίηση | καταστολή[38] | |||
| ακετυλίωση | ενεργοποίηση[40] | H3K9ac ενεργοποίηση [39] | ενεργοποίηση [39] | H3K27ac- ενεργοποίηση [41] | ενεργοποίηση | ||||
Τροποποίηση[Επεξεργασία | επεξεργασία κώδικα]

Έχει περιγραφεί ένας τεράστιος κατάλογος τροποποιήσεων ιστόνης, αλλά εξακολουθεί να λείπει η λειτουργική κατανόηση των περισσότερων. Συλλογικά, πιστεύεται ότι οι τροποποιήσεις ιστονών μπορεί να αποτελούν τη βάση ενός κώδικα ιστόνης, όπου οι συνδυασμοί τροποποιήσεων ιστονών έχουν συγκεκριμένες σημασίες. Ωστόσο, τα περισσότερα λειτουργικά δεδομένα αφορούν μεμονωμένες σημαντικές τροποποιήσεις ιστόνης που είναι βιοχημικά επιδεκτικές λεπτομερούς μελέτης.
Χημεία[Επεξεργασία | επεξεργασία κώδικα]
Μεθυλίωση λυσίνης[Επεξεργασία | επεξεργασία κώδικα]
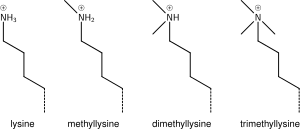
Η προσθήκη μιας, δύο ή πολλών μεθυλοομάδων στη λυσίνη έχει μικρή επίδραση στη χημεία των ιστονών. Η μεθυλίωση αφήνει ανέπαφο το φορτίο της λυσίνης και προσθέτει έναν ελάχιστο αριθμό ατόμων, έτσι οι στερεοχημικές αλληλεπιδράσεις είναι ως επί το πλείστον ανεπηρέαστες. Ωστόσο, πρωτεΐνες που περιέχουν περιοχές Tudor, χρωμο ή PHD, μεταξύ άλλων, μπορούν να αναγνωρίσουν τη μεθυλίωση της λυσίνης με εξαιρετική ευαισθησία και να διαφοροποιήσουν τη μονο, δι και τριμεθυλ λυσίνη, στο βαθμό που, για ορισμένες λυσίνες (π.χ.: H4K20) μονο, δι και τρι - μεθυλίωση φαίνεται να έχει διαφορετικές σημασίες. Εξαιτίας αυτού, η μεθυλίωση της λυσίνης τείνει να είναι ένα πολύ κατατοπιστικό σημάδι και κυριαρχεί στις γνωστές λειτουργίες τροποποίησης ιστονών.
Σεροτονυλίωση (serotonylation) γλουταμίνης[Επεξεργασία | επεξεργασία κώδικα]
Πρόσφατα αποδείχθηκε ότι η προσθήκη μιας ομάδας σεροτονίνης στη θέση 5 γλουταμίνης του Η3, συμβαίνει σε σεροτονινεργικά κύτταρα όπως οι νευρώνες. Αυτό είναι μέρος της διαφοροποίησης των σεροτονινεργικών κυττάρων. Αυτή η μετα-μεταφραστική τροποποίηση συμβαίνει σε συνδυασμό με την τροποποίηση της H3K4me3. Η σεροτονυλίωση ενισχύει τη δέσμευση του γενικού παράγοντα μεταγραφής TFIID στο πλαίσιο TATA.[42]
Μεθυλίωση αργινίνης[Επεξεργασία | επεξεργασία κώδικα]
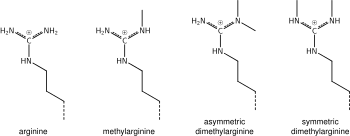
Αυτό που ειπώθηκε παραπάνω για τη χημεία της μεθυλίωσης της λυσίνης ισχύει επίσης για τη μεθυλίωση της αργινίνης και για ορισμένες πρωτεϊνικές περιοχές - π.χ. οι περιοχές Tudor – που μπορεί να είναι ειδικές για τη μεθυλαργινίνη αντί για τη μεθυλλυσίνη. Η αργινίνη είναι γνωστό ότι είναι μονο- ή δι-μεθυλιωμένη και η μεθυλίωση μπορεί να είναι συμμετρική ή ασύμμετρη, δυνητικά με διαφορετικές σημασίες.
Κιτρουλλινοποίηση της αργινίνης[Επεξεργασία | επεξεργασία κώδικα]
Τα ένζυμα που ονομάζονται απιμινάσες πεπτιδυλαργινίνης (peptidylarginine deiminases, PADs) υδρολύουν την ομάδα ιμίνης των αργινινών και προσαρτούν μια κετοομάδα, έτσι ώστε να υπάρχει ένα λιγότερο θετικό φορτίο στο υπόλειμμα του αμινοξέος. Αυτή η διαδικασία έχει εμπλακεί στην ενεργοποίηση της γονιδιακής έκφρασης καθιστώντας τις τροποποιημένες ιστόνες λιγότερο στενά συνδεδεμένες με το DNA και καθιστώντας έτσι τη χρωματίνη πιο προσιτή.[43] Τα PAD μπορούν επίσης να παράγουν το αντίθετο αποτέλεσμα αφαιρώντας ή αναστέλλοντας τη μονομεθυλίωση των υπολειμμάτων αργινίνης στις ιστόνες και έτσι ανταγωνίζονται τη θετική επίδραση που έχει η μεθυλίωση της αργινίνης στη μεταγραφική δραστηριότητα.[44]
Ακετυλίωση λυσίνης[Επεξεργασία | επεξεργασία κώδικα]
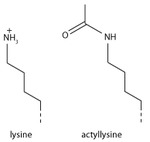
Η προσθήκη μιας ακετυλομάδας έχει σημαντική χημική επίδραση στη λυσίνη καθώς εξουδετερώνει το θετικό φορτίο. Αυτό μειώνει την ηλεκτροστατική έλξη μεταξύ της ιστόνης και της αρνητικά φορτισμένης ραχοκοκαλιάς του DNA, χαλαρώνοντας τη δομή της χρωματίνης. Οι πολύ ακετυλιωμένες ιστόνες σχηματίζουν μια πιο προσιτή χρωματίνη και τείνουν να συνδέονται με ενεργή μεταγραφή. Η ακετυλίωση της λυσίνης φαίνεται να είναι λιγότερο ακριβής ως προς τη σημασία από τη μεθυλίωση, καθώς οι ακετυλομεταφοράσες των ιστονών τείνουν να δρουν σε περισσότερες από μία λυσίνες. Προφανώς αυτό αντανακλά την ανάγκη αλλαγής πολλών λυσινών για να έχουν σημαντική επίδραση στη δομή της χρωματίνης. Η τροποποίηση περιλαμβάνει το την H3K27ac.
Φωσφορυλίωση σερίνης/θρεονίνης/τυροσίνης[Επεξεργασία | επεξεργασία κώδικα]
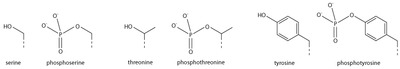
Η προσθήκη μιας αρνητικά φορτισμένης ομάδας φωσφορικών μπορεί να οδηγήσει σε σημαντικές αλλαγές στη δομή της πρωτεΐνης, οδηγώντας στον καλά χαρακτηρισμένο ρόλο της φωσφορυλίωσης στον έλεγχο της πρωτεϊνικής λειτουργίας. Δεν είναι σαφές ποιες δομικές επιπτώσεις έχει η φωσφορυλίωση των ιστονών, αλλά η φωσφορυλίωση τους έχει σαφείς λειτουργίες ως μετα-μεταφραστική τροποποίηση, και έχουν απεικονιστεί τομείς σύνδεσης όπως το BRCT.
Επιδράσεις στη μεταγραφή[Επεξεργασία | επεξεργασία κώδικα]
Οι περισσότερες καλά μελετημένες τροποποιήσεις ιστονών εμπλέκονται στον έλεγχο της μεταγραφής.
Ενεργά μεταγραμμένα γονίδια[Επεξεργασία | επεξεργασία κώδικα]
Δύο τροποποιήσεις ιστόνης συνδέονται ιδιαίτερα με την ενεργή μεταγραφή:
- Τριμεθυλίωση H3 λυσίνης 4 (H3K4me3)
- Αυτή η τριμεθυλίωση συμβαίνει στον υποκινητή των ενεργών γονιδίων [45][46][47] και εκτελείται από το σύμπλοκο COMPASS.[48][49][50]. Παρά τη διατήρηση αυτού του συμπλέγματος και της τροποποίησης της ιστόνης από ζυμομύκητες μέχρι τα θηλαστικά, δεν είναι απολύτως σαφές τι ρόλο παίζει αυτή η τροποποίηση. Ωστόσο, είναι ένας εξαιρετικός δείκτης ενεργών προαγωγών και το επίπεδο αυτής της τροποποίησης ιστόνης στον προαγωγέα ενός γονιδίου συσχετίζεται ευρέως με τη μεταγραφική δραστηριότητα του γονιδίου. Ο σχηματισμός αυτού του δείκτη συνδέεται με τη μεταγραφή με έναν μάλλον περίπλοκο τρόπο: νωρίς στην μεταγραφή ενός γονιδίου, η RNA πολυμεράση II υφίσταται αλλαγή από 'έναρξη' σε 'επιμήκυνση', που χαρακτηρίζεται από μια αλλαγή στις καταστάσεις φωσφορυλίωσης της RNA πολυμεράσης II στον τερματικό τομέα C (C terminal domain, CTD ). Το ίδιο ένζυμο που φωσφορυλιώνει το CTD φωσφορυλιώνει επίσης το σύμπλεγμα Rad6,[51][52] το οποίο με τη σειρά του προσθέτει έναν δείκτη ουβικιτίνης στο H2B K123 (K120 στα θηλαστικά).[53] Το H2BK123Ub εμφανίζεται σε όλες τις μεταγραφόμενες περιοχές, αλλά αυτό το σημάδι απαιτείται στην COMPASS για την τριμεθυλίωση του H3K4 στους προαγωγείς.[54][55]
- Τριμεθυλίωση H3 λυσίνης 36 (H3K36me3)
- Αυτή η τριμεθυλίωση συμβαίνει στο σώμα των ενεργών γονιδίων και εναποτίθεται από την μεθυλτρανσφεράσης Set 2.[56] Αυτή η πρωτεΐνη συνδέεται με την επιμήκυνση RNA πολυμεράσης II και η H3K36Me3 είναι ενδεικτική ενεργά μεταγραφόμενων γονιδίων.[57] Η H3K36Me3 αναγνωρίζεται από το σύμπλεγμα αποακετυλάσης ιστόνης Rpd3, το οποίο αφαιρεί τις τροποποιήσεις ακετυλίου από τις περιβάλλουσες ιστόνες, αυξάνοντας τη συμπίεση της χρωματίνης και καταστέλλοντας την ψευδή μεταγραφή.[58][59][60] Η αυξημένη συμπίεση της χρωματίνης αποτρέπει τους μεταγραφικούς παράγοντες από την πρόσβαση στο DNA και μειώνει την πιθανότητα έναρξης νέων μεταγραφικών συμβάντων εντός του σώματος του γονιδίου. Αυτή η διαδικασία επομένως βοηθά να διασφαλιστεί ότι η μεταγραφή δεν διακόπτεται.
Κατεσταλμένα γονίδια[Επεξεργασία | επεξεργασία κώδικα]
Τρεις τροποποιήσεις ιστόνης συνδέονται ιδιαίτερα με τα κατεσταλμένα γονίδια:
- Τριμεθυλίωση H3 λυσίνης 27 (H3K27me3)
- Αυτή η τροποποίηση ιστόνης εναποτίθεται από το πολυκομβικό σύμπλοκο PRC2.[61] Είναι ένας σαφής δείκτης γονιδιακής καταστολής,[62] και πιθανότατα δεσμεύεται από άλλες πρωτεΐνες για να ασκήσει κατασταλτική λειτουργία. Ένα άλλο πολυκομβικό σύμπλοκο, το PRC1, μπορεί να δεσμεύσει την H3K27me3[62] και προσθέτει την τροποποίηση ιστόνης H2AK119Ub που βοηθά στη συμπύκνωση της χρωματίνης.[63][64] Με βάση αυτά τα δεδομένα φαίνεται ότι η PRC1 στρατολογείται μέσω της δράσης της PRC2, ωστόσο, πρόσφατες μελέτες δείχνουν ότι η PRC1 στρατολογείται στις ίδιες τοποθεσίες απουσία PRC2.[65][66]
- Δι και τρι-μεθυλίωση της H3 λυσίνης 9 (H3K9me2/3)
- Η H3K9me2/3 είναι ένας καλά χαρακτηρισμένος δείκτης για την ετεροχρωματίνη και, ως εκ τούτου, συνδέεται ισχυρά με την γονιδιακή καταστολή. Ο σχηματισμός της ετεροχρωματίνης έχει μελετηθεί καλύτερα στη ζύμη Schizosaccharomyces pombe, όπου ξεκινά με τη στρατολόγηση του συμπλόκου μεταγραφικής αποσιώπισης που επάγεται από RNA (RNA-induced transcriptional silencing, RITS) σε δίκλωνα RNA που παράγονται από κεντρομεριδιακές επαναλήψεις.[67] Το RITS στρατολογεί την Clr4 μεθυλομεταφοράση ιστόνης που εναποθέτει την H3K9me2/3.[68] Αυτή η διαδικασία ονομάζεται μεθυλίωση ιστόνης. Η H3K9Me2/3 χρησιμεύει ως θέση δέσμευσης για τη στρατολόγηση του Swi6 (πρωτεΐνη ετεροχρωματίνης 1 ή (heterochromatin protein 1, HP1), ένας άλλος κλασικός δείκτης ετεροχρωματίνης), [69][70] που με τη σειρά του επιστρατεύει περαιτέρω κατασταλτικές δραστηριότητες συμπεριλαμβανομένων τροποποιητών ιστόνης όπως αποακετυλάσες ιστόνης και μεθυλομεταφοράσες ιστόνης.[71]
- Τριμεθυλίωση της H4 λυσίνης 20 (H4K20me3)
- Αυτή η τροποποίηση συνδέεται στενά με την ετεροχρωματίνη,[72][73] αν και η λειτουργική της σημασία παραμένει ασαφής. Αυτό το σημάδι τοποθετείται από τη μεθυλομεταφοράση Suv4-20h, η οποία τουλάχιστον εν μέρει στρατολογείται από την πρωτεΐνη ετεροχρωματίνης 1.[72]
Δισθενείς προαγωγείς[Επεξεργασία | επεξεργασία κώδικα]
Η ανάλυση των τροποποιήσεων ιστονών σε εμβρυϊκά βλαστοκύτταρα (και άλλα βλαστοκύτταρα) αποκάλυψε πολλούς προαγωγείς γονιδίων που φέρουν και τα δύο H3K4Me3 ]] όσο και H3K27Me3, με άλλα λόγια αυτοί οι προαγωγείς εμφανίζουν ενεργοποίηση και καταστολή σημαδιών ταυτόχρονα. Αυτός ο περίεργος συνδυασμός τροποποιήσεων σηματοδοτεί γονίδια που είναι έτοιμα για μεταγραφή. Δεν απαιτούνται στα βλαστοκύτταρα, αλλά απαιτούνται γρήγορα μετά τη διαφοροποίηση σε ορισμένες γενεαλογίες. Μόλις το κύτταρο αρχίσει να διαφοροποιείται, αυτοί οι δισθενείς προαγωγείς αναλύονται είτε σε ενεργές, είτε σε κατασταλτικές καταστάσεις ανάλογα με την επιλεγμένη γενεαλογία.[74]
Άλλες λειτουργίες[Επεξεργασία | επεξεργασία κώδικα]
Επιδιόρθωση βλάβης στο DNA[Επεξεργασία | επεξεργασία κώδικα]
Η επισήμανση σημείων βλάβης του DNA είναι μια σημαντική λειτουργία για τις τροποποιήσεις ιστονών. Χωρίς δείκτη επιδιόρθωσης, το DNA θα καταστρεφόταν από ζημιές που συσσωρεύονταν από πηγές όπως η υπεριώδης ακτινοβολία του ήλιου.
- Φωσφορυλίωση του H2AX στη σερίνη 139 (γH2AX)
- Η φωσφορυλιωμένη H2AX (επίσης γνωστή ως γάμμα H2AX) είναι δείκτης για διασπάσεις διπλού κλώνου DNA,[75] και αποτελεί μέρος της απόκρισης σε βλάβη του DNA.[30][76] Η H2AX φωσφορυλιώνεται νωρίς μετά την ανίχνευση θραύσης διπλού κλώνου DNA και σχηματίζει μια περιοχή που εκτείνεται σε πολλές κιλοβάσεις εκατέρωθεν της βλάβης.[75][77][78] Η γάμμα H2AX δρα ως δεσμευτική θέση για την πρωτεΐνη MDC1, η οποία με τη σειρά της στρατολογεί βασικές πρωτεΐνες επιδιόρθωσης του DNA [79] (Αυτό το περίπλοκο θέμα εξετάζεται καλά στο [80]) και ως εκ τούτου, η γάμμα H2AX αποτελεί ένα ζωτικό μέρος του μηχανισμού που διασφαλίζει τη σταθερότητα του γονιδιώματος.
- Ακετυλίωση H3 λυσίνης 56 (H3K56Ac)
- Η H3K56Acx απαιτείται για τη σταθερότητα του γονιδιώματος.[81][82], η H3K56 ακετυλιώνεται από το σύμπλοκο p300/Rtt109,[83][84][85] αλλά αποακετυλιώνεται ταχέως γύρω από θέσεις βλάβης του DNA. Απαιτείται επίσης ακετυλίωση της H3K56 για τη σταθεροποίηση στις διχάλες αντιγραφής που έχουν σταματήσει, αποτρέποντας την επικίνδυνη κατάρρευση στις διχάλες αναπαραγωγής.[86][87] Αν και γενικά τα θηλαστικά κάνουν πολύ μεγαλύτερη χρήση τροποποιήσεων ιστονών από τους μικροοργανισμούς, ένας σημαντικός ρόλος της H3K56Ac στην αντιγραφή του DNA υπάρχει μόνο στους μύκητες και αυτό έχει γίνει στόχος για την ανάπτυξη αντιβιοτικών.[88]
- Τριμεθυλίωση της H3 λυσίνης 36 (H3K36me3)
- Η H3K36me3 έχει την ικανότητα να στρατολογεί το σύμπλεγμα MSH2-MSH6 (hMutSα) της οδού επιδιόρθωσης ασυμφωνίας DNA.[89] Με συνέπεια, περιοχές του ανθρώπινου γονιδιώματος με υψηλά επίπεδα της H3K36me3 συσσωρεύουν λιγότερες σωματικές μεταλλάξεις λόγω της δραστηριότητας επιδιόρθωσης ασυμφωνίας.[90]
Συμπύκνωση χρωμοσωμάτων[Επεξεργασία | επεξεργασία κώδικα]
- Φωσφορυλίωση της Η3 στη σερίνη 10 (φωσφο-H3S10)
- Η μιτωτική κινάση aurora B φωσφορυλιώνει την ιστόνη Η3 στη σερίνη 10, προκαλώντας έναν καταρράκτη αλλαγών που μεσολαβούν στη συμπύκνωση των μιτωτικών χρωμοσωμάτων.[91][92] Επομένως, τα συμπυκνωμένα χρωμοσώματα χρωματίζονται πολύ έντονα για αυτό το σημάδι, αλλά η φωσφορυλίωση της H3S10 είναι επίσης παρούσα σε ορισμένες θέσεις χρωμοσωμάτων εκτός της μίτωσης, για παράδειγμα στην περικεντρική ετεροχρωματίνη των κυττάρων κατά τη διάρκεια του G2. Η φωσφορυλίωση της H3S10 έχει επίσης συνδεθεί με βλάβη του DNA που προκαλείται από το σχηματισμό βρόχου R-loop σε θέσεις υψηλής μεταγραφής. [93]
- Φωσφορυλίωση της H2B στη σερίνη 10/14 (φωσφο-H2BS10/14)
- Η φωσφορυλίωση του H2B στη σερίνη 10 (ζύμη) ή τη σερίνη 14 (θηλαστικά) συνδέεται επίσης με τη συμπύκνωση της χρωματίνης, αλλά για τον πολύ διαφορετικό σκοπό της μεσολάβησης της συμπύκνωσης χρωμοσωμάτων κατά την απόπτωση.[94][95] Αυτό το σημάδι δεν είναι απλώς ένας όψιμος παρατηρητής στην απόπτωση, καθώς οι μεταλλάξεις αυτού του υπολείμματος που φέρουν οι ζυμομύκητες είναι ανθεκτικές στον αποπτωτικό κυτταρικό θάνατο που προκαλείται από το υπεροξείδιο του υδρογόνου.
Εθισμός[Επεξεργασία | επεξεργασία κώδικα]
Οι επιγενετικές τροποποιήσεις των ουρών ιστονών σε συγκεκριμένες περιοχές του εγκεφάλου είναι κεντρικής σημασίας στους εθισμούς.[96][97][98] Μόλις εμφανιστούν συγκεκριμένες επιγενετικές αλλοιώσεις, φαίνεται να είναι μακροχρόνιες "μοριακές ουλές" που μπορεί να ευθύνονται για την εμμονή των εθισμών.[96]
Οι καπνιστές τσιγάρου (περίπου το 15% του πληθυσμού των ΗΠΑ) είναι συνήθως εθισμένοι στη νικοτίνη.[99] Μετά από 7 ημέρες θεραπείας με νικοτίνη ποντικών, η ακετυλίωση τόσο της ιστόνης Η3 όσο και της ιστόνης Η4 αυξήθηκε στον υποκινητή FosB στον επικλινή πυρήνα του εγκεφάλου, προκαλώντας αύξηση 61% στην έκφραση FosB.[100]. Αυτό θα αυξήσει επίσης την έκφραση της παραλλαγής ματίσματος δέλτα FosB. Στον επικλινή πυρήνα του εγκεφάλου, το δέλτα FosB λειτουργεί ως "παρατεταμένος μοριακός διακόπτης" και "κύρια πρωτεΐνη ελέγχου" στην ανάπτυξη ενός εθισμού.[101][102]
Περίπου το 7% του πληθυσμού των ΗΠΑ είναι εθισμένος στο αλκοόλ. Σε αρουραίους που εκτέθηκαν σε αλκοόλ για έως και 5 ημέρες, παρατηρήθηκε αύξηση της ακετυλίωσης της ιστόνης 3 λυσίνης 9 στον υποκινητή της προνοσισεπτίνης στο σύμπλεγμα αμυγδαλή του εγκεφάλου. Αυτή η ακετυλίωση είναι ένα σημάδι ενεργοποίησης για την προνοσισεπτίνη. Το σύστημα υποδοχέα οπιοειδών νοσισεπτίνης/νοσισεπτίνης εμπλέκεται στις ενισχυτικές ή ρυθμιστικές επιδράσεις του αλκοόλ.[103]
Ο εθισμός στη μεθαμφεταμίνη εμφανίζεται σε περίπου 0,2% του πληθυσμού των ΗΠΑ.[104] Η χρόνια χρήση μεθαμφεταμίνης προκαλεί μεθυλίωση της λυσίνης στη θέση 4 της ιστόνης 3 που βρίσκεται στους προαγωγείς του c-fos και στα γονίδια C-C υποδοχέας 2 χημειοκίνης (ccr2), ενεργοποιώντας αυτά τα γονίδια στον επικλινή πυρήνα (nucleus accumbens, NAc).[105] Το c-fos είναι γνωστό ότι είναι σημαντικό στον εθισμό.[106] Το γονίδιο ccr2 είναι επίσης σημαντικό στον εθισμό, καθώς η μεταλλακτική αδρανοποίηση αυτού του γονιδίου βλάπτει τον εθισμό.[105]
Σύνθεση[Επεξεργασία | επεξεργασία κώδικα]
Το πρώτο βήμα του διπλασιασμού της δομής της χρωματίνης είναι η σύνθεση πρωτεϊνών ιστόνης: H1, H2A, H2B, H3, H4. Αυτές οι πρωτεΐνες συντίθενται κατά τη φάση S του κυτταρικού κύκλου. Υπάρχουν διάφοροι μηχανισμοί που συμβάλλουν στην αύξηση της σύνθεσης ιστόνης.
Ζυμομύκητες[Επεξεργασία | επεξεργασία κώδικα]
Οι ζυμομύκητες φέρουν ένα ή δύο αντίγραφα κάθε γονιδίου ιστόνης, τα οποία δεν είναι ομαδοποιημένα αλλά μάλλον διάσπαρτα στα χρωμοσώματα. Η μεταγραφή του γονιδίου ιστόνης ελέγχεται από πολλές γονιδιακές ρυθμιστικές πρωτεΐνες όπως παράγοντες μεταγραφής που συνδέονται με περιοχές προαγωγέων ιστόνης. Σε εκκολαπτόμενους ζυμομύκητες, το υποψήφιο γονίδιο για την ενεργοποίηση της έκφρασης του γονιδίου ιστόνης είναι το SBF. Ο SBF είναι ένας μεταγραφικός παράγοντας που ενεργοποιείται στην όψιμη φάση G1, όταν διαχωρίζεται από τον καταστολέα του Whi5. Αυτό συμβαίνει όταν το Whi5 φωσφορυλιώνεται από το Cdc8 που είναι ένα G1/S Cdk.[107] Η καταστολή της έκφρασης του γονιδίου ιστόνης εκτός των φάσεων S εξαρτάται από τις πρωτεΐνες Hir που σχηματίζουν ανενεργή δομή χρωματίνης στον τόπο των γονιδίων ιστόνης, προκαλώντας τον αποκλεισμό των μεταγραφικών ενεργοποιητών.[108][109]
Μετάζωα[Επεξεργασία | επεξεργασία κώδικα]
Στα μετάζωα η αύξηση του ρυθμού σύνθεσης ιστόνης οφείλεται στην αύξηση της επεξεργασίας του προ-mRNA στην ώριμη μορφή του καθώς και στη μείωση της αποικοδόμησης του mRNA. Αυτό οδηγεί σε αύξηση του ενεργού mRNA για μετάφραση πρωτεϊνών ιστόνης. Ο μηχανισμός για την ενεργοποίηση του mRNA έχει βρεθεί ότι είναι η αφαίρεση ενός τμήματος του 3' άκρου του κλώνου του mRNA και εξαρτάται από τη συσχέτιση με την πρωτεΐνη δέσμευσης στελέχους-βρόχου (stem-loop binding protein ,SLBP).[110] Το SLBP σταθεροποιεί επίσης τα mRNA ιστόνης κατά τη φάση S αναστέλλοντας την αποικοδόμηση από τη νουκλεάση 3'hExo .[111] Τα επίπεδα SLBP ελέγχονται από πρωτεΐνες του κυτταρικού κύκλου, με αποτέλεσμα η SLBP να συσσωρεύεται καθώς τα κύτταρα εισέρχονται στη φάση S και αποικοδομούνται καθώς τα κύτταρα φεύγουν από τη φάση S. Τα SLBP επισημαίνονται για αποικοδόμηση με φωσφορυλίωση σε δύο υπολείμματα θρεονίνης από κινάσες εξαρτώμενες από κυκλίνη, πιθανώς την κυκλίνη A/cdk2, στο τέλος της φάσης S.[112] Τα μετάζωα έχουν επίσης πολλά αντίγραφα γονιδίων ιστονών συγκεντρωμένα σε χρωμοσώματα που εντοπίζονται σε δομές που ονομάζονται σώματα Cajal όπως προσδιορίζονται από ανάλυση σύλληψης χρωμοσωμικής διαμόρφωσης σε όλο το γονιδίωμα (4C-Seq).[113]
Σχέση μεταξύ ελέγχου κυτταρικού κύκλου και σύνθεσης[Επεξεργασία | επεξεργασία κώδικα]
Η πυρηνική πρωτεΐνη Αταξία-Τηλαγγειεκτασία (Nuclear protein Ataxia-Telangiectasia, NPAT), επίσης γνωστή ως πυρηνικός συνενεργοποιητής πρωτεϊνών της μεταγραφής ιστόνης, είναι ένας μεταγραφικός παράγοντας που ενεργοποιεί τη μεταγραφή του γονιδίου ιστόνης στα χρωμοσώματα 1 και 6 των ανθρώπινων κυττάρων. H NPAT είναι επίσης ένα υπόστρωμα της κυκλίνης E-Cdk2, το οποίο απαιτείται για τη μετάβαση μεταξύ της φάσης G1 και της φάσης S. Το NPAT ενεργοποιεί την έκφραση του γονιδίου ιστόνης μόνο αφού έχει φωσφορυλιωθεί από την G1/S-Cdk κυκλίνη E-Cdk2 στην πρώιμη φάση S.[114] Αυτό δείχνει μια σημαντική ρυθμιστική σύνδεση μεταξύ του ελέγχου του κυτταρικού κύκλου και της σύνθεσης ιστόνης.
Ιστορικό[Επεξεργασία | επεξεργασία κώδικα]
Οι ιστόνες ανακαλύφθηκαν το 1884 από τον Άλμπρεχτ Κόσελ.[115] Η λέξη "ιστόνη" χρονολογείται από τα τέλη του 19ου αιώνα και προέρχεται από τη γερμανική λέξη "Histon", μια λέξη αβέβαιης προέλευσης, ίσως από την αρχαία ελληνική ἵστημι (hístēmi, "κάνω στάση"). ή ἱστός.
Στις αρχές της δεκαετίας του 1960, πριν γίνουν γνωστοί οι τύποι των ιστονών και προτού οι ιστόνες ήταν γνωστό ότι διατηρούνται σε μεγάλο βαθμό σε ταξινομικά διαφορετικούς οργανισμούς, ο James F. Bonner και οι συνεργάτες του ξεκίνησαν μια μελέτη αυτών των πρωτεϊνών που ήταν γνωστό ότι συνδέονται στενά με το DNA στον πυρήνα ανώτερων οργανισμών.[116] Ο Bonner και ο μεταδιδακτορικός συνεργάτης του Ru Chih C. Huang έδειξαν ότι η απομονωμένη χρωματίνη δεν θα υποστήριζε τη μεταγραφή RNA στον δοκιμαστικό σωλήνα, αλλά εάν οι ιστόνες εξάγονταν από τη χρωματίνη, το RNA θα μπορούσε να μεταγραφεί από το υπόλοιπο DNA.[117] Η εργασία τους έγινε κλασική αναφορά.[118] Ο Paul T'so και ο James Bonner είχαν συγκαλέσει ένα Παγκόσμιο Συνέδριο για τη Χημεία και τη Βιολογία των Ιστόνης το 1964, στο οποίο κατέστη σαφές ότι δεν υπήρχε συναίνεση για τον αριθμό των ειδών ιστόνης και ότι κανείς δεν ήξερε πώς θα συγκρίνονται όταν απομονωθεί από διαφορετικούς οργανισμούς.[119][116] Ο Bonner και οι συνεργάτες του ανέπτυξαν στη συνέχεια μεθόδους για τον διαχωρισμό κάθε τύπου ιστόνης, καθάρισαν μεμονωμένες ιστόνες, συνέκριναν συνθέσεις αμινοξέων στην ίδια ιστόνη από διαφορετικούς οργανισμούς και συνέκριναν αλληλουχίες αμινοξέων της ίδιας ιστόνης από διαφορετικούς οργανισμούς σε συνεργασία με τον Emil Smith από το UCLA.[120] Για παράδειγμα, βρήκαν ότι η αλληλουχία Histone IV διατηρείται σε μεγάλο βαθμό μεταξύ του αρακά και του θύμου μόσχου.[120] Ωστόσο, η εργασία τους για τα βιοχημικά χαρακτηριστικά μεμονωμένων ιστονών δεν αποκάλυψε πώς οι ιστόνες αλληλεπιδρούσαν μεταξύ τους ή με το DNA στο οποίο ήταν στενά συνδεδεμένες.[119]
Επίσης στη δεκαετία του 1960, ο Vincent Allfrey και ο Alfred Mirsky είχαν προτείνει, με βάση τις αναλύσεις τους για τις ιστόνες, ότι η ακετυλίωση και η μεθυλίωση των ιστονών θα μπορούσαν να παρέχουν έναν μεταγραφικό μηχανισμό ελέγχου, αλλά δεν είχαν διαθέσιμο το είδος της λεπτομερούς ανάλυσης που οι μετέπειτα ερευνητές ήταν σε θέση να δείξουν πώς μια τέτοια ρύθμιση θα μπορούσε να είναι ειδική για το γονίδιο.[121] Μέχρι τις αρχές της δεκαετίας του 1990, οι ιστόνες απορρίπτονταν από τους περισσότερους ως αδρανές υλικό συσκευασίας για το ευκαρυωτικό πυρηνικό DNA, μια άποψη που βασίζεται εν μέρει στα μοντέλα του Mark Ptashne και άλλων, οι οποίοι πίστευαν ότι η μεταγραφή ενεργοποιήθηκε από αλληλεπιδράσεις πρωτεΐνης-DNA και πρωτεϊνών - πρωτεϊνών σε γυμνά εκμαγεία DNA σε μεγάλο βαθμό, όπως συμβαίνει στα βακτήρια.
Κατά τη δεκαετία του 1980, ο Yahli Lorch και ο Roger Kornberg[122] έδειξαν ότι ένα νουκλεόσωμα σε έναν πυρήνα προαγωγέα εμποδίζει την έναρξη της μεταγραφής in vitro, και ο Michael Grunstein[123] έδειξε ότι οι ιστόνες καταστέλλουν τη μεταγραφή in vivo, οδηγώντας στην ιδέα του νουκλεοσώματος ως γενικού γονιδιακού καταστολέα. Η ανακούφιση από την καταστολή πιστεύεται ότι περιλαμβάνει τόσο την τροποποίηση ιστόνης όσο και τη δράση των συμπλεγμάτων αναδιαμόρφωσης της χρωματίνης. Οι Vincent Allfrey και Alfred Mirsky είχαν προτείνει νωρίτερα έναν ρόλο τροποποίησης ιστόνης στη μεταγραφική ενεργοποίηση,[124] να θεωρείται ως μοριακή εκδήλωση της επιγενετικής. Ο Michael Grunstein [125] και ο Ντέιβιντ Άλις [126] βρήκαν υποστήριξη για αυτήν την πρόταση, όσον αφορά τη σημασία της ακετυλίωσης ιστόνης για τη μεταγραφή σε ζυμομύκητες και τη δραστηριότητα του μεταγραφικού ενεργοποιητή Gcn5 ως ακετυλομεταφοράσης ιστόνης.
Η ανακάλυψη της ιστόνης H5 φαίνεται να χρονολογείται από τη δεκαετία του 1970,[127] και τώρα θεωρείται ισομορφή της ιστόνης Η1.[2][4][5][6]
Παραπομπές[Επεξεργασία | επεξεργασία κώδικα]
- ↑ Youngson RM (2006). Collins Dictionary of Human Biology. Glasgow: HarperCollins. ISBN 978-0-00-722134-9.
- ↑ 2,0 2,1 2,2 2,3 Cox M, Nelson DR, Lehninger AL (2005). Lehninger Principles of Biochemistry
 . San Francisco: W.H. Freeman. ISBN 978-0-7167-4339-2.
. San Francisco: W.H. Freeman. ISBN 978-0-7167-4339-2.
- ↑ «Histone H2A variants H2AX and H2AZ». Current Opinion in Genetics & Development 12 (2): 162–9. April 2002. doi:. PMID 11893489. https://www.sciencedirect.com/science/article/abs/pii/S0959437X02002824.
- ↑ 4,0 4,1 «Histone Variants Database 2.0». National Center for Biotechnology Information. Ανακτήθηκε στις 13 Ιανουαρίου 2017.
- ↑ 5,0 5,1 «Recognition and classification of histones using support vector machine». Journal of Computational Biology 13 (1): 102–12. 2006. doi:. PMID 16472024. Αρχειοθετήθηκε από το πρωτότυπο στις 2020-11-28. https://web.archive.org/web/20201128030045/http://eprints.ucm.es/9328/1/31.Reche_etal_MI_2006.pdf. Ανακτήθηκε στις 2024-01-06.
- ↑ 6,0 6,1 Hartl DL, Freifelder D, Snyder LA (1988). Basic Genetics
 . Boston: Jones and Bartlett Publishers. ISBN 978-0-86720-090-4.
. Boston: Jones and Bartlett Publishers. ISBN 978-0-86720-090-4.
- ↑ «Histone structure and nucleosome stability». Expert Review of Proteomics 2 (5): 719–29. October 2005. doi:. PMID 16209651.
- ↑ 8,0 8,1 «Crystal structure of the nucleosome core particle at 2.8 A resolution». Nature 389 (6648): 251–60. September 1997. doi:. PMID 9305837. Bibcode: 1997Natur.389..251L. https://www.nature.com/articles/38444.
- ↑ Farkas D (1996). DNA simplified: the hitchhiker's guide to DNA. Washington, D.C: AACC Press. ISBN 978-0-915274-84-0.
- ↑ 10,0 10,1 Seal, Ruth L.; Denny, Paul; Bruford, Elspeth A.; Gribkova, Anna K.; Landsman, David; Marzluff, William F.; McAndrews, Monica; Panchenko, Anna R. και άλλοι. (October 2022). «A standardized nomenclature for mammalian histone genes». Epigenetics & Chromatin 15 (1): 34. doi:. ISSN 1756-8935. PMID 36180920.
- ↑ «Histone H3.3 maintains genome integrity during mammalian development». Genes & Development 29 (13): 1377–92. July 2015. doi:. PMID 26159997.
- ↑ Draizen, Eli J.; Shaytan, Alexey K.; Mariño-Ramírez, Leonardo; Talbert, Paul B.; Landsman, David; Panchenko, Anna R. (2016). «HistoneDB 2.0: a histone database with variants--an integrated resource to explore histones and their variants». Database: The Journal of Biological Databases and Curation 2016: baw014. doi:. ISSN 1758-0463. PMID 26989147.
- ↑ El Kennani, Sara; Adrait, Annie; Shaytan, Alexey K.; Khochbin, Saadi; Bruley, Christophe; Panchenko, Anna R.; Landsman, David; Pflieger, Delphine και άλλοι. (2017). «MS_HistoneDB, a manually curated resource for proteomic analysis of human and mouse histones». Epigenetics & Chromatin 10: 2. doi:. ISSN 1756-8935. PMID 28096900.
- ↑ Marashi, F.; Prokopp, K.; Stein, J.; Stein, G. (April 1984). «Evidence for a human histone gene cluster containing H2B and H2A pseudogenes». Proceedings of the National Academy of Sciences of the United States of America 81 (7): 1936–1940. doi:. ISSN 0027-8424. PMID 6326092.
- ↑ Kardalinou, E.; Eick, S.; Albig, W.; Doenecke, D. (August 1993). «Association of a human H1 histone gene with an H2A pseudogene and genes encoding H2B.1 and H3.1 histones». Journal of Cellular Biochemistry 52 (4): 375–383. doi:. ISSN 0730-2312. PMID 8227173. https://pubmed.ncbi.nlm.nih.gov/8227173/.
- ↑ 16,0 16,1 «On the origin of the histone fold». BMC Structural Biology 7: 17. March 2007. doi:. PMID 17391511.
- ↑ «Structure of histone-based chromatin in Archaea». Science 357 (6351): 609–612. August 2017. doi:. PMID 28798133. Bibcode: 2017Sci...357..609M.
- ↑ 18,0 18,1 18,2 «Structure and function of archaeal histones». PLOS Genetics 14 (9): e1007582. September 2018. doi:. PMID 30212449. Bibcode: 2018BpJ...114..446H.
- ↑ «Long distance PELDOR measurements on the histone core particle». Journal of the American Chemical Society 131 (4): 1348–9. February 2009. doi:. PMID 19138067.
- ↑ «The tale beyond the tail: histone core domain modifications and the regulation of chromatin structure». Nucleic Acids Research 34 (9): 2653–62. 19 May 2006. doi:. PMID 16714444.
- ↑ «Scratching the (lateral) surface of chromatin regulation by histone modifications». Nature Structural & Molecular Biology 20 (6): 657–61. June 2013. doi:. PMID 23739170. https://www.nature.com/articles/nsmb.2581.
- ↑ Allison LA (2012). Fundamental Molecular Biology (Second έκδοση). United States of America: John Wiley & Sons. σελ. 102. ISBN 9781118059814.
- ↑ «Those amazing dinoflagellate chromosomes». Cell Research 13 (4): 215–7. August 2003. doi:. PMID 12974611.
- ↑ «Chromatin: packaging without nucleosomes». Current Biology 22 (24): R1040-3. December 2012. doi:. PMID 23257187.
- ↑ «Origin of H1 linker histones». FASEB Journal 15 (1): 34–42. January 2001. doi:. PMID 11149891. https://faseb.onlinelibrary.wiley.com/doi/epdf/10.1096/fj.00-0237rev.
- ↑ «The origin of chromosomal histones in a 30S ribosomal protein». Gene 726: 144155. February 2020. doi:. PMID 31629821. https://www.sciencedirect.com/science/article/abs/pii/S0378111919308145.
- ↑ «Nuclear and chromatin composition of mammalian gametes and early embryos». Biochemistry and Cell Biology 70 (10–11): 856–66. 1992. doi:. PMID 1297351. https://cdnsciencepub.com/doi/10.1139/o92-134.
- ↑ «Variant histone H2A.Z is globally localized to the promoters of inactive yeast genes and regulates nucleosome positioning». PLOS Biology 3 (12): e384. December 2005. doi:. PMID 16248679.
- ↑ «Precise deposition of histone H2A.Z in chromatin for genome expression and maintenance». Biochim Biophys Acta 1819 (3–4): 290–302. October 2011. doi:. PMID 22027408. https://www.sciencedirect.com/science/article/abs/pii/S1874939911001805.
- ↑ 30,0 30,1 «A critical role for histone H2AX in recruitment of repair factors to nuclear foci after DNA damage». Current Biology 10 (15): 886–95. 2000. doi:. PMID 10959836.
- ↑ «The histone variant H3.3 marks active chromatin by replication-independent nucleosome assembly». Molecular Cell 9 (6): 1191–200. June 2002. doi:. PMID 12086617.
- ↑ «The language of covalent histone modifications». Nature 403 (6765): 41–5. January 2000. doi:. PMID 10638745. Bibcode: 2000Natur.403...41S. https://ui.adsabs.harvard.edu/abs/2000Natur.403...41S/abstract.
- ↑ «Translating the histone code». Science 293 (5532): 1074–80. August 2001. doi:. PMID 11498575. http://www.gs.washington.edu/academics/courses/braun/55104/readings/jenuwein.pdf.
- ↑ «Immunohistochemical Analysis of Histone H3 Modifications in Germ Cells during Mouse Spermatogenesis». Acta Histochemica et Cytochemica 44 (4): 183–90. August 2011. doi:. PMID 21927517.
- ↑ «Histone H3K4 demethylases are essential in development and differentiation». Biochemistry and Cell Biology 85 (4): 435–43. August 2007. doi:. PMID 17713579. https://cdnsciencepub.com/doi/10.1139/O07-057.
- ↑ 36,0 36,1 36,2 36,3 36,4 36,5 36,6 36,7 «High-resolution profiling of histone methylations in the human genome». Cell 129 (4): 823–37. May 2007. doi:. PMID 17512414.
- ↑ 37,0 37,1 37,2 «DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells». Molecular and Cellular Biology 28 (8): 2825–39. April 2008. doi:. PMID 18285465. PMC 2293113. https://archive.org/details/sim_molecular-and-cellular-biology_2008-04_28_8/page/2825.
- ↑ 38,0 38,1 38,2 «Determination of enriched histone modifications in non-genic portions of the human genome». BMC Genomics 10: 143. March 2009. doi:. PMID 19335899.
- ↑ 39,0 39,1 39,2 «The landscape of histone modifications across 1% of the human genome in five human cell lines». Genome Research 17 (6): 691–707. June 2007. doi:. PMID 17567990.
- ↑ «H3 lysine 4 is acetylated at active gene promoters and is regulated by H3 lysine 4 methylation». PLOS Genetics 7 (3): e1001354. March 2011. doi:. PMID 21483810.
- ↑ «Histone H3K27ac separates active from poised enhancers and predicts developmental state». Proceedings of the National Academy of Sciences of the United States of America 107 (50): 21931–6. December 2010. doi:. PMID 21106759.
- ↑ «Histone serotonylation is a permissive modification that enhances TFIID binding to H3K4me3». Nature 567 (7749): 535–539. March 2019. doi:. PMID 30867594. Bibcode: 2019Natur.567..535F.
- ↑ «Citrullination regulates pluripotency and histone H1 binding to chromatin». Nature 507 (7490): 104–8. March 2014. doi:. PMID 24463520. PMC 4843970. Bibcode: 2014Natur.507..104C. https://www.repository.cam.ac.uk/handle/1810/254537.
- ↑ «Histone deimination antagonizes arginine methylation». Cell 118 (5): 545–53. September 2004. doi:. PMID 15339660.
- ↑ «The Paf1 complex is required for histone H3 methylation by COMPASS and Dot1p: linking transcriptional elongation to histone methylation». Molecular Cell 11 (3): 721–9. March 2003. doi:. PMID 12667454.
- ↑ «Targeted recruitment of Set1 histone methylase by elongating Pol II provides a localized mark and memory of recent transcriptional activity». Molecular Cell 11 (3): 709–19. March 2003. doi:. PMID 12667453.
- ↑ «Genomic maps and comparative analysis of histone modifications in human and mouse». Cell 120 (2): 169–81. January 2005. doi:. PMID 15680324.
- ↑ «COMPASS, a histone H3 (Lysine 4) methyltransferase required for telomeric silencing of gene expression». The Journal of Biological Chemistry 277 (13): 10753–5. March 2002. doi:. PMID 11805083.
- ↑ «The Saccharomyces cerevisiae Set1 complex includes an Ash2 homologue and methylates histone 3 lysine 4». The EMBO Journal 20 (24): 7137–48. December 2001. doi:. PMID 11742990.
- ↑ «A trithorax-group complex purified from Saccharomyces cerevisiae is required for methylation of histone H3». Proceedings of the National Academy of Sciences of the United States of America 99 (1): 90–4. January 2002. doi:. PMID 11752412. Bibcode: 2002PNAS...99...90N.
- ↑ «The Bur1/Bur2 complex is required for histone H2B monoubiquitination by Rad6/Bre1 and histone methylation by COMPASS». Molecular Cell 20 (4): 589–99. November 2005. doi:. PMID 16307922.
- ↑ «Regulation of the ubiquitin-conjugating enzyme hHR6A by CDK-mediated phosphorylation». The EMBO Journal 21 (8): 2009–18. April 2002. doi:. PMID 11953320.
- ↑ «Rad6-dependent ubiquitination of histone H2B in yeast». Science 287 (5452): 501–4. January 2000. doi:. PMID 10642555. Bibcode: 2000Sci...287..501R. https://www.science.org/doi/10.1126/science.287.5452.501.
- ↑ «Ubiquitination of histone H2B regulates H3 methylation and gene silencing in yeast». Nature 418 (6893): 104–8. July 2002. doi:. PMID 12077605. Bibcode: 2002Natur.418..104S. https://www.nature.com/articles/nature00883.
- ↑ «Methylation of histone H3 by COMPASS requires ubiquitination of histone H2B by Rad6». The Journal of Biological Chemistry 277 (32): 28368–71. August 2002. doi:. PMID 12070136.
- ↑ «Set2 is a nucleosomal histone H3-selective methyltransferase that mediates transcriptional repression». Molecular and Cellular Biology 22 (5): 1298–306. March 2002. doi:. PMID 11839797. PMC 134702. https://archive.org/details/sim_molecular-and-cellular-biology_2002-03_22_5/page/1298.
- ↑ «Association of the histone methyltransferase Set2 with RNA polymerase II plays a role in transcription elongation». The Journal of Biological Chemistry 277 (51): 49383–8. December 2002. doi:. PMID 12381723.
- ↑ «Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription». Cell 123 (4): 581–92. November 2005. doi:. PMID 16286007.
- ↑ «Cotranscriptional set2 methylation of histone H3 lysine 36 recruits a repressive Rpd3 complex». Cell 123 (4): 593–605. November 2005. doi:. PMID 16286008.
- ↑ «Eaf3 chromodomain interaction with methylated H3-K36 links histone deacetylation to Pol II elongation». Molecular Cell 20 (6): 971–8. December 2005. doi:. PMID 16364921.
- ↑ «Histone methyltransferase activity associated with a human multiprotein complex containing the Enhancer of Zeste protein». Genes & Development 16 (22): 2893–905. November 2002. doi:. PMID 12435631.
- ↑ 62,0 62,1 «Role of histone H3 lysine 27 methylation in Polycomb-group silencing». Science 298 (5595): 1039–43. November 2002. doi:. PMID 12351676. Bibcode: 2002Sci...298.1039C. https://www.science.org/doi/10.1126/science.1076997.
- ↑ «Polycomb group proteins Ring1A/B link ubiquitylation of histone H2A to heritable gene silencing and X inactivation». Developmental Cell 7 (5): 663–76. November 2004. doi:. PMID 15525528.
- ↑ «Role of histone H2A ubiquitination in Polycomb silencing». Nature 431 (7010): 873–8. October 2004. doi:. PMID 15386022. Bibcode: 2004Natur.431..873W.
- ↑ «RYBP-PRC1 complexes mediate H2A ubiquitylation at polycomb target sites independently of PRC2 and H3K27me3». Cell 148 (4): 664–78. February 2012. doi:. PMID 22325148.
- ↑ «PCGF homologs, CBX proteins, and RYBP define functionally distinct PRC1 family complexes». Molecular Cell 45 (3): 344–56. February 2012. doi:. PMID 22325352.
- ↑ «RNAi-mediated targeting of heterochromatin by the RITS complex». Science 303 (5658): 672–6. January 2004. doi:. PMID 14704433. Bibcode: 2004Sci...303..672V.
- ↑ «Regulation of chromatin structure by site-specific histone H3 methyltransferases». Nature 406 (6796): 593–9. August 2000. doi:. PMID 10949293. Bibcode: 2000Natur.406..593R. https://www.nature.com/articles/35020506.
- ↑ «Selective recognition of methylated lysine 9 on histone H3 by the HP1 chromo domain». Nature 410 (6824): 120–4. March 2001. doi:. PMID 11242054. Bibcode: 2001Natur.410..120B. https://www.nature.com/articles/35065138.
- ↑ «Methylation of histone H3 lysine 9 creates a binding site for HP1 proteins». Nature 410 (6824): 116–20. March 2001. doi:. PMID 11242053. Bibcode: 2001Natur.410..116L. https://www.nature.com/articles/35065132.
- ↑ «Binding of DNA-bending non-histone proteins destabilizes regular 30-nm chromatin structure». PLOS Computational Biology 13 (1): e1005365. January 2017. doi:. PMID 28135276. Bibcode: 2017PLSCB..13E5365B.
- ↑ 72,0 72,1 «A silencing pathway to induce H3-K9 and H4-K20 trimethylation at constitutive heterochromatin». Genes & Development 18 (11): 1251–62. June 2004. doi:. PMID 15145825.
- ↑ «Heterochromatin and tri-methylated lysine 20 of histone H4 in animals». Journal of Cell Science 117 (Pt 12): 2491–501. May 2004. doi:. PMID 15128874.
- ↑ «A bivalent chromatin structure marks key developmental genes in embryonic stem cells». Cell 125 (2): 315–26. April 2006. doi:. PMID 16630819.
- ↑ 75,0 75,1 «DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139». The Journal of Biological Chemistry 273 (10): 5858–68. March 1998. doi:. PMID 9488723.
- ↑ «Genomic instability in mice lacking histone H2AX». Science 296 (5569): 922–7. May 2002. doi:. PMID 11934988. Bibcode: 2002Sci...296..922C.
- ↑ «Distribution and dynamics of chromatin modification induced by a defined DNA double-strand break». Current Biology 14 (19): 1703–11. October 2004. doi:. PMID 15458641.
- ↑ «Megabase chromatin domains involved in DNA double-strand breaks in vivo». The Journal of Cell Biology 146 (5): 905–16. September 1999. doi:. PMID 10477747.
- ↑ «MDC1 is a mediator of the mammalian DNA damage checkpoint». Nature 421 (6926): 961–6. February 2003. doi:. PMID 12607005. Bibcode: 2003Natur.421..961S. https://www.nature.com/articles/nature01446.
- ↑ «Assembly and function of DNA double-strand break repair foci in mammalian cells». DNA Repair 9 (12): 1219–28. December 2010. doi:. PMID 21035408. https://www.sciencedirect.com/science/article/abs/pii/S1568786410003150.
- ↑ «Characterization of lysine 56 of histone H3 as an acetylation site in Saccharomyces cerevisiae». The Journal of Biological Chemistry 280 (28): 25949–52. July 2005. doi:. PMID 15888442.
- ↑ «A role for cell-cycle-regulated histone H3 lysine 56 acetylation in the DNA damage response». Nature 436 (7048): 294–8. July 2005. doi:. PMID 16015338. Bibcode: 2005Natur.436..294M. https://www.nature.com/articles/nature03714.
- ↑ «Yeast Rtt109 promotes genome stability by acetylating histone H3 on lysine 56». Science 315 (5812): 649–52. February 2007. doi:. PMID 17272722. Bibcode: 2007Sci...315..649D.
- ↑ «Rtt109 acetylates histone H3 lysine 56 and functions in DNA replication». Science 315 (5812): 653–5. February 2007. doi:. PMID 17272723. Bibcode: 2007Sci...315..653H. https://www.science.org/doi/10.1126/science.1133234.
- ↑ «CBP/p300-mediated acetylation of histone H3 on lysine 56». Nature 459 (7243): 113–7. May 2009. doi:. PMID 19270680. Bibcode: 2009Natur.459..113D.
- ↑ «Acetylation of lysine 56 of histone H3 catalyzed by RTT109 and regulated by ASF1 is required for replisome integrity». The Journal of Biological Chemistry 282 (39): 28587–96. September 2007. doi:. PMID 17690098.
- ↑ «Histone H3 lysine 56 acetylation and the response to DNA replication fork damage». Molecular and Cellular Biology 32 (1): 154–72. January 2012. doi:. PMID 22025679.
- ↑ «Modulation of histone H3 lysine 56 acetylation as an antifungal therapeutic strategy». Nature Medicine 16 (7): 774–80. July 2010. doi:. PMID 20601951.
- ↑ «The histone mark H3K36me3 regulates human DNA mismatch repair through its interaction with MutSα». Cell 153 (3): 590–600. April 2013. doi:. PMID 23622243.
- ↑ «Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes». Cell 170 (3): 534–547.e23. July 2017. doi:. PMID 28753428.
- ↑ «A cascade of histone modifications induces chromatin condensation in mitosis». Science 343 (6166): 77–80. January 2014. doi:. PMID 24385627. Bibcode: 2014Sci...343...77W. https://www.science.org/doi/10.1126/science.1244508.
- ↑ «Regulation of chromatin structure by histone H3S10 phosphorylation». Chromosome Research 14 (4): 393–404. 2006. doi:. PMID 16821135. https://link.springer.com/article/10.1007/s10577-006-1063-4.
- ↑ «R loops are linked to histone H3 S10 phosphorylation and chromatin condensation». Molecular Cell 52 (4): 583–90. November 2013. doi:. PMID 24211264.
- ↑ «Apoptotic phosphorylation of histone H2B is mediated by mammalian sterile twenty kinase». Cell 113 (4): 507–17. May 2003. doi:. PMID 12757711.
- ↑ «Sterile 20 kinase phosphorylates histone H2B at serine 10 during hydrogen peroxide-induced apoptosis in S. cerevisiae». Cell 120 (1): 25–36. January 2005. doi:. PMID 15652479.
- ↑ 96,0 96,1 «Transcriptional and epigenetic mechanisms of addiction». Nature Reviews. Neuroscience 12 (11): 623–37. October 2011. doi:. PMID 21989194.
- ↑ Hitchcock LN, Lattal KM (2014). «Histone-mediated epigenetics in addiction». Epigenetics and Neuroplasticity—Evidence and Debate. Prog Mol Biol Transl Sci. Progress in Molecular Biology and Translational Science. 128. Academic Press. σελίδες 51–87. doi:10.1016/B978-0-12-800977-2.00003-6. ISBN 9780128009772. PMC 5914502
 . PMID 25410541.
. PMID 25410541.
- ↑ «Epigenetic regulation in substance use disorders». Current Psychiatry Reports 12 (2): 145–53. April 2010. doi:. PMID 20425300.
- ↑ «Is nicotine addictive?».
- ↑ «Molecular mechanism for a gateway drug: epigenetic changes initiated by nicotine prime gene expression by cocaine». Science Translational Medicine 3 (107): 107ra109. November 2011. doi:. PMID 22049069.
- ↑ «Molecular neurobiology of addiction: what's all the (Δ)FosB about?». The American Journal of Drug and Alcohol Abuse 40 (6): 428–37. November 2014. doi:. PMID 25083822. https://www.tandfonline.com/doi/abs/10.3109/00952990.2014.933840?journalCode=iada20.
- ↑ «DeltaFosB: a sustained molecular switch for addiction». Proceedings of the National Academy of Sciences of the United States of America 98 (20): 11042–6. September 2001. doi:. PMID 11572966. Bibcode: 2001PNAS...9811042N.
- ↑ «Ethanol induces epigenetic modulation of prodynorphin and pronociceptin gene expression in the rat amygdala complex». Journal of Molecular Neuroscience 49 (2): 312–9. February 2013. doi:. PMID 22684622. https://link.springer.com/article/10.1007/s12031-012-9829-y.
- ↑ «What is the scope of methamphetamine abuse in the United States?».
- ↑ 105,0 105,1 «Epigenetic landscape of amphetamine and methamphetamine addiction in rodents». Epigenetics 10 (7): 574–80. 2015. doi:. PMID 26023847.
- ↑ «Using c-fos to study neuronal ensembles in corticostriatal circuitry of addiction». Brain Research 1628 (Pt A): 157–73. December 2015. doi:. PMID 25446457.
- ↑ «Cln3 activates G1-specific transcription via phosphorylation of the SBF bound repressor Whi5». Cell 117 (7): 887–98. June 2004. doi:. PMID 15210110.
- ↑ «Identification of a new set of cell cycle-regulatory genes that regulate S-phase transcription of histone genes in Saccharomyces cerevisiae». Molecular and Cellular Biology 12 (11): 5249–59. November 1992. doi:. PMID 1406694. PMC 360458. https://archive.org/details/sim_molecular-and-cellular-biology_1992-11_12_11/page/5249.
- ↑ «A role for transcriptional repressors in targeting the yeast Swi/Snf complex». Molecular Cell 4 (1): 75–83. July 1999. doi:. PMID 10445029.
- ↑ «A novel zinc finger protein is associated with U7 snRNP and interacts with the stem-loop binding protein in the histone pre-mRNP to stimulate 3'-end processing». Genes & Development 16 (1): 58–71. January 2002. doi:. PMID 11782445.
- ↑ «A 3' exonuclease that specifically interacts with the 3' end of histone mRNA». Molecular Cell 12 (2): 295–305. August 2003. doi:. PMID 14536070.
- ↑ «Phosphorylation of stem-loop binding protein (SLBP) on two threonines triggers degradation of SLBP, the sole cell cycle-regulated factor required for regulation of histone mRNA processing, at the end of S phase». Molecular and Cellular Biology 23 (5): 1590–601. March 2003. doi:. PMID 12588979. PMC 151715. https://archive.org/details/sim_molecular-and-cellular-biology_2003-03_23_5/page/1590.
- ↑ «Cajal bodies are linked to genome conformation». Nature Communications 7: 10966. March 2016. doi:. PMID 26997247. Bibcode: 2016NatCo...710966W.
- ↑ «NPAT links cyclin E-Cdk2 to the regulation of replication-dependent histone gene transcription». Genes & Development 14 (18): 2283–97. September 2000. doi:. PMID 10995386.
- ↑ Luck JM (1965). «Histone Chemistry: the Pioneers». Στο: Bonner J, Ts'o P. The Nucleohistones. San Francisco, London, and Amsterdam: Holden-Day, Inc.
- ↑ 116,0 116,1 «Chapters from my life». Annual Review of Plant Physiology and Plant Molecular Biology 45: 1–23. 1994. doi:.
- ↑ «Histone, a suppressor of chromosomal RNA synthesis». Proceedings of the National Academy of Sciences of the United States of America 48 (7): 1216–22. July 1962. doi:. PMID 14036409. Bibcode: 1962PNAS...48.1216H.
- ↑ «Huang R C & Bonner J. Histone, a suppressor of chromosomal RNA synthesis. Proc. Nat. Acad. Sci. US 48:1216-22, 1962.». Citation Classics (12): 79. 20 March 1978. http://garfield.library.upenn.edu/classics1978/A1978EP01300002.pdf.
- ↑ 119,0 119,1 James Bonner and Paul T'so (1965) The Nucleohistones. Holden-Day Inc, San Francisco, London, Amsterdam.
- ↑ 120,0 120,1 «Calf and pea histone IV. 3. Complete amino acid sequence of pea seedling histone IV; comparison with the homologous calf thymus histone». The Journal of Biological Chemistry 244 (20): 5669–79. October 1969. doi:. PMID 5388597.
- ↑ «Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis». Proceedings of the National Academy of Sciences of the United States of America 51 (5): 786–94. May 1964. doi:. PMID 14172992. Bibcode: 1964PNAS...51..786A.
- ↑ «Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones». Cell 49 (2): 203–10. April 1987. doi:. PMID 3568125. https://pubmed.ncbi.nlm.nih.gov/3568125/.
- ↑ «Extremely conserved histone H4 N terminus is dispensable for growth but essential for repressing the silent mating loci in yeast». Cell 55 (1): 27–39. October 1988. doi:. PMID 3048701. https://www.cell.com/cell/pdf/0092-8674(88)90006-2.pdf?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2F0092867488900062%3Fshowall%3Dtrue.
- ↑ «RNA synthesis and histone acetylation during the course of gene activation in lymphocytes». Proceedings of the National Academy of Sciences of the United States of America 55 (4): 805–12. April 1966. doi:. PMID 5219687. Bibcode: 1966PNAS...55..805P.
- ↑ «Yeast histone H4 N-terminal sequence is required for promoter activation in vivo». Cell 65 (6): 1023–31. June 1991. doi:. PMID 2044150. https://www.cell.com/cell/pdf/0092-8674(91)90554-C.pdf?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2F009286749190554C%3Fshowall%3Dtrue.
- ↑ «Tetrahymena histone acetyltransferase A: a homolog to yeast Gcn5p linking histone acetylation to gene activation». Cell 84 (6): 843–51. March 1996. doi:. PMID 8601308.
- ↑ «The conformation of histone H5. Isolation and characterisation of the globular segment». European Journal of Biochemistry 88 (2): 363–71. August 1978. doi:. PMID 689022.
Εξωτερικοί σύνδεσμοι[Επεξεργασία | επεξεργασία κώδικα]
- HistoneDB 2.0 - Database of histones and variants at NCBI
- Chromatin, Histones & Cathepsin; PMAP The Proteolysis Map-animation
|
