Ανθρώπινο γονιδίωμα
Το ανθρώπινο γονιδίωμα (human genome) είναι το πλήρες σύνολο των αλληλουχιών των νουκλεϊκών οξέων στους ανθρώπους, κωδικοποιημένο ως DNA σε 23 ζεύγη χρωμοσωμάτων στον κυτταρικό πυρήνα και σε ένα μικρό μόριο DNA που βρίσκεται στα ατομικά μιτοχόνδρια. Αυτό συνήθως εξετάζεται ξεχωριστά ως το πυρηνικό γονιδίωμα και το μιτοχονδριακό γονιδίωμα.[1] Τα ανθρώπινα γονιδιώματα περιλαμβάνουν και γονίδια DNA που κωδικοποιούν πρωτεΐνες και μη κωδικοποιητικό DNA. Τα απλοειδή ανθρώπινα γονιδιώματα, που περιέχονται στα γεννητικά κύτταρα (τους κυτταρικούς γαμέτες ωαρίων και σπερματοζωαρίων δημιουργούνται στη φάση της μείωσης της σεξουαλικής αναπαραγωγής πριν η γονιμοποίηση δημιουργήσει ένα ζυγωτό) που αποτελείται από τρία δισεκατομμύρια ζεύγη βάσεων DNA, ενώ τα διπλοειδή γονιδιώματα (που βρίσκονται στα σωματικά κύτταρα) έχουν δυο φορές το περιεχόμενο του DNA. Ενώ υπάρχουν σημαντικές διαφορές μεταξύ των γονιδιωμάτων των ανθρώπων (0,1% μέχρι 0,6% μαζί με τα Indels ),[2] αυτές είναι σημαντικά πιο μικρές από τις διαφορές μεταξύ των ανθρώπων και των πλησιέστερων ζωντανών συγγενών τους των χιμπαντζήδων (περίπου 4%[3]) και των μπονόμπο.
Οι πρώτες αλληλουχίες ανθρωπίνου γονιδιώματος δημοσιεύτηκαν σε σχεδόν πλήρως πρόχειρη μορφή τον Φεβρουάριο του 2001 από το Human Genome Project[4] και την Celera Corporation.[5] Η ολοκλήρωση του έργου της αλληλουχίας του ανθρώπινου γονιδιώματος δημοσιεύτηκε στο Completion of the Human Genome Project Sequence το 2004.[6] Το ανθρώπινο γονιδίωμα ήταν το πρώτο από όλα τα σπονδυλωτά του οποίου ολοκληρώθηκε η αλληλουχία. Μέχρι το 2012, έχει ολοκληρωθεί η αλληλουχία σε χιλιάδες ανθρώπινα γονιδιώματα και σε πολλά περισσότερα έχει χαρτογραφηθεί σε χαμηλότερα επίπεδα ανάλυσης. Αυτά τα δεδομένα χρησιμοποιούνται παγκοσμίως στη βιοϊατρική, στην ανθρωπολογία, στην ιατροδικαστική και σε άλλους κλάδους της επιστήμης. Υπάρχει πλατιά προσδοκία ότι οι γονιδιωματικές μελέτες θα οδηγήσουν σε προόδους στη διάγνωση και αντιμετώπιση των ασθενειών και σε νέες ιδέες σε πολλά πεδία της βιολογίας, συμπεριλαμβανομένης της ανθρώπινης εξέλιξης.
Αν και η αλληλουχία του ανθρώπινου γονιδιώματος έχει καθοριστεί (σχεδόν) πλήρως από την αλληλουχία του DNA, δεν είναι ακόμα πλήρως κατανοητή. Τα περισσότερα (αν και προφανώς όχι όλα) τα γονίδια έχουν ταυτοποιηθεί με συνδυασμό υψηλών αποτελεσμάτων πειραματικών και βιοπληροφορικών προσεγγίσεων, απαιτείται ακόμα πολλή εργασία για την παραπέρα διευκρίνιση των βιολογικών λειτουργιών των πρωτεϊνών τους και των προϊόντων RNA. Πρόσφατα αποτελέσματα προτείνουν ότι οι περισσότερες τεράστιες ποσότητες του μη κωδικοποιητικού DNA στο γονιδίωμα έχουν συσχετιστεί με βιοχημικές δραστηριότητες, συμπεριλαμβανομένης της ρύθμισης έκφρασης των γονιδίων (regulation of gene expression), της χρωμοσωμικής αρχιτεκτονικής και σημάτων που ελέγχουν την επιγενετική κληρονομικότητα (epigenetic inheritance).
Εκτιμάται ότι υπάρχουν 19.000-20.000 ανθρώπινα γονίδια που κωδικοποιούν πρωτεΐνες.[7] Η εκτίμηση του αριθμού των ανθρώπινων γονιδίων έχει αναθεωρηθεί επανειλημμένα προς τα κάτω από τις αρχικές προβλέψεις των 100.000 ή περισσοτέρων, λόγω της βελτίωσης των μεθόδων εύρεσης γονιδιώματος και μπορεί να συνεχίσει να μειώνεται.[6][8] Οι αλληλουχίες που κωδικοποιούν πρωτεΐνη εκφράζουν μόνο ένα πολύ μικρό κλάσμα του γονιδιώματος (περίπου 1,5%) και οι υπόλοιπες συνδυάζονται με μόρια μη κωδικοποιητικού RNA, ρυθμιστικές αλληλουχίες DNA, μακριές διάσπαρτες αλληλουχίες (LINEs), κοντά διάσπαρτα πυρηνικά στοιχεία (SINEs), εσώνια, και αλληλουχίες για τις οποίες μέχρι τώρα δεν έχει καθοριστεί καμία λειτουργία.[9]
Τον Ιούνιο του 2016, ανακοινώθηκε επίσημα το (Human Genome Project - Write ή HGP-Write), ένα σχέδιο σύνθεσης του ανθρώπινου γονιδιώματος.[10][11]
Μοριακή οργάνωση και περιεκτικότητα σε γονίδια[Επεξεργασία | επεξεργασία κώδικα]
Το συνολικό μήκος του ανθρώπινου γονιδιώματος είναι πάνω από 3 δισεκατομμύρια ζεύγη βάσεων. Το γονιδίωμα είναι οργανωμένο σε 22 ζεύγη χρωμοσωμάτων, συν το χρωμόσωμα Χ (ένα για τους άνδρες, δύο για τις γυναίκες) και, μόνο για τους άνδρες ένα χρωμόσωμα Υ. Αυτά είναι όλα μεγάλα γραμμικά μόρια DNA που περιέχονται μέσα στον κυτταρικό πυρήνα. Το γονιδίωμα περιλαμβάνει επίσης το μιτοχονδριακό DNA, ένα συγκριτικά μικρό κυκλικό μόριο που είναι παρόν σε κάθε μιτοχόνδριο. Οι βασικές πληροφορίες για αυτά τα μόρια και τη γονιδιακή τους περιεκτικότητα, βασίζονται σε ένα γονιδίωμα αναφοράς (reference genome) που δεν εκφράζει την αλληλουχία οποιουδήποτε ατόμου, και παρέχονται στον επόμενο πίνακα. (Πηγή δεδομένων: Ensembl genome browser release 87 Αρχειοθετήθηκε 2021-03-09 στο Wayback Machine., Δεκέμβριος του 2016 για τις περισσότερες τιμές·Ensembl genome browser release 68, Ιούλιος του 2012 για miRNA, rRNA, snRNA, snoRNA.)
| Χρωμόσωμα | Μήκος (mm) |
Ζεύγη βάσεων |
Παραλλαγές | Γονίδια που κωδικοποιούν πρωτεΐνη |
Ψευδο- γονίδια |
Συνολικό μήκος ncRNA |
Συνολικό μικρό ncRNA |
miRNA | rRNA | snRNA | snoRNA | Διάφορα ncRNA |
Σύνδεσμοι | Θέση κεντρομερούς (Mbp) |
Αθροιστικά (%) |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
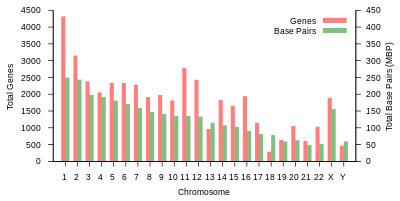
| 1 | 85 | 248.956.422 | 12.151.146 | 2058 | 1220 | 1200 | 496 | 134 | 66 | 221 | 145 | 192 | EBI | 125 | 7,9 |
| 2 | 83 | 242.193.529 | 12.945.965 | 1309 | 1023 | 1037 | 375 | 115 | 40 | 161 | 117 | 176 | EBI | 93,3 | 16,2 |
| 3 | 67 | 198.295.559 | 10.638.715 | 1078 | 763 | 711 | 298 | 99 | 29 | 138 | 87 | 134 | EBI | 91 | 23 |
| 4 | 65 | 190.214.555 | 10.165.685 | 752 | 727 | 657 | 228 | 92 | 24 | 120 | 56 | 104 | EBI | 50,4 | 29,6 |
| 5 | 62 | 181.538.259 | 9.519.995 | 876 | 721 | 844 | 235 | 83 | 25 | 106 | 61 | 119 | EBI | 48,4 | 35,8 |
| 6 | 58 | 170.805.979 | 9.130.476 | 1048 | 801 | 639 | 234 | 81 | 26 | 111 | 73 | 105 | EBI | 61 | 41,6 |
| 7 | 54 | 159.345.973 | 8.613.298 | 989 | 885 | 605 | 208 | 90 | 24 | 90 | 76 | 143 | EBI | 59,9 | 47,1 |
| 8 | 50 | 145.138.636 | 8.221.520 | 677 | 613 | 735 | 214 | 80 | 28 | 86 | 52 | 82 | EBI | 45,6 | 52 |
| 9 | 48 | 138.394.717 | 6.590.811 | 786 | 661 | 491 | 190 | 69 | 19 | 66 | 51 | 96 | EBI | 49 | 56,3 |
| 10 | 46 | 133.797.422 | 7.223.944 | 733 | 568 | 579 | 204 | 64 | 32 | 87 | 56 | 89 | EBI | 40,2 | 60,9 |
| 11 | 46 | 135.086.622 | 7.535.370 | 1298 | 821 | 710 | 233 | 63 | 24 | 74 | 76 | 97 | EBI | 53,7 | 65,4 |
| 12 | 45 | 133.275.309 | 7.228.129 | 1034 | 617 | 848 | 227 | 72 | 27 | 106 | 62 | 115 | EBI | 35,8 | 70 |
| 13 | 39 | 114.364.328 | 5.082.574 | 327 | 372 | 397 | 104 | 42 | 16 | 45 | 34 | 75 | EBI | 17,9 | 73,4 |
| 14 | 36 | 107.043.718 | 4.865.950 | 830 | 523 | 533 | 239 | 92 | 10 | 65 | 97 | 79 | EBI | 17,6 | 76,4 |
| 15 | 35 | 101.991.189 | 4.515.076 | 613 | 510 | 639 | 250 | 78 | 13 | 63 | 136 | 93 | EBI | 19 | 79,3 |
| 16 | 31 | 90.338.345 | 5.101.702 | 873 | 465 | 799 | 187 | 52 | 32 | 53 | 58 | 51 | EBI | 36,6 | 82 |
| 17 | 28 | 83.257.441 | 4.614.972 | 1197 | 531 | 834 | 235 | 61 | 15 | 80 | 71 | 99 | EBI | 24 | 84,8 |
| 18 | 27 | 80.373.285 | 4.035.966 | 270 | 247 | 453 | 109 | 32 | 13 | 51 | 36 | 41 | EBI | 17,2 | 87,4 |
| 19 | 20 | 58.617.616 | 3.858.269 | 1472 | 512 | 628 | 179 | 110 | 13 | 29 | 31 | 61 | EBI | 26,5 | 89,3 |
| 20 | 21 | 64.444.167 | 3.439.621 | 544 | 249 | 384 | 131 | 57 | 15 | 46 | 37 | 68 | EBI | 27,5 | 91,4 |
| 21 | 16 | 46.709.983 | 2.049.697 | 234 | 185 | 305 | 71 | 16 | 5 | 21 | 19 | 24 | EBI | 13,2 | 92,6 |
| 22 | 17 | 50.818.468 | 2.135.311 | 488 | 324 | 357 | 78 | 31 | 5 | 23 | 23 | 62 | EBI | 14,7 | 93,8 |
| X | 53 | 156.040.895 | 5.753.881 | 842 | 874 | 271 | 258 | 128 | 22 | 85 | 64 | 100 | EBI | 60,6 | 99,1 |
| Y | 20 | 57.227.415 | 211.643 | 71 | 388 | 71 | 30 | 15 | 7 | 17 | 3 | 8 | EBI | 12,5 | 100 |
| mtDNA | 0,0054 | 16.569 | 929 | 13 | 0 | 0 | 24 | 0 | 2 | 0 | 0 | 0 | EBI | N/A | 100 |
| σύνολο | 3.088.286.401 | 155.630.645 | 20412 | 14600 | 14727 | 5037 | 1756 | 532 | 1944 | 1521 | 2213 | ν |
Ο παραπάνω Πίνακας 1 συνοψίζει τη φυσική οργάνωση και την περιεκτικότητα σε γονίδια του ανθρωπίνου γονιδιώματος αναφοράς, με συνδέσμους στις πρωτότυπες αναλύσεις, όπως δημοσιεύονται στη βάση δεδομένων Ensembl στο European Bioinformatics Institute (EBI) και στο Wellcome Trust Sanger Institute. Τα μήκη των χρωμοσωμάτων εκτιμήθηκαν πολλαπλασιάζοντας τον αριθμό των ζευγών βάσεων επί 0,34 νανόμετρα, την απόσταση μεταξύ των ζευγών βάσεων στη διπλή έλικα του DNA. Μια πρόσφατη εκτίμηση των μηκών του ανθρώπινου γονιδιώματος βασίστηκε σε ενημερωμένες αναφορές δεδομένων 205,00 cm για το διπλοειδές ανδρικό γονιδίωμα και 208,23 cm για το γυναικείο, που αντιστοιχούν σε βάρη 6,41 και 6,51 πικογραμμαρίων (pg), αντιστοίχως.[12] Ο αριθμός των πρωτεϊνών βασίζεται στον αριθμό των αρχικών προδρόμων μεταγραφημάτων mRNA και δεν περιλαμβάνει προϊόντα του εναλλακτικού ματίσματος προ-mRNA, ή τροποποιήσεις στη δομή της πρωτεΐνης που συμβαίνουν μετά τη μετάφραση.
Οι ποικιλομορφίες είναι μοναδικές διαφορές αλληλουχίας του DNA που έχουν ταυτοποιηθεί στις ατομικές ανθρώπινες αλληλουχίες του γονιδιώματος αναλυμένες από το Ensembl έως τον Δεκέμβριο του 2016. Ο αριθμός των ταυτοποιημένων παραλλαγών αναμένεται να αυξηθεί καθώς επιπλέον προσωπικά γονιδιώματα ταξινομούνται και αναλύονται. Πέρα από την περιεκτικότητα σε γονίδια που εμφανίζεται σε αυτόν τον πίνακα, ένας μεγάλος αριθμός μη εκφραζόμενων λειτουργικών αλληλουχιών έχει ταυτοποιηθεί στο ανθρώπινο γονιδίωμα (δείτε παρακάτω). Σύνδεσμοι ανοίγουν παράθυρα στις παραπεμπόμενες χρωμοσωμικές αλληλουχίες στον πλοηγητή γονιδιώματος EBI.
Μικρά μη κωδικοποιητικά RNA είναι RNA από έως και 200 βάσεις που δεν έχουν δυνατότητα πρωτεϊνικής κωδικοποίησης. Αυτά συμπεριλαμβάνουν: μικροRNA, ή miRNAs (μεταμεταγραφικούς ρυθμιστές (post-transcriptional regulators)) της έκφρασης των γονιδίων, μικρό πυρηνικό RNA, ή snRNAs (τα συστατικά RNA των ματισωμάτων (spliceosomes)) και μικρών πυρηνισκικών (small nucleolar RNAs), ή snoRNA (που εμπλέκονται στην καθοδήγηση χημικών τροποποιήσεων σε άλλα μόρια RNA). Μακριά μη κωδικοποιητικά RNA είναι μόρια RNA μεγαλύτερα από 200 βάσεις που δεν έχουν δυνατότητα πρωτεϊνικής κωδικοποίησης. Αυτά συμπεριλαμβάνουν: ριβοσωμικά RNAs, ή rRNAs (τα συστατικά RNA των ριβοσωμάτων) και ποικίλα άλλα μακριά RNAs που εμπλέκονται στη ρύθμιση της έκφρασης των γονιδίων (regulation of gene expression), επιγενετικές τροποποιήσεις των νουκλεοτιδίων του DNA και πρωτεϊνών ιστόνης, καθώς και ρύθμιση της ενεργότητας των γονιδίων που κωδικοποιούν πρωτεΐνες. Μικρές διαφορές μεταξύ συνολικών αριθμών μη κωδικοποιητικών RNA και των αριθμών των ειδικών τύπων των μικρών ncRNAs εμφανίζονται από τις προηγούμενες τιμές που παίρνουν ως πηγή το Ensembl έκδοση 87 και τα τελευταία από το Ensembl έκδοση 68.
Ολοκλήρωση της αλληλουχίας του ανθρώπινου γονιδιώματος[Επεξεργασία | επεξεργασία κώδικα]
Αν και έχει προσδιοριστεί πλήρως το ανθρώπινο γονιδίωμα για πρακτικούς σκοπούς, υπάρχουν ακόμα εκατοντάδες κενά στην αλληλουχία. Μια πρόσφατη μελέτη σημείωσε περισσότερα από 160 ευχρωματικά κενά, από τα οποία έκλεισαν 50 κενά.[13] Όμως, υπάρχουν ακόμα πολυάριθμα κενά στα ετεροχρωματικά τμήματα του γονιδιώματος που είναι πολύ πιο δύσκολα στην αλληλουχία DNA λόγω των πολυάριθμων επαναλαμβανόμενων DNA και άλλων δυσεπίλυτων χαρακτηριστικών ακολουθιών.
Περιεχόμενο πληροφοριών[Επεξεργασία | επεξεργασία κώδικα]
Το ανθρώπινο γονιδίωμα αναφοράς (GRC v38) έχει συμπιεστεί επιτυχώς σε ~5,2-φορές (οριακά λιγότερο από 550 MB) σε 155 λεπτά χρησιμοποιώντας έναν επιτραπέζιο υπολογιστή με 6,4 GB RAM.[14]
Το απλοειδές ανθρώπινο γονιδίωμα (των 23 χρωμοσωμάτων) έχει μήκος περίπου 3 δισεκατομμύρια ζεύγη βάσεων και περιέχει περίπου 30.000 γονίδια.[15] Επειδή κάθε ζεύγος βάσεων μπορεί να κωδικοποιηθεί από 2 δυαδικά ψηφία, αυτό είναι περίπου 750 megabytes δεδομένων. Ένα μεμονωμένο σωματικό (διπλοειδές) κύτταρο περιέχει δυο φορές αυτήν την ποσότητα, δηλαδή περίπου 6 δισεκατομμύρια ζεύγη βάσεων. Οι άνδρες έχουν λιγότερα ζεύγη βάσεων από τις γυναίκες επειδή το χρωμόσωμα Υ έχει περίπου 57 εκατομμύρια ζεύγη βάσεων, ενώ το χρωμόσωμα Χ έχει έχει περίπου 156 εκατομμύρια, αλλά από πλευράς πληροφοριών οι άνδρες έχουν περισσότερα επειδή το δεύτερο Χ περιέχει σχεδόν τις ίδιες πληροφορίες όπως το πρώτο. Επειδή τα ατομικά γονιδιώματα διαφέρουν στην αλληλουχία κατά λιγότερο από 1% μεταξύ τους, οι παραλλαγές ενός δεδομένου ανθρωπίνου γονιδιώματος από μια κοινή αναφορά μπορεί να συμπιεστεί χωρίς απώλειες σε χοντρικά 4 megabytes.[16]
Ο ρυθμός εντροπίας του γονιδιώματος διαφέρει σημαντικά μεταξύ κωδικοποιητικών και μη κωδικοποιητικών αλληλουχιών. Είναι κοντά στο μέγιστο των 2 δυαδικών ψηφίων ανά ζεύγος βάσεων για τις αλληλουχίες κωδικοποίησης (περίπου 45 εκατομμύρια ζεύγη βάσεων), αλλά λιγότερο για τα μη κωδικοποιητικά τμήματα. Κυμαίνεται μεταξύ 1,5 και 1,9 δυαδικά ψηφία ανά ζεύγος βάσης για το μεμονωμένο χρωμόσωμα, εκτός από το χρωμόσωμα Υ, που έχει ρυθμό εντροπίας κάτω από 0,9 δυαδικά ψηφία ανά ζεύγος βάσεων.[17]
Κωδικοποιητικό συγκριτικά με το μη κωδικοποιητικό DNA[Επεξεργασία | επεξεργασία κώδικα]
Το περιεχόμενο του ανθρωπίνου γονιδιώματος διαιρείται συνήθως σε κωδικοποιητικές και μη κωδικοποιητικές αλληλουχίες DNA. Το κωδικοποιητικό DNA καθορίζεται ως αυτές οι αλληλουχίες που μπορούν να μεταγραφούν στο mRNA και να μεταφραστούν σε πρωτεΐνες στον ανθρώπινο κύκλο ζωής· αυτές οι αλληλουχίες καταλαμβάνουν μόνο ένα μικρό κλάσμα του γονιδιώματος (μικρότερο από 2%). Το μη κωδικοποιητικό DNA αποτελείται από όλες αυτές τις αλληλουχίες (περίπου 98% του γονιδιώματος) που δεν χρησιμοποιούνται να κωδικοποιήσουν πρωτεΐνες.
Κάποιο μη κωδικοποιητικό DNA περιέχει γονίδια για μόρια RNA με σημαντικές βιολογικές λειτουργίες (μη κωδικοποιητικό RNA, παραδείγματος χάρη ριβοσωμικό RNA και μεταφορικό RNA). Η εξερεύνηση των λειτουργιών και της εξελικτικής προέλευσης του μη κωδικοποιητικού DNA είναι ένας σημαντικός σκοπός της σύγχρονης αναζήτησης του γονιδιώματος, που περιλαμβάνει το έργο ENCODE (Εγκυκλοπαίδεια των στοιχείων του DNA (Encyclopedia of DNA Elements)), που αποσκοπεί στην έρευνα ολόκληρου του ανθρωπίνου γονιδιώματος, με τη χρήση ποικιλίας πειραματικών εργαλείων που τα αποτελέσματά τους είναι ενδεικτικά της μοριακής δραστικότητας.
Επειδή το μη κωδικοποιητικό DNA ξεπερνά κατά πολύ το κωδικοποιητικό DNA, η έννοια του γονιδιώματος με ακολουθίες έχει γίνει μια πιο εστιασμένη αναλυτική έννοια παρά η κλασική έννοια των γονιδίων με κωδικοποιητικό DNA.[18][19]
Κωδικοποιητικές αλληλουχίες (γονίδια με κωδικοποιητικές πρωτεΐνες)[Επεξεργασία | επεξεργασία κώδικα]
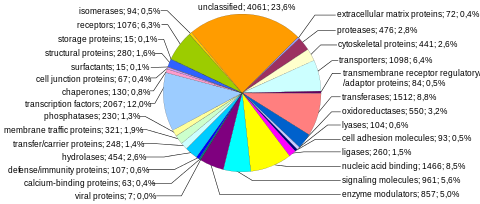
Οι αλληλουχίες κωδικοποιητικών πρωτεϊνών αντιπροσωπεύουν το πιο πλατιά μελετημένο και καλύτερα κατανοητό συστατικό του ανθρωπίνου γονιδιώματος. Αυτές οι αλληλουχίες οδηγούν τελικά στην παραγωγή όλων των ανθρωπίνων πρωτεϊνών, αν και αρκετές βιολογικές διαδικασίες (π.χ. ανασυνδυασμοί DNA και εναλλακτικό μάτισμα προ-mRNA (alternative pre-mRNA splicing) μπορεί να οδηγήσουν στην παραγωγή πολύ περισσοτέρων μοναδικών πρωτεϊνών από τον αριθμό των γονιδίων που κωδικοποιούν πρωτεΐνες.
Η πλήρης τμηματική ικανότητα κωδικοποίησης πρωτεϊνών του γονιδιώματος περιέχεται στο εξονίωμα (exome) και αποτελείται από αλληλουχίες DNA κωδικοποιημένες από εξώνια που μπορούν να μεταφραστούν σε πρωτεΐνες. Λόγω της βιολογικής σπουδαιότητάς της και επειδή αποτελεί λιγότερο από το 2% του γονιδιώματος, η αλληλουχία του εξονιώματος ήταν το πρώτο μεγάλο ορόσημο του έργου του ανθρωπίνου γονιδιώματος.
Αριθμός γονιδίων της κωδικοποιητικής πρωτεΐνης. Περίπου 20.000 ανθρώπινες πρωτεΐνες έχουν καταχωριστεί σε βάσεις δεδομένων όπως η Uniprot.[21] Ιστορικά, εκτιμήσεις για τον αριθμό των γονιδίων στις πρωτεΐνες έχουν ποικίλει πολύ, φτάνοντας μέχρι 2.000.000 στο τέλος της δεκαετίας του 1960,[22] αλλά αρκετοί ερευνητές επισήμαναν στις αρχές της δεκαετίας του 1970 ότι το εκτιμώμενο μεταλλαξιογόνο φορτίο (|mutational load) από επιβλαβείς μεταλλάξεις τοποθετούσε ένα ανώτερο όριο περίπου 40.000 για τον συνολικό αριθμό των λειτουργικών θέσεων (συμπεριλαμβανομένων των κωδικοποιητικών πρωτεϊνών και των λειτουργικών μη κωδικοποιητικών γονιδίων).[23]
Ο αριθμός των ανθρωπίνων γονιδίων που κωδικοποιούν πρωτεΐνες δεν είναι σημαντικά μεγαλύτερος από αυτόν πολλών λιγότερο περίπλοκων οργανισμών, όπως ο καινοραβδίτης ο κομψός (Caenorhabditis elegans (ασκαρίδα)) και η φρουτόμυγα. Αυτή η διαφορά μπορεί να προέκυψε από την εκτεταμένη χρήση του εναλλακτικού ματίσματος προ-mRNA στους ανθρώπους, που δίνει τη δυνατότητα οικοδόμησης ένας πολύ μεγάλου αριθμού τμηματικών πρωτεϊνών μέσω της επιλεκτικής ενσωμάτωσης των εξωνίων.
Η χωρητικότητα κωδικοποιητικών πρωτεϊνών ανά χρωμόσωμα. Τα γονίδια που κωδικοποιούν πρωτεΐνες κατανέμονται ανόμοια στα χρωμοσώματα, από μερικές δεκάδες μέχρι περισσότερα από 2000, με ιδιαίτερα υψηλή πυκνότητα γονιδίων στα χρωμοσώματα 19, 11 και 1 (Πίνακας 1). Κάθε χρωμόσωμα περιέχει διάφορες περιοχές φτωχές και πλούσιες σε γονίδια, που μπορεί να συσχετίζονται με χρωμοσωμικές ζώνες και περιεχόμενο γουανίνης-κυτοσίνης (GC-content).[24] Η σπουδαιότητα αυτών των μη τυχαίων μοτίβων της πυκνότητας των γονιδίων δεν είναι καλά κατανοητή.[25]
Μέγεθος γονιδίων που κωδικοποιούν πρωτεΐνες. Το μέγεθος των γονιδίων που κωδικοποιούν πρωτεΐνες στο ανθρώπινο γονιδίωμα εμφανίζει τεράστια ποικιλία (Πίνακας 2). Το μέσο μέγεθος ενός γονιδίου που κωδικοποιεί πρωτεΐνη είναι 26.288 bp (διάμεσος = 66.577 bp; Πίνακας 2 στο [26]). Παραδείγματος χάρη, το γονίδιο για την ιστόνη H1a (HIST1HIA) είναι σχετικά μικρό και απλό, που του λείπουν εσώνια με 781 nt αλληλουχίες κωδικοποίησης mRNA και μια πρωτεΐνη 215 αμινοξέων (648 nt ανοικτού πλαισίου ανάγνωσης). H δυστροφίνη (Dystrophin) (DMD) έχει το μεγαλύτερο γονίδιο που κωδικοποιεί πρωτεΐνη στο ανθρώπινο γονιδίωμα αναφοράς, εκτεινόμενο συνολικά σε 2,2 MB, ενώ η τιτίνη (TTN) έχει τη μακρύτερη αλληλουχία κωδικοποίησης (114.414 bp), με τον μεγαλύτερο αριθμό εξωνίων (363),[27] και το μακρύτερο απλό εξώνιο (17.106 bp). Όσον αφορά το συνολικό γονιδίωμα, το μέσο μέγεθος ενός εξωνίου είναι 122 bp (διάμεσος = 145 bp), ο μέσος αριθμός εξωνίων είναι is 7 (διάμεσος = 8,8) και η μέση αλληλουχία κωδικοποίησης κωδικοποιεί 367 αμινοξέα (διάμεσος = 447 αμινοξέα· Πίνακας 21 στο [9] ).
| Πρωτεΐνη | Χρωμ | Γονίδιο | Μήκος | Εξώνια | Μήκος εξωνίων | Μήκος εσωνίων | Εναλ. μάτισμα |
|---|---|---|---|---|---|---|---|
| Πρωτεΐνη ευπάθειας καρκίνου μαστού τύπου 2 (BRCA2) | 13 | BRCA2 | 83,736 | 27 | 11,386 | 72,350 | ναι |
| Ρυθμιστής διαμεμβρανικής αγωγιμότητας κυστικής ίνωσης | 7 | CFTR | 202,881 | 27 | 4,440 | 198,441 | ναι |
| Κυτόχρωμα b | MT | MTCYB | 1,140 | 1 | 1,140 | 0 | όχι |
| Δυστροφίνη | X | DMD | 2,220,381 | 79 | 10,500 | 2,209,881 | ναι |
| Αφυδρογονάση της 3-φωσφορικής γλυκεριναλδεΰδης | 12 | GAPDH | 4,444 | 9 | 1,425 | 3,019 | ναι |
| Υπομονάδα βήτα της αιμογλοβίνης | 11 | HBB | 1,605 | 3 | 626 | 979 | όχι |
| Ιστόνη H1A | 6 | HIST1H1A | 781 | 1 | 781 | 0 | όχι |
| Τιτίνη | 2 | TTN | 281,434 | 364 | 104,301 | 177,133 | ναι |
Πίνακας 2. Παραδείγματα γονιδίων που κωδικοποιούν ανθρώπινες πρωτεΐνες. Χρωμ, χρωμόσωμα. Εναλ. μάτισμα, εναλλακτικό μάτισμα προ-mRNA. (Πηγή δεδομένων: Ensembl genome browser έκδοση 68, Ιούλιος 2012)
Πρόσφατα, μια συστηματική μετα-ανάλυση των ενημερωμένων δεδομένων του ανθρωπίνου γονιδιώματος [26] βρήκε ότι το μεγαλύτερο κωδικοποιητικό γονίδιο στο ανθρώπινο γονιδίωμα αναφοράς είναι το RBFOX1 (πρωτεΐνη πρόσδεσης RNA, fox-1 ομόλογο 1), που επεκτείνεται συνολικά σε 2,47 MB. Σε ολόκληρο το γονιδίωμα, εξετάζοντας ένα επιμελημένο σύνολο των γονιδίων που κωδικοποιούν πρωτεΐνες, το διάμεσο μέγεθος ενός εξωνίου εκτιμάται τώρα στις 133 bp (μέσος όρος = 309 bp), ο διάμεσος αριθμός εξωνίων εκτιμάται τώρα στο 8 (μέος όρος = 11) και η διάμεση αλληλουχία κωδικοποίησης εκτιμάται ότι κωδικοποιεί 425 αμινοξέα (μέσος όρος = 553 αμινοξέα Πίνακες 2 και 5 στο [26]).
Μη κωδικοποιητικό DNA (ncDNA)[Επεξεργασία | επεξεργασία κώδικα]
Ως μη κωδικοποιητικό DNA ορίζονται όλες οι αλληλουχίες στο γονιδίωμα που δεν βρίσκονται σε εξώνια κωδικοποιητικών πρωτεϊνών και έτσι δεν αναπαριστάνονται ποτέ στην αλληλουχία των αμινοξέων των εκφραζόμενων πρωτεϊνών. Σύμφωνα με αυτόν τον ορισμό, περισσότερο από το 98% των ανθρωπίνων γονιδιωμάτων αποτελείται από ncDNA.
Πολυάριθμες τάξεις που δεν κωδικοποιούν DNA έχουν ταυτοποιηθεί, συμπεριλαμβανομένων γονιδίων που δεν κωδικοποιούν RNA (π.χ. tRNA και rRNA), ψευδογονιδίων, εσωνίων, αμετάφραστων περιοχών του mRNA, ρυθμιστικών αλληλουχιών DNA, επαναλαμβανόμενων αλληλουχιών DNA και αλληλουχιών σχετικών με κινητά γενετικά στοιχεία.
Πολυάριθμες αλληλουχίες που συμπεριλαμβάνονται στα γονίδια ορίζονται επίσης ότι δεν κωδικοποιούν DNA. Αυτές συμπεριλαμβάνουν γονίδια που δεν κωδικοποιούν RNA (π.χ. tRNA, rRNA) και αμετάφραστα συστατικά γονιδίων κωδικοποίησης πρωτεϊνών (π.χ. εσώνια και 5' και 3' αμετάφραστες περιοχές του mRNA).
Αλληλουχίες που κωδικοποιούν πρωτεΐνες (ειδικότερα, κωδικοποιητικά εξωνίων) αποτελούν λιγότερο από το 1,5% του ανθρώπινου γονιδιώματος.[9] Επίσης, περίπου το 26% του ανθρωπίνου γονιδιώματος είναι εσώνια.[28] Εκτός από γονίδια (εξώνια και εσώνια) και γνωστές ρυθμιστικές αλληλουχίες (8–20%), το ανθρώπινο γονιδίωμα περιέχει περιοχές που δεν κωδικοποιούν DNA. Ο ακριβής αριθμός που δεν κωδικοποιεί DNA και παίζει ρόλο στη φυσιολογία του κυττάρου έχει αμφισβητηθεί έντονα. Πρόσφατη ανάλυση από το έργο ENCODE δείχνει ότι το 80% του συνολικού ανθρωπίνου γονιδιώματος είναι μεταγραφόμενο, συνδέεται με ρυθμιστικές πρωτεΐνες, ή συσχετίζεται με κάποια άλλη βιοχημική ενέργεια.[8]
Όμως, παραμένει αμφισβητούμενο εάν όλη αυτή η βιοχημική δραστικότητα συνεισφέρει στην κυτταρική φυσιολογία, ή εάν ένα σημαντικό ποσοστό από αυτή είναι το αποτέλεσμα μεταγραφικού και βιοχημικού θορύβου, που πρέπει να φιλτραριστεί ενεργά από τον οργανισμό.[29] Αποκλείοντας τις αλληλουχίες που κωδικοποιούν πρωτεΐνες, εσώνια και ρυθμιστικές περιοχές, μεγάλο μέρος που δεν κωδικοποιεί DNA αποτελείται από: Πολλές αλληλουχίες DNA που δεν παίζουν ρόλο στη γονιδιακή έκφραση έχουν σημαντικές βιολογικές λειτουργίες. Μελέτες της συγκριτικής γονιδιωματικής δείχνουν ότι περίπου το 5% του γονιδιώματος περιέχει αλληλουχίες που δεν κωδικοποιούν DNA που είναι πολύ διατηρούμενες, μερικές φορές σε χρονικές κλίμακες που αναπαριστάνουν εκατοντάδες εκατομμύρια έτη, που σημαίνει ότι αυτές οι μη κωδικοποιητικές περιοχές είναι κάτω από ισχυρή εξελικτική πίεση και θετική επιλογή.[30]
Πολλές από αυτές τις αλληλουχίες ρυθμίζουν τη δομή του χρωμοσώματος περιορίζοντας τις περιοχές σχηματισμού ετεροχρωματίνης και ρυθμίζουν δομικά χαρακτηριστικά των χρωμοσωμάτων, όπως τα τελομερή και τα κεντρομερή. Άλλες μη κωδικοποιητικές περιοχές χρησιμεύουν ως προελεύσεις της αναπαραγωγής του DNA. Τελικά πολλές περιοχές μεταγράφονται σε λειτουργικές που δεν κωδικοποιούν RNA και ρυθμίζουν την έκφραση γονιδίων που κωδικοποιούν πρωτεΐνες (π.χ.[31] ), μετάφραση και σταθερότητα του mRNA, τροποποιήσεις δομής χρωματίνης (συμπεριλαμβανόμενης της ιστόνης, παραδείγματος χάρη[32]), μεθυλίωση DNA (π.χ.[33] ), ανασυνδυασμό DNA (παραδείγματος χάρη[34] ) και διασταυρούμενη ρύθμιση άλλες που δεν κωδικοποιούν RNAs (π.χ.[35] ). Είναι επίσης πιθανό ότι πολλές μεταγραφόμενες μη κωδικοποιητικές περιοχές δεν εξυπηρετούν κανένα ρόλο και αυτή η μεταγραφή είναι το προϊόν μη ειδικής ενέργειας ενεργότητας RNA πολυμεράσης.[29]
Ψευδογονίδια[Επεξεργασία | επεξεργασία κώδικα]
Τα ψευδογονίδια είναι ανενεργά αντίγραφα των γονιδίων που κωδικοποιούν πρωτεΐνες, τα οποία δημιουργούνται συχνά από διπλασιασμό γονιδίου (gene duplication) και έχουν γίνει μη λειτουργικά μέσω της συσσώρευσης απενεργοποιητικών μεταλλάξεων. Ο Πίνακας 1 εμφανίζει ότι ο αριθμός των ψευδογονιδίων στο ανθρώπινο γονιδίωμα είναι της τάξης των 13.000,[36] και σε κάποια χρωμοσώματα είναι σχεδόν ο ίδιος με τον αριθμό των λειτουργικών πρωτεϊνών που κωδικοποιούν πρωτεΐνες. Ο διπλασιασμός των γονιδίων είναι ένας μείζων μηχανισμός μέσω του οποίου δημιουργείται νέο γενετικό υλικό κατά τη μοριακή εξέλιξη.
Παραδείγματος χάρη, η οικογένεια γονιδίων οσφρητικών υποδοχέων είναι ένα από τα καλύτερα τεκμηριωμένα παραδείγματα ψευδογονιδίων στο ανθρώπινο γονιδίωμα. Περισσότερο από το 60 % των γονιδίων σε αυτήν την οικογένεια είναι μη λειτουργικά ψευδογονίδια στους ανθρώπους. Συγκριτικά μόνο το 20% των γονιδίων στην οικογένεια γονιδίων οσφρητικών υποδοχέων των ποντικιών είναι ψευδογονίδια. Η έρευνα δείχνει ότι αυτό είναι ένα χαρακτηριστικό σχετιζόμενο με το είδος, καθώς τα πιο κοντινά σχετικά πρωτεύοντα έχουν όλα αναλογικά λιγότερα ψευδογονίδια. Αυτή η γενετική ανακάλυψη βοηθά στην εξήγηση της λιγότερο οξείας αίσθησης της γεύσης στους ανθρώπους σχετικά με άλλα θηλαστικά.[37]
Γονίδια που δεν κωδικοποιούν RNA (ncRNA)[Επεξεργασία | επεξεργασία κώδικα]
Τα μη κωδικοποιητικά μόρια του RNA παίζουν πολλούς βασικούς ρόλους στα κύτταρα, ειδικότερα σε πολλές αντιδράσεις της πρωτεϊνικής σύνθεσης και της επεξεργασίας του RNA. Το μη κωδικοποιητικό RNA περιλαμβάνει το tRNA, το ριβοσωμικό RNA, το μικροRNA, το snRNA και άλλα γονίδια που δεν κωδικοποιούν RNA συμπεριλαμβανομένων περίπου 60.000 μακριά μη κωδικοποιητικά lncRNA (lncRNAs).[8][38][39][40] Αν και ο αριθμός των αναφερόμενων γονιδίων lncRNA συνεχίζει να αυξάνεται και ο ακριβής αριθμός στο ανθρώπινο γονιδίωμα δεν έχει ακόμα προσδιοριστεί, υποστηρίζεται ότι πολλά από αυτά είναι μη λειτουργικά.[41]
Πολλά ncRNAs είναι κρίσιμα στοιχεία στη ρύθμιση και έκφραση των γονιδίων. Το μη κωδικοποιητικό RNA συνεισφέρει επίσης στην επιγενετική, στη μεταγραφή, στο μάτισμα RNA και στον μεταφραστικό μηχανισμό. Ο ρόλος του RNA στη γενετική ρύθμιση και πάθηση προσφέρει ένα νέο δυνητικό επίπεδο ανεξερεύνητης πολυπλοκότητας.[42]
Εσώνια και αμετάφραστες περιοχές του mRNA[Επεξεργασία | επεξεργασία κώδικα]
Επιπλέον των μορίων ncRNA που κωδικοποιούνται από ξεχωριστά γονίδια, τα αρχικά μεταγραφήματα των πρωτεϊνών που κωδικοποιούν γονίδια περιέχουν συνήθως εκτεταμένες μη κωδικοποιητικές αλληλουχίες, με τη μορφή εσωνίων, 5'-αμετάφραστες περιοχές (5'-UTR) και 3'-αμετάφραστες περιοχές (3'-UTR). Στα περισσότερα γονίδια που κωδικοποιούν πρωτεΐνες του ανθρωπίνου γονιδιώματος, το μήκος των αλληλουχιών των εσωνίων είναι από 10- μέχρι 100-φορές το μήκος των αλληλουχιών των εξωνίων (Πίνακας 2).
Ρυθμιστικές αλληλουχίες DNA[Επεξεργασία | επεξεργασία κώδικα]
Το ανθρώπινο γονιδίωμα έχει πολλές διαφορετικές ρυθμιστικές αλληλουχίες που είναι κρίσιμες στον έλεγχο της γονιδιακής έκφρασης. Συντηρητικές εκτιμήσεις δείχνουν ότι αυτές οι αλληλουχίες αποτελούν το 8% του γονιδιώματος,[43] όμως παρεκβολές από το έργο ENCODE δίνουν ότι 20[44]-40%[45]-40% του γονιδιώματος είναι γονιδιακή ρυθμιστική αλληλουχία. Μερικοί τύποι μη κωδικοποιητικού DNA είναι γενετικοί "διακόπτες" που δεν κωδικοποιούν πρωτεΐνες, αλλά ρυθμίζουν πότε και πού εκφράζονται τα γονίδια (που αποκαλούνται ενισχυτές).[46]
Οι ρυθμιστικές αλληλουχίες είναι γνωστές από τα τέλη της δεκαετίας του 1960.[47] Η πρώτη ταυτοποίηση των ρυθμιστικών αλληλουχιών στο ανθρώπινο γονιδίωμα βασιζόντουσαν στην τεχνολογία ανασυνδυασμένου DNA.[48] Αργότερα, με την εμφάνιση της γονιδιωματικής αλληλουχίας, η ταυτοποίηση αυτών των αλληλουχιών μπορεί να παραχθεί από την εξελικτική διατήρηση. Ο εξελικτικός κλάδος μεταξύ των πρωτευόντων και των ποντικιών, παραδείγματος χάρη, συνέβη 70–90 εκατομμύρια χρόνια πριν.[49] Έτσι οι υπολογιστικές συγκρίσεις των γονιδιακών αλληλουχιών που ταυτοποιούν τις διατηρημένες μη κωδικοποιητικές αλληλουχίες (conserved non-coding sequences) θα είναι ένδειξη της σπουδαιότητάς τους σε δραστηριότητες όπως η γονιδιακή ρύθμιση.[50]
Άλλα γονιδιώματα έχουν ταξινομηθεί με την ίδια επιδίωξη της υποβοήθησης μεθόδων που καθοδηγούνται από τη διατήρηση, π.χ. το γονιδίωμα της τετραοδοντίδας.[51] Όμως οι ρυθμιστικές αλληλουχίες εξαφανίζονται και επαναεξελίσσονται με υψηλούς ρυθμούς.[52][53][54]
Μέχρι το 2012, οι προσπάθειες έχουν μετατοπιστεί προς την εύρεση αλληλεπιδράσεων μεταξύ DNA και ρυθμιστικών πρωτεϊνών με την τεχνική ChIP-Seq, ή στα κενά όπου το DNA δεν είναι πακεταρισμένο με ιστόνες (υπερευαίσθητες θέσης δεοξυριβονουκλεάσης), που και οι δυο τους λένε πού υπάρχουν ενεργές ρυθμιστικές αλληλουχίες στον εξεταζόμενο τύπο κυττάρου.[43]
Επαναλαμβανόμενες αλληλουχίες DNA[Επεξεργασία | επεξεργασία κώδικα]
Οι επαναλαμβανόμενες αλληλουχίες DNA αποτελούν περίπου το 50% του ανθρωπίνου γονιδιώματος.[55]
Περίπου το 8% του ανθρωπίνου γονιδιώματος αποτελείται από διαδοχικές σε σειρά συστοιχίες DNA ή διαδοχικές επαναλήψεις, αλληλουχίες επανάληψης χαμηλής πολυπλοκότητας που έχουν πολλαπλά γειτονικά αντίγραφα (π.χ. "CAGCAGCAG...").[56]Οι διαδοχικές αλληλουχίες μπορεί να είναι μεταβλητών μηκών, από δύο νουκλεοτίδια μέχρι δεκάδες νουκλεοτίδια. Αυτές οι αλληλουχίες είναι πολύ μεταβλητές, ακόμα και μεταξύ στενά συσχετισμένων ατόμων και έτσι χρησιμοποιούνται για γενεαλογικό έλεγχο DNA και ιατροδικαστική ανάλυση DNA.[57]
Οι επαναλαμβανόμενες αλληλουχίες με λιγότερα από δέκα νουκλεοτίδια (π.χ. η δινουκλεοτιδική επανάληψη (AC)n) ορίζονται ως μικροδορυφορικές αλληλουχίες. Μεταξύ των μικροδορυφορικών αλληλουχιών, οι επαναλήψεις τρινουκλεοτιδίων είναι ιδιαίτερης σπουδαιότητας, επειδή μερικές φορές συμβαίνουν στις κωδικοποιητικές περιοχές των γονιδίων για πρωτεΐνες και μπορεί να οδηγήσουν σε γενετικές διαταραχές. Παραδείγματος χάρη, η νόσος του Huntington είναι αποτέλεσμα επέκτασης της επανάληψης του τρινουκλεοτιδίου (CAG)n στο γονίδιο Huntingtin στο ανθρώπινο χρωμόσωμα 4. Τα τελομερή (τα άκρα των γραμμικών χρωμοσωμάτων) καταλήγουν με μια μικροδορυφορική εξανουκλεοτιδική επανάληψη της αλληλουχίας (TTAGGG)n.
Οι τυχαίες επαναλήψεις μεγαλύτερων αλληλουχιών (συστοιχίες επαναλαμβανόμενων αλληλουχιών μήκους 10–60 νουκλεοτιδίων) ονομάζονται μινιδορυφόροι (minisatellites).
Μεταθετά γενετικά στοιχεία (μεταθετόνια (transposons) και τα απομεινάρια τους[Επεξεργασία | επεξεργασία κώδικα]
Τα μεταθετά γενετικά στοιχεία, που είναι αλληλουχίες DNA που μπορούν να αναδιπλασιαστούν και να εισάγουν αντίγραφα του εαυτού τους σε άλλες θέσεις στο γονιδίωμα ξενιστή, είναι άφθονα στο ανθρώπινο γονιδίωμα. Το πιο πλούσιο προγονικό μεταθετόνιο, Alu, έχει περίπου 50.000 ενεργά αντίγραφα,[58] και μπορεί να εισαχθεί σε ενδογονιδιακές και διαγονιδιακές περιοχές[59] Μια άλλη οικογένεια, η LINE-1, έχει 100 περίπου ενεργά αντίγραφα ανά γονιδίωμα (ο αριθμός ποικίλει μεταξύ των ανθρώπων).[60] Μαζί με μη λειτουργικά απομεινάρια των παλιών μεταθετονίων, συναντώνται σε πάνω από το μισό ανθρώπινο DNA.[61], Τα μεταθετόνια έχουν παίξει βασικό ρόλο στη γλυπτική του ανθρωπίνου γονιδιώματος και μερικές φορές ονομάζονται "jumping genes (γονίδια που πηδούν)". Μερικές από αυτές της αλληλουχίες αντιπροσωπεύουν ενδογενείς ρετροϊούς (endogenous retroviruses), αντίγραφα DNA των ιογενών αλληλουχιών που έχουν ενσωματωθεί μόνιμα στο γονιδίωμα και έχουν μεταβιβαστεί τώρα σε διαδοχικές γενιές.
Τα μεταθετά στοιχεία στο ανθρώπινο γονιδίωμα μπορούν να ταξινομηθούν σε ρετρομεταθετόνια LTR (μακριάς τελικής επανάληψης) (8,3% του συνολικού γονιδιώματος), σε βραχέα διάσπαρτα πυρηνικά στοιχεία (SINEs) (13,1% του συνολικού γονιδιώματος) συμπεριλαμβανομένων στοιχείων Alu, μακρά διάσπαρτα πυρηνικά στοιχεία (LINEs) (20,4% του συνολικού γονιδιώματος), SVAs και μεταθετόνια DNA τάξης II (2,9% του συνολικού γονιδιώματος).
Γονιδιωματική ποικιλία στους ανθρώπους[Επεξεργασία | επεξεργασία κώδικα]
Ανθρώπινο γονιδίωμα αναφοράς[Επεξεργασία | επεξεργασία κώδικα]
Με την εξαίρεση των μονοζυγωτικών διδύμων, όλοι οι άνθρωποι εμφανίζουν σημαντική ποικιλία στις γονιδιωματικές αλληλουχίες DNA. Το ανθρώπινο γονιδίωμα αναφοράς (HRG) χρησιμοποιείται ως τυπική αναφορά αλληλουχίας.
Υπάρχουν αρκετά σημαντικά στοιχεία που αφορούν το ανθρώπινο γονιδίωμα αναφοράς:
- Το HRG είναι μια απλοειδής αλληλουχία. Κάθε χρωμόσωμα εμφανίζεται μια φορά.
- Το HRG είναι μια σύνθετη αλληλουχία και δεν αντιστοιχεί σε κανένα ανθρώπινο άτομο.
- Το HRG ενημερώνεται περιοδικά για να διορθώσει σφάλματα, ασάφειες και άγνωστα "κενά".
- Το HRG δεν αντιπροσωπεύει κατά κανένα τρόπο ένα "ιδανικό" ή "τέλειο" ανθρώπινο άτομο. Είναι απλά μια προτυποποιημένη αναπαράσταση που χρησιμοποιείται για συγκριτικούς σκοπούς.
Το Genome Reference Consortium είναι υπεύθυνο για την ενημέρωση του HRG. Η έκδοση 38 εκδόθηκε τον Δεκέμβριο του 2013.[62]
Μέτρηση της ανθρώπινης γενετικής ποικιλίας[Επεξεργασία | επεξεργασία κώδικα]
Οι περισσότερες μελέτες της ανθρώπινης γενετικής ποικιλίας έχει εστιαστεί στους μονονουκλεοτιδικούς πολυμορφισμούς (single-nucleotide polymorphisms ή SNPs), που είναι υποκαταστάσεις σε μεμονωμένες βάσης κατά μήκος ενός γονιδιώματος. Οι περισσότερες αναλύσεις εκτιμούν ότι SNPs συμβαίνουν με αναλογία 1 ανά 1000 ζεύγη βάσεων, κατά μέσο όρο, στο ευχρωματικό ανθρώπινο γονιδίωμα, αν και δεν εμφανίζουν ομοιόμορφη πυκνότητα. Συνεπώς ακολουθεί η δημοφιλής δήλωση ότι "είμαστε όλοι, ανεξάρτητα από φυλή, γενετικά ίδιοι κατά 99,9%",[63] αν και αυτό μπορεί να εξειδικευτεί κάπως από τους περισσότερους γενετιστές. Παραδείγματος χάρη, ένα πολύ μεγαλύτερο κλάσμα του γονιδιώματος θεωρείται τώρα ότι εμπλέκεται στην ποικιλία του αριθμού αντιγράφων (copy number variation).[64] Μια μεγάλης κλίμακας συνεργατική προσπάθεια καταλογοποίησης των ποικιλιών SNP στο ανθρώπινο γονιδίωμα έχει αναληφθεί από το International HapMap Project.
Οι γονιδιωματικές θέσεις και τα μήκη συγκεκριμένων τύπων μικρών επαναλαμβανόμενων αλληλουχιών μεταβάλλονται πολύ από άτομο σε άτομο, που είναι η βάση του αποτυπώματος DNA (DNA fingerprinting) και των τεχνολογιών ελέγχου πατρότητας με DNA. Τα ετεροχρωματικά τμήματα του ανθρώπινου γονιδιώματος, που έχει αρκετές εκατοντάδες εκατομμύρια ζεύγη βάσεων, θεωρείται επίσης να είναι αρκετά μεταβλητό στον ανθρώπινο πληθυσμό (είναι τόσο επαναληπτικές και τόσο μακριές που δεν μπορεί να υπολογιστεί η αλληλουχία τους με ακρίβεια με την τρέχουσα τεχνολογία). Αυτές οι περιοχές περιέχουν λίγα γονίδια και είναι ασαφές εάν κάποιο σημαντικό φαινοτυπικό αποτέλεσμα προκύπτει από την τυπική ποικιλία στις επαναλήψεις ή την ετεροχρωματίνη.
Οι περισσότερες μεγάλες γονιδιωματικές μεταλλάξεις στους γαμέτες των γεννητικών κυττάρων καταλήγουν προφανώς σε μη βιώσιμα έμβρυα· όμως, ένας αριθμός ανθρωπίνων ασθενειών σχετίζεται με γονιδιωματικές ανωμαλίες μεγάλης κλίμακας. Τα σύνδρομο Down, σύνδρομο Τέρνερ και ένας αριθμός άλλων ασθενειών προκύπτουν από μη αποχωρισμό ολόκληρων χρωμοσωμάτων. Τα καρκινικά κύτταρα έχουν συχνά ανευπλοειδία των χρωμοσωμάτων και των χρωμοσωμικών βραχιόνων, αν και μια σχέση αιτίας και αποτελέσματος μεταξύ ανευπλοειδίας και καρκίνου δεν έχει καθοριστεί.
Χάρτης ανθρώπινης γονιδιωματικής ποικιλίας[Επεξεργασία | επεξεργασία κώδικα]
Ενώ μια γονιδιωματική αλληλουχία καταχωρίζει τη σειρά κάθε βάσης DNA στο γονιδίωμα, ένας γονιδιωματικός χάρτης ταυτοποιεί τα σημεία αναφοράς. Ο γονιδιωματικός χάρτης είναι λιγότερο λεπτομερής από τη γονιδιωματική αλληλουχία και βοηθά στην πλοήγηση του γονιδιώματος.[65][66]
Παράδειγμα χάρτη ποικιλιών είναι ο HapMap που αναπτύχθηκε από το International HapMap Project. Ο HapMap είναι ένας απλότυπος (haplotype) χάρτης του ανθρωπίνου γονιδιώματος, "που περιγράφει τα συνηθισμένα πρότυπα της ανθρώπινης ποικιλίας της αλληλουχίας DNA."[67] Καταλογοποιεί τα πρότυπα των ποικιλιών μικρής κλίμακας στο γονιδίωμα που περιλαμβάνουν απλά γράμματα DNA, ή βάσεις.
Οι ερευνητές δημοσίευσαν τον πρώτο χάρτη με βάση την αλληλουχία της δομικής ποικιλίας του ανθρωπίνου γονιδιώματος μεγάλης κλίμακας στο περιοδικό Nature τον Μάιο του 2008.[68][69] Οι δομικές ποικιλίες μεγάλης κλίμακας είναι διαφορές στο γονιδίωμα μεταξύ ανθρώπων και ποικίλουν από μερικές χιλιάδες μέχρι μερικά εκατομμύρια βάσεις DNA· κάποιες είναι προσθήκες ή απώλειες των εκτάσεων της γονιδιωματικής αλληλουχίας και άλλες εμφανίζονται ως αναδιατάξεις των εκτάσεων της αλληλουχίας. Αυτές οι ποικιλίες περιλαμβάνουν διαφορές στον αριθμό των αντιγράφων των ατόμων που έχουν ιδιαίτερα γονίδια, διαγραφές, μετατοπίσεις και αντιστροφές.
Συχνότητα μονονουκλεοτιδικών πολυμορφισμών στο ανθρώπινο γονιδίωμα[Επεξεργασία | επεξεργασία κώδικα]
Οι μονονουκλεοτιδικοί πολυμορφισμοί (SNPs) δεν εμφανίζονται ομοιόμορφα στο ανθρώπινο γονιδίωμα. Στην πραγματικότητα, υπάρχει τεράστια ποικιλία στη συχνότητα SNPs μεταξύ των γονιδίων, που αντανακλά τις διαφορετικές επιλεκτικές πιέσεις σε κάθε γονίδιο καθώς και διαφορετικούς ρυθμούς μεταλλάξεων και ανασυνδυασμών στο γονιδίωμα. Όμως, οι μελέτες στους SNPs επιλέγονται προς τις κωδικοποιητικές περιοχές, τα δεδομένα που δημιουργούνται από αυτές είναι απίθανο να ανακλούν τη συνολική κατανομή των SNPs σε όλο το γονιδίωμα. Γι' αυτό, το πρωτόκολλο SNP Consortium σχεδιάστηκε να ταυτοποιεί τα SNPs χωρίς μεροληψίες προς τις κωδικοποιητικές περιοχές και τα 100.000 SNPs ανακλούν γενικά ποικιλίες αλληλουχιών στα ανθρώπινα χρωμοσώματα του Consortium. Το SNP Consortium σκοπεύει να επεκτείνει τον αριθμό των SNPs που ταυτοποιούνται στο γονιδίωμα στις 300.000 μέχρι το τέλος του πρώτου τριμήνου του 2001.[70]
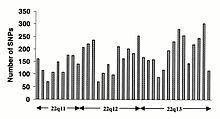
Αλλαγές στις μη κωδικοποιητικές αλληλουχίες και οι συνώνυμες αλλαγές στις κωδικοποιητικές αλληλουχίες είναι γενικά πιο κοινές από τις μη συνώνυμες αλλαγές, που ανακλά ότι η μεγαλύτερη επιλεκτική πίεση μειώνει την ποικιλία σε θέσεις που υπαγορεύουν ταυτότητα αμινοξέων. Οι μεταβατικές αλλαγές είναι πιο συνηθισμένες από τις μεταστροφές (transversions), με τα δινουκλεοτίδια CpG να εμφανίζουν τον υψηλότερο ρυθμό μεταλλάξεων, πιθανόν λόγω απαμίνωσης.
Προσωπικά γονιδιώματα[Επεξεργασία | επεξεργασία κώδικα]
Μια αλληλουχία προσωπικού γονιδιώματος είναι μια (σχεδόν) πλήρης αλληλουχία των χημικών ζευγών βάσεων που αποτελούν το DNA ενός μεμονωμένου ατόμου. Επειδή οι ιατρικές θεραπείες έχουν διαφορετικά αποτελέσματα σε διαφορετικά άτομα λόγω των γενετικών ποικιλιών όπως μονονουκλεοτιδικοί πολυμορφισμοί (SNPs), η ανάλυση των προσωπικών γονιδιωμάτων μπορεί να οδηγήσει σε εξατομικευμένη ιατρική θεραπεία με βάση τους ατομικούς γονότυπους.[71]
Η πρώτη αλληλουχία προσωπικού γονιδιώματος που καθορίστηκε ήταν αυτή του Craig Venter το 2007. Τα προσωπικά γονιδιώματα δεν έχουν ταξινομηθεί στο δημόσιο Human Genome Project για να προστατευτεί η ταυτότητα των εθελοντών που έδωσαν δείγματα DNA. Αυτή η αλληλουχία προέρχεται από το DNA πολλών εθελοντών από διαφορετικούς πληθυσμούς.[72] Όμως, νωρίς στην προσπάθεια εύρεσης της αλληλουχίας του γονιδιώματος από την Celera Genomics με επικεφαλής Venter ελήφθη η απόφαση για αλλαγή από την εύρεση της ακολουθίας ενός σύνθετου δείγματος σε ένα μεμονωμένο άτομο χρησιμοποιώντας το DNA του, που αργότερα αποκαλύφθηκε ότι ήταν του ίδιου του Venter. Συνεπώς, η αλληλουχία του ανθρώπινου γονιδιώματος της Celera που κυκλοφόρησε το 2000 ήταν κυρίως ενός ατόμου. Επόμενη αντικατάσταση των πρώιμων σύνθετων δεδομένων και καθορισμός της διπλοειδούς αλληλουχίας, που αναπαριστά και τα δύο σύνολα των χρωμοσωμάτων, αντί για την απλοειδή αλληλουχία που αναφέρθηκε αρχικά, επέτρεψε την κυκλοφορία του πρώτου προσωπικού γονιδιώματος.[73] Τον Απρίλιο του 2008, ολοκληρώθηκε επίσης το γονιδίωμα του James Watson. Από τότε εκατοντάδες αλληλουχίες προσωπικών γονιδιωμάτων έχου κυκλοφορήσει,[74] συμπεριλαμβανομένου του γονιδιώματος του Ντέσμοντ Τούτου,[75][76] και ενός Παλαιοεσκιμώου.[77] Τον Νοέμβριο του 2013, μια ισπανική οικογένεια ανακοίνωσε τα προσωπικά τους γονιδιώματα σύμφωνα με τη δημόσια άδεια των Creative Commons. Η εργασία καθοδηγήθηκε από τον Manuel Corpas και τα δεδομένα πάρθηκαν από απευθείας γενετικό έλεγχο με το 23andMe και το Beijing Genomics Institute). Αυτά πιστεύεται ότι αποτελούν τα πρώτα δημοσία γονιδιώματα οικογένειας.[78]
Η αλληλούχιση των ατομικών γονιδιωμάτων αποκάλυψε παραπέρα επίπεδα της γενετικής πολυπλοκότητας που δεν είχε εκτιμηθεί πριν. Τα προσωπικά γονιδιώματα βοήθησαν στην αποκάλυψη σημαντικών επιπέδων ποικιλίας στο ανθρώπινο γονιδίωμα που αποδίδεται όχι μόνο στα SNPs, αλλά επίσης σε δομικές ποικιλίες. Όμως, η εφαρμογή τέτοιας γνώσης στη θεραπεία ασθενειών είναι ακόμα στα πολύ αρχικά στάδια.[79] Η αλληλούχιση του εξονιώματος (Exome sequencing) έχει γίνει όλο και πιο δημοφιλής ως εργαλείο υποβοήθησης στη διάγνωση γενετικών ασθενειών, επειδή το εξονίωμα συνεισφέρει μόνο το 1% της γονιδιωματικής αλληλουχίας, αλλά είναι ο κύριος παράγοντας για περίπου το 85% των μεταλλάξεων που συνεισφέρουν σημαντικά στις ασθένειες.[80]
Ανθρώπινες αδρανοποιήσεις[Επεξεργασία | επεξεργασία κώδικα]
Στους ανθρώπους, οι αδρανοποιήσεις γονιδίων (gene knockouts) συμβαίνουν φυσικά ως ετεροζυγωτή ή ομοζυγωτή απώλεια λειτουργίας (loss-of-function) των αδρανοποιήσεων των γονιδίων. Αυτές τις αδρανοποιήσεις είναι συχνά δύσκολο να ξεχωριστούν, ειδικότερα στα ετερογενή γενετικά υπόβαθρα. Είναι επίσης δύσκολο να βρεθούν επειδή συμβαίνουν σε χαμηλές συχνότητες.
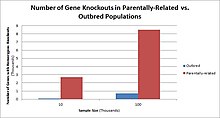
Πληθυσμοί με υψηλούς ρυθμούς συγγένειας εξ αίματος (consanguinity), όπως χώρες με υψηλούς ρυθμούς γάμων πρώτων εξαδέλφων, εμφανίζουν τις υψηλότερες συχνότητες των ομόζυγων αδρανοποιήσεων γονιδίων. Τέτοιοι πληθυσμοί περιλαμβάνουν το Πακιστάν, την Ισλανδία και πληθυσμούς Άμις. Αυτοί οι πληθυσμοί με υψηλό βαθμό γονικής συγγένειας είναι αντικείμενο έρευνας της ανθρώπινης αδρανοποίησης που έχει βοηθήσει στον καθορισμό της λειτουργίας των συγκεκριμένων γονιδίων στους ανθρώπους. Με τη διάκριση των ειδικών αδρανοποιήσεων, οι ερευνητές μπορούν να χρησιμοποιήσουν φαινοτυπικές αναλύσεις αυτών των ατόμων ώστε να βοηθηθεί ο χαρακτηρισμός των γονιδίων που έχουν αδρανοποιηθεί.
Οι αδρανοποιήσεις σε συγκεκριμένα γονίδια μπορούν να προκληθούν από γενετικές ασθένειες, αδρανοποιήσεις σε συγκεκριμένα γονίδια μπορεί να προκαλέσουν γενετικές διαταραχές και δυνητικά μπορεί να έχουν ευεργετικά αποτελέσματα, ή ακόμα να μην καταλήγουν καν σε φαινοτυπικό αποτέλεσμα. Όμως, ο καθορισμός του φαινοτυπικού αποτελέσματος μιας αδρανοποίησης στους ανθρώπους μπορεί να είναι μια πρόκληση. Οι προκλήσεις στον χαρακτηρισμό και την κλινική ερμηνεία των αδρανοποιήσεων περιλαμβάνουν δυσκολίες στην κλήση παραλλαγών DNA, που καθορίζουν τη διαταραχή της πρωτεϊνικής λειτουργίας και εξέτασης του βαθμού επηρεασμού του μωσαϊκισμού (mosaicism) που έχει στον φαινότυπο.[82]
Μια μεγάλη μελέτη που εξέτασε τις ανθρώπινες αδρανοποιήσεις είναι ο κίνδυνος μυοκαρδιακού εμφράγματος στο Πακιστάν. Βρέθηκε ότι άτομα που είχαν αδρανοποίηση του γονιδίου APOC3 λόγω ετερόζυγης απώλεια λειτουργίας είχαν χαμηλότερα τριγλυκερίδια στο αίμα μετά την κατανάλωση γεύματος υψηλών λιπαρών σε σύγκριση με τα άτομα χωρίς μετάλλαξη. Όμως, άτομα που είχαν ομόζυγη απώλεια λειτουργίας αδρανοποίησης γονιδίου του γονιδίου APOC3 εμφάνισαν το πιο χαμηλό επίπεδο τριγλυκεριδίων στο αίμα μετά τον έλεγχο φορτίου λιπαρών, επειδή δεν παράγουν τη λειτουργική πρωτεΐνη APOC3.[83]
Ανθρώπινες γενετικές διαταραχές[Επεξεργασία | επεξεργασία κώδικα]
Οι περισσότερες πλευρές της ανθρώπινης βιολογίας περιλαμβάνουν και γενετικούς (κληρονομημένους) και μη γενετικούς (περιβαλλοντικούς) παράγοντες. Κάποιες πλευρές επηρεασμού κληρονομημένων παραλλαγών της βιολογίας μας δεν είναι ιατρικής φύσης (ύψος, χρώμα ματιών, ικανότητα γεύσης ή οσμής συγκεκριμένων παραγόντων, κλπ. Επιπλέον, κάποιες γενετικές διαταραχές προκαλούν διαταραχές μόνο σε συνδυασμό με τους κατάλληλους περιβαλλοντικούς παράγοντες (όπως η δίαιτα). Με αυτούς τους περιορισμούς, οι γενετικές διαταραχές μπορούν να περιγραφούν ως κλινικά καθορισμένες ασθένειες που προκαλούνται από γονιδιωματικές ποικιλίες αλληλουχιών DNA. Στις περισσότερες απλές περιπτώσεις, η διαταραχή μπορεί να συσχετίζεται με παραλλαγή σε ένα μόνο γονίδιο. Παραδείγματος χάρη, η κυστική ίνωση προκαλείται από μεταλλάξεις στο γονίδιο CFTR και είναι η πιο κοινή υπολειπόμενη διαταραχή σε καυκασιανούς πληθυσμούς με πάνω από 1.300 γνωστές διαφορετικές μεταλλάξεις.[84]
Οι μεταλλάξεις που προκαλούν ασθένειες σε συγκεκριμένα γονίδια είναι συνήθως σοβαρές από πλευράς λειτουργίας γονιδίων -και είναι ευτυχώς σπάνιες- έτσι οι γενετικές διαταραχές είναι παρόμοιες παρομοίως ατομικά σπάνιες. Όμως, επειδή υπάρχουν πολλά γονίδια που μπορεί να ποικίλουν στην πρόκληση γενετικών διαταραχών, αποτελούν συνολικά ένα σημαντικό συστατικό των γνωστών ιατρικών συνθηκών, ιδιαίτερα στην παιδιατρική. Μοριακά χαρακτηρισμένες γενετικές διαταραχές είναι αυτές για τις οποίες το υποκείμενο αιτιώδες γονίδιο έχει ταυτοποιηθεί, μέχρι τώρα υπάρχουν περίπου 2.200 τέτοιες διαταραχές που αναφέρονται στη βάση δεδομένων OMIM.[84]
Μελέτες των γενετικών διαταραχών εκτελούνται συχνά μέσω μελετών με βάση οικογένειες. Σε κάποιες περιπτώσεις χρησιμοποιούνται προσεγγίσεις με βάση πληθυσμούς, ιδιαίτερα στην περίπτωση των λεγόμενων ιδρυτικών πληθυσμών όπως αυτών στη Φινλανδία, στον γαλλικό Καναδά, στη Γιούτα, στη Σαρδηνία κλπ. Η διάγνωση και η αντιμετώπιση των γενετικών διαταραχών εκτελούνται συνήθως από έναν γενετιστή-γιατρό εκπαιδευμένο στην κλινική/ιατρική γενετική. Τα αποτελέσματα του έργου του ανθρωπίνου γονιδιώματος είναι πιθανόν να παράσχουν αυξημένη διαθεσιμότητα των γενετικών ελέγχων για διαταραχές των γονιδίων και ενδεχομένως βελτιωμένη αντιμετώπιση. Οι γονείς μπορεί να εξεταστούν για κληρονομικές συνθήκες και να δοθούν οι αντίστοιχες συμβουλές για τις συνέπειες, την πιθανότητα να κληρονομηθούν και πώς να αποφευχθούν ή να βελτιωθούν στους απογόνους τους.
Όπως σημειώθηκε παραπάνω, υπάρχουν πολλά διαφορετικά είδη παραλλαγών της αλληλουχίας του DNA, που ποικίλει από πλήρη επιπλέον ή ελλείποντα χρωμοσώματα μέχρι μονονουκλεοτιδικές αλλαγές. Υποτίθεται, γενικά, ότι οι περισσότερες φυσικές γενετικές ποικιλίες στους ανθρώπινους πληθυσμούς είναι φαινοτυπικά ουδέτερες, δηλαδή έχουν μικρή ή καθόλου ανιχνεύσιμη επίπτωση στη φυσιολογία του ατόμου (αν και μπορεί να υπάρχουν αποσπασματικές διαφορές στη προσαρμοστικότητα που καθορίζεται από τα εξελικτικά χρονικά πλαίσια). Οι γενετικές διαταραχές μπορεί να προκληθούν από οποιονδήποτε ή από όλους τους τύπους παραλλαγών αλληλουχιών. Για να χαρακτηριστεί μοριακά μια νέα γενετική διαταραχή, είναι απαραίτητο να καθοριστεί ένας αιτιώδης σύνδεσμος μεταξύ μιας συγκεκριμένης γονιδιωματικής παραλλαγής αλληλουχίας και της κλινικής υπό εξέταση ασθένειας. Τέτοιες μελέτες συνιστούν το πεδίο της ανθρώπινης μοριακής γενετικής.
Με την έλευση του ανθρωπίνου γονιδιώματος και του International HapMap Project, έγινε εφικτή η εξερεύνηση ανεπαίσθητων γενετικών επιδράσεων σε πολλές κοινές συνθήκες ασθενειών, όπως ο διαβήτης, το άσθμα, η ημικρανία, η σχιζοφρένεια κλπ. Αν και κάποιοι αιτιώδεις συνδέσεις έχουν γίνει μεταξύ γονιδιωματικών παραλλαγών αλληλουχιών σε συγκεκριμένα γονίδια και κάποιων από αυτές στις ασθένειες, αν και γενικά με μεγάλη δημοσιότητα στα μέσα, αυτές δεν θεωρούνται συνήθως ότι είναι γενετικές διαταραχές αυτές καθεαυτές επειδή τα αίτια τους είναι περίπλοκα και εμπλέκουν πολλούς γενετικούς και περιβαλλοντικούς παράγοντες. Υπάρχουν, συνεπώς, πολλές διαφωνίες σε συγκεκριμένες περιπτώσεις εάν οι συγκεκριμένες ιατρικές συνθήκες θα πρέπει να οριστούν ως γενετικές διαταραχές. Ο παρακάτω κατηγοριοποιημένος πίνακας παρέχει τον επιπολασμό (συχνότητα) καθώς και τα γονίδια ή τα χρωμοσώματα που σχετίζονται με κάποιες ανθρώπινες γενετικές διαταραχές.
| Διαταραχή | Συχνότητα | Εμπλεκόμενα χρωμοσώματα ή γονίδια |
|---|---|---|
| Χρωμοσωμικές συνθήκες | ||
| Σύνδρομο Down | 1:600 | Χρωμόσωμα 21 |
| Σύνδρομο Κλαϊνεφέλτερ | 1:500–1000 άνδρες | Πρόσθετο χρωμόσωμα Χ |
| Σύνδρομο Τέρνερ | 1:2000 γυναίκες | Απώλεια του χρωμοσώματος Χ |
| Δρεπανοκυτταρική αναιμία | 1 στις 50 γεννήσεις σε περιοχές της Αφρικής· πιο σπάνια αλλού[85] | β-σφαιρίνη (στο χρωμόσωμα 11) |
| Καρκίνοι | ||
| Καρκίνος του μαστού/Καρκίνος των ωοθηκών (ευπάθεια) | ~5% των περιπτώσεων αυτών των τύπων καρκίνου | BRCA1, BRCA2 |
| Οικογενής αδενοματώδης πολυποδίαση (FAP)] (κληρονομική μη πολυποδίαση κόλου) | 1:3500 | Αδενοματώδης πολυποδίαση κόλου (APC) |
| Σύνδρομο Lynch | 5–10% όλων των περιπτώσεων καρκίνου του εντέρου | MLH1, MSH2, MSH6, PMS2 |
| Νευρολογικές συνθήκες | ||
| Νόσος του Χάντινγκτον | 1:20000 | Χάντινγκτον |
| Νόσος Αλτσχάιμερ ‐ πρώιμη έναρξη | 1:2500 | PS1, PS2, APP |
| Άλλες συνθήκες | ||
| Κυστική ίνωση | 1:2500 | CFTR |
| Μυική δυστροφία Duchenne | 1:3500 αγόρια | Δυστροφίνη |
Εξέλιξη[Επεξεργασία | επεξεργασία κώδικα]
Οι μελέτες της συγκριτικής γονιδιωματικής (Comparative genomics) των γονιδιωμάτων των θηλαστικών προτείνουν ότι περίπου το 5% του ανθρώπινου γονιδιώματος έχει διατηρηθεί από την εξέλιξη από τον διαχωρισμό των υφιστάμενων γενεαλογιών περίπου 200 εκατομμύρια χρόνια πριν, που περιέχει τη μεγάλη πλειοψηφία των γονιδίων.[86][87] Το δημοσιευμένο γονιδίωμα του χιμπαντζή του κοινού διαφέρει από αυτό του ανθρώπινου γονιδιώματος κατά 1,23% σε συγκρίσεις ευθείας αλληλουχίας.[88] Περίπου 20% αυτού του αριθμού υπολογίζεται από τις ποικιλίες μέσα σε κάθε είδος, αφήνοντας μόνο ~1,06% απόκλιση ομοιόμορφης αλληλουχίας μεταξύ ανθρώπων και χιμπαντζήδων στα κοινά γονίδια.[89] Αυτή η διαφορά νουκλεοτιδίου προς νουκλεοτίδιο φαίνεται μικροσκοπική, όμως, από το τμήμα του κάθε γονιδιώματος που δεν είναι κοινό, συμπεριλαμβάνοντας περίπου το 6% των λειτουργικών γονιδίων που είναι μοναδικά είτε στον άνθρωπο είτε στους χιμπαντζήδες.[90]
Με άλλα λόγια, οι αξιοσημείωτες παρατηρούμενες διαφορές μεταξύ ανθρώπων και χιμπατζήδων μπορεί να οφείλονται εξίσου ή περισσότερο στην ποικιλία επιπέδου γονιδιώματος σε αριθμό, λειτουργία και έκφραση των γονιδίων παρά στις αλλαγές αλληλουχιών του DNA σε κοινά γονίδια. Πράγματι, ακόμα και ανάμεσα στους ανθρώπους, έχει βρεθεί να υπάρχει μια προηγουμένως μη εκτιμώμενη ποσότητα ποικιλότητας του αριθμού των αντιγράφων (copy number variation ή CNV) που αποτελεί μέχρι το 5 – 15% του ανθρωπίνου γονιδιώματος. Με άλλα λόγια, μεταξύ των ανθρώπων, μπορεί να υπάρχουν +/- 500.000.000 ζεύγη βάσεων DNA, που κάποια είναι ενεργά γονίδια, άλλα απενεργοποιημένα, ή ενεργά σε διαφορετικά επίπεδα. Η πλήρης σημασία αυτής της ανακάλυψης παραμένει να βρεθεί. Κατά μέσο όρο, ένα τυπικό ανθρώπινο γονίδιο κωδικοποιητικό πρωτεΐνης διαφέρει από το ορθόλογο του χιμπαντζή κατά μόνο δύο υποκαταστάσεις αμινοξέων· σχεδόν το ένα τρίτο των ανθρώπινων γονιδίων έχει ακριβώς την ίδια πρωτεϊνική μετάφραση όπως τα ορθόλογα του χιμπαντζή. Μια σημαντική διαφορά μεταξύ των δύο γονιδιωμάτων είναι το ανθρώπινο χρωμόσωμα 2, που είναι ισοδύναμο με το προϊόν συγχώνευσης των χρωμοσωμάτων 12 και 13 του χιμπαντζή.[91] (αργότερα μετονομάστηκαν σε χρωμοσώματα 2A και 2B, αντίστοιχα).
Οι άνθρωποι έχουν υποστεί μια αξιοσημείωτη απώλεια των γονιδίων του οσφρητικού υποδοχέα κατά τη διάρκεια της πρόσφατης μας εξέλιξης, που εξηγεί τη σχετικά χοντροκομμένη αίσθηση της όσφρησης συγκρινόμενη με τα περισσότερα άλλα θηλαστικά. Τα εξελικτικά στοιχεία προτείνουν ότι η εμφάνιση της έγχρωμης όρασης στους ανθρώπους και σε αρκετά άλλα πρωτεύοντα είδη έχει μειώσει την ανάγκη της όσφρησης.[92]
Τον Σεπτέμβριο του 2016, αναφέρθηκε ότι με βάση τις ανθρώπινες γενετικές μελέτες του DNA, όλοι οι μη Αφρικανοί στον κόσμο σήμερα μπορεί να εντοπιστούν σε έναν μόνο πληθυσμό που έφυγε από την Αφρική μεταξύ 50.000 και 80.000 χρόνια πριν.[93]
Μιτοχονδριακό DNA[Επεξεργασία | επεξεργασία κώδικα]
Το ανθρώπινο μιτοχονδριακό DNA είναι τεράστιου ενδιαφέροντος για τους γενετιστές, επειδή παίζει χωρίς αμφιβολία ρόλο στις μιτοχονδριακές ασθένειες. Ρίχνει επίσης φως στην ανθρώπινη εξέλιξη· παραδείγματος χάρη, η ανάλυση της ποικιλομορφίας στο ανθρώπινο γονιδιακό γονιδίωμα έχει οδηγήσει στον ισχυρισμό ενός πρόσφατου κοινού προγόνου για όλους τους ανθρώπους στη μητρική γραμμή καταγωγής.
Λόγω της έλλειψης ενός συστήματος ελέγχου αντιγραφής σφαλμάτων, το μιτοχονδριακό DNA (mtDNA) έχει πιο γρήγορο ρυθμό ποικιλομορφίας από το πυρηνικό DNA. Αυτός ο 20πλάσιος υψηλότερος ρυθμός μεταλλάξεων επιτρέπει στο mtDNA να χρησιμοποιηθεί για πιο ακριβή εντοπισμό της μητρικής προγονικότητας. Μελέτες του mtDNA σε πληθυσμούς έχουν επιτρέψει τον εντοπισμό των αρχαίων διαδρομών μετανάστευσης, όπως η μετανάστευση των Ιθαγενών Αμερικανών από τη Σιβηρία ή των Πολυνησίων από τη νοτιοανατολική Ασία. Έχει επίσης χρησιμοποιηθεί για να δείξει ότι δεν υπάρχει εντοπισμός DNA Νεάντερταλ στο ευρωπαϊκό κληρονομημένο μείγμα γονιδίων μέσω καθαρά μητρικής γραμμής.[94]
Επιγονιδίωμα[Επεξεργασία | επεξεργασία κώδικα]
Η επιγενετική περιγράφει μια ποικιλία χαρακτηριστικών του ανθρώπινου γονιδιώματος που υπερβαίνει την πρωτογενή αλληλουχία του DNA, τέτοια όπως η συσκευασία χρωματίνης, οι τροποποιήσεις της ιστόνης και η μεθυλίωση του DNA που είναι σημαντικές στη ρύθμιση της έκφρασης των γονιδίων, στην αντιγραφή του γονιδιώματος και σε άλλες κυτταρικές διεργασίες. Επιγενετικοί δείκτες ενισχύουν και εξασθενούν τη μεταγραφή συγκεκριμένων γονιδίων, αλλά δεν επηρεάζουν την ενεργή αλληλουχία των νουκλεοτιδίων του DNA. Η μεθυλίωση του DNA είναι μια σημαντική μορφή επιγενετικού ελέγχου στην έκφραση των γονιδίων και ένα από τις καλύτερα μελετημένα θέματα στην επιγενετική. Κατά την ανάπτυξη, η κατατομή (προφίλ) μεθυλίωσης του ανθρώπινου DNA υφίσταται δραματικές αλλαγές. Στα πρώιμα κύτταρα της γεννητικής γραμμής, το γονιδίωμα έχει πολύ χαμηλά επίπεδα μεθυλίωσης. Αυτά τα χαμηλά επίπεδα περιγράφουν γενικά ενεργά γονίδια. Καθώς προχωρά η ανάπτυξη, οι γονικές ετικέτες αποτύπωσης οδηγούν σε αυξημένη δραστηριότητα μεθυλίωσης.[95][96]
Τα επιγενετικά πρότυπα μπορούν να ταυτοποιηθούν μεταξύ ιστών είτε σε ένα άτομο, είτε μεταξύ ατόμων. Ταυτόσημα γονίδια που έχουν διαφορές μόνο στην επιγενετική τους κατάσταση ονομάζονται επιαλληλόμορφα (epialleles). Τα επιαλληλόμορφα μπορούν να τοποθετηθούν σε τρεις κατηγορίες: αυτά που καθορίζονται άμεσα από ένα ατομικό γενότυπο, αυτά που επηρεάζονται από τον γενότυπο και αυτά που είναι ολότελα ανεξάρτητα από τον γενότυπο. Το επιγονιδίωμα επηρεάζεται επίσης σημαντικά από περιβαλλοντικούς παράγοντες. Δίαιτα, τοξίνες και ορμόνες επηρεάζουν την επιγενετική κατάσταση. Μελέτες στον διαιτολογικό χειρισμό έχουν δείξει ότι δίαιτες με έλλειμμα μεθυλίου συσχετίζονται με υπομεθυλίωση του επιγονιδιώματος. Τέτοιες μελέτες καθιστούν την επιγενετική συνδετικό κρίκο μεταξύ του περιβάλλοντος και του γονιδιώματος.[97]
Παραπομπές[Επεξεργασία | επεξεργασία κώδικα]
- ↑ Brown, Terence A. (2002). «The Human Genome» (στα Αγγλικά). Wiley-Liss.
- ↑ «An integrated map of genetic variation from 1,092 human genomes». Nature 491 (7422): 56–65. Nov 2012. doi:. PMID 23128226. Bibcode: 2012Natur.491...56T.
- ↑ «Comparing the human and chimpanzee genomes: searching for needles in a haystack». Genome Research 15 (12): 1746–58. Dec 2005. doi:. PMID 16339373.
- ↑ International Human Genome Sequencing Consortium Publishes Sequence and Analysis of the Human Genome
- ↑ Pennisi, Elizabeth (2001-02-16). «The Human Genome». Science (AAAS). http://science.sciencemag.org/content/291/5507/1177. Ανακτήθηκε στις 2019-02-03.
- ↑ 6,0 6,1 International Human Genome Sequencing Consortium (Oct 2004). «Finishing the euchromatic sequence of the human genome». Nature 431 (7011): 931–45. doi:. PMID 15496913. Bibcode: 2004Natur.431..931H. http://www.nature.com/nature/journal/v431/n7011/full/nature03001.html.
- ↑ «Multiple evidence strands suggest that there may be as few as 19,000 human protein-coding genes». Human Molecular Genetics 23 (22): 5866–78. November 2014. doi:. PMID 24939910.
- ↑ 8,0 8,1 8,2 «Genomics. ENCODE project writes eulogy for junk DNA». Science 337 (6099): 1159–1161. Sep 2012. doi:. PMID 22955811.
- ↑ 9,0 9,1 9,2 International Human Genome Sequencing Consortium (Feb 2001). «Initial sequencing and analysis of the human genome». Nature 409 (6822): 860–921. doi:. PMID 11237011. Bibcode: 2001Natur.409..860L.
- ↑ Pollack, Andrew (2 June 2016). «Scientists Announce HGP-Write, Project to Synthesize the Human Genome». New York Times. https://www.nytimes.com/2016/06/03/science/human-genome-project-write-synthetic-dna.html. Ανακτήθηκε στις 2 June 2016.
- ↑ «The Genome Project-Write». Science 353 (6295): 126–7. July 2016. doi:. PMID 27256881. Bibcode: 2016Sci...353..126B.
- ↑ Piovesan, A; Pelleri, MC; Antonaros, F; Strippoli, P; Caracausi, M; Vitale, L (2019). «On the length, weight and GC content of the human genome». BMC Research notes 12 (1): 106. doi:. PMID 30813969.
- ↑ «Resolving the complexity of the human genome using single-molecule sequencing». Nature 517 (7536): 608–11. Jan 2015. doi:. PMID 25383537. Bibcode: 2015Natur.517..608C.
- ↑ Pratas, D., Pinho, A. J., and Ferreira, P. J. S. G. Efficient compression of genomic sequences. Data Compression Conference, Snowbird, Utah, 2016.
- ↑ «Human Genome Project Completion: Frequently Asked Questions». National Human Genome Research Institute (NHGRI) (στα Αγγλικά). Ανακτήθηκε στις 2 Φεβρουαρίου 2019.
- ↑ «Human genomes as email attachments». Bioinformatics 25 (2): 274–5. January 2009. doi:. PMID 18996942.
- ↑ Zhandong Liu, Santosh S Venkatesh and Carlo C Maley, Sequence space coverage, entropy of genomes and the potential to detect non-human DNA in human samples, BMC Genomics 2008, 9:509, [1] doi:10.1186/1471-2164-9-509, fig. 6, using the Lempel-Ziv estimators of entropy rate.
- ↑ Waters K (7 Μαρτίου 2007). «Molecular Genetics». Stanford Encyclopedia of Philosophy. Ανακτήθηκε στις 18 Ιουλίου 2013.
- ↑ Gannett L (26 Οκτωβρίου 2008). «The Human Genome Project». Stanford Encyclopedia of Philosophy. Ανακτήθηκε στις 18 Ιουλίου 2013.
- ↑ PANTHER Pie Chart at the PANTHER Classification System homepage. Retrieved May 25, 2011
- ↑ List of human proteins in the Uniprot Human reference proteome; accessed 28 Jan 2015
- ↑ «Metabolic stability and epigenesis in randomly constructed genetic nets». Journal of Theoretical Biology 22 (3): 437–67. Mar 1969. doi:. PMID 5803332. http://www.sciencedirect.com/science/article/pii/0022519369900150.
- ↑ Ohno, S. ς (1972). «An argument for the genetic simplicity of man and other mammals». Journal of Human Evolution 1 (6): 651–662. doi:.
- ↑ «Relationship between gene expression and GC-content in mammals: statistical significance and biological relevance». Human Molecular Genetics 14 (3): 421–7. February 2005. doi:. PMID 15590696.
- ↑ M. Huang, H. Zhu, B. Shen, G. Gao, "A non-random gait through the human genome", 3rd International Conference on Bioinformatics and Biomedical Engineering (UCBBE, 2009), 1–3
- ↑ 26,0 26,1 26,2 «GeneBase 1.1: a tool to summarize data from NCBI gene datasets and its application to an update of human gene statistics». Database : The Journal of Biological Databases and Curation 2016: baw153 . 2016. doi:. PMID 28025344.
- ↑ «The complete gene sequence of titin, expression of an unusual approximately 700-kDa titin isoform, and its interaction with obscurin identify a novel Z-line to I-band linking system». Circulation Research 89 (11): 1065–72. 2001. doi:. PMID 11717165.
- ↑ «Synergy between sequence and size in large-scale genomics». Nature Reviews Genetics 6 (9): 699–708. Sep 2005. doi:. PMID 16151375.
- ↑ 29,0 29,1 «Nuclear export as a key arbiter of "mRNA identity" in eukaryotes». Biochimica et Biophysica Acta 1819 (6): 566–77. Jun 2012. doi:. PMID 22248619.
- ↑ «Functional evolution of noncoding DNA». Current Opinion in Genetics & Development 12 (6): 634–9. Dec 2002. doi:. PMID 12433575. http://linkinghub.elsevier.com/retrieve/pii/S0959437X02003556.
- ↑ «Intergenic transcription is required to repress the Saccharomyces cerevisiae SER3 gene». Nature 429 (6991): 571–4. Jun 2004. doi:. PMID 15175754. Bibcode: 2004Natur.429..571M.
- ↑ «Long noncoding RNA as modular scaffold of histone modification complexes». Science 329 (5992): 689–93. Aug 2010. doi:. PMID 20616235. Bibcode: 2010Sci...329..689T.
- ↑ «Parental imprinting of the mouse H19 gene». Nature 351 (6322): 153–5. May 1991. doi:. PMID 1709450. Bibcode: 1991Natur.351..153B.
- ↑ «Recombination regulation by transcription-induced cohesin dissociation in rDNA repeats». Science 309 (5740): 1581–4. Sep 2005. doi:. PMID 16141077. Bibcode: 2005Sci...309.1581K.
- ↑ «A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?». Cell 146 (3): 353–8. Aug 2011. doi:. PMID 21802130.
- ↑ «The GENCODE pseudogene resource». Genome Biology 13 (9): R51. 2012. doi:. PMID 22951037.
- ↑ «Human specific loss of olfactory receptor genes». Proceedings of the National Academy of Sciences of the United States of America 100 (6): 3324–7. Mar 2003. doi:. PMID 12612342. Bibcode: 2003PNAS..100.3324G.
- ↑ «The landscape of long noncoding RNAs in the human transcriptome». Nature Genetics 47 (3): 199–208. Mar 2015. doi:. PMID 25599403.
- ↑ «Non-coding RNA genes and the modern RNA world». Nature Reviews Genetics 2 (12): 919–29. Dec 2001. doi:. PMID 11733745. http://www.nature.com/nrg/journal/v2/n12/full/nrg1201-919a.html.
- ↑ «The vast, conserved mammalian lincRNome». PLoS Computational Biology 9 (2): e1002917. 2013. doi:. PMID 23468607. Bibcode: 2013PLSCB...9E2917M.
- ↑ «Non-coding RNA: what is functional and what is junk?». Frontiers in Genetics 6: 2. 2015. doi:. PMID 25674102.
- ↑ «Non-coding RNA». Human Molecular Genetics 15 Spec No 1: R17–29. Apr 2006. doi:. PMID 16651366.
- ↑ 43,0 43,1 «An integrated encyclopedia of DNA elements in the human genome». Nature 489 (7414): 57–74. Sep 2012. doi:. PMID 22955616. Bibcode: 2012Natur.489...57T.
- ↑ Birney E (5 Σεπτεμβρίου 2012). «ENCODE: My own thoughts». Ewan's Blog: Bioinformatician at large.
- ↑ «What does our genome encode?». Genome Research 22 (9): 1602–11. Sep 2012. doi:. PMID 22955972.
- ↑ «Regulating Evolution». Scientific American: 60–67. May 2008. http://www.scientificamerican.com/article/regulating-evolution/.
- ↑ «The promoter-operator region of the lac operon of Escherichia coli». J. Mol. Biol. 38 (3): 413–20. 1968. doi:. PMID 4887877.
- ↑ «DNA sequences required for regulated expression of beta-globin genes in murine erythroleukemia cells». Cell 38 (1): 265–73. 1984. doi:. PMID 6088069.
- ↑ «Estimation of divergence times from multiprotein sequences for a few mammalian species and several distantly related organisms». Proceedings of the National Academy of Sciences of the United States of America 98 (5): 2497–502. Feb 2001. doi:. PMID 11226267. PMC 30166. Bibcode: 2001PNAS...98.2497N. http://www.pnas.org/cgi/content/full/051611498.
- ↑ «Identification of a coordinate regulator of interleukins 4, 13, and 5 by cross-species sequence comparisons». Science 288 (5463): 136–40. Apr 2000. doi:. PMID 10753117. Bibcode: 2000Sci...288..136L. Summary Αρχειοθετήθηκε 2009-11-06 στο Wayback Machine.
- ↑ Meunier M. «Genoscope and Whitehead announce a high sequence coverage of the Tetraodon nigroviridis genome». Genoscope. Αρχειοθετήθηκε από το πρωτότυπο στις 16 Οκτωβρίου 2006. Ανακτήθηκε στις 12 Σεπτεμβρίου 2006.
- ↑ «Comparative studies of gene expression and the evolution of gene regulation». Nature Reviews Genetics 13 (7): 505–16. Jul 2012. doi:. PMID 22705669.
- ↑ «Five-vertebrate ChIP-seq reveals the evolutionary dynamics of transcription factor binding». Science 328 (5981): 1036–40. May 2010. doi:. PMID 20378774. Bibcode: 2010Sci...328.1036S.
- ↑ «Species-specific transcription in mice carrying human chromosome 21». Science 322 (5900): 434–8. Oct 2008. doi:. PMID 18787134. Bibcode: 2008Sci...322..434W.
- ↑ «Repetitive DNA and next-generation sequencing: computational challenges and solutions». Nature Reviews Genetics 13 (1): 36–46. Jan 2012. doi:. PMID 22124482.
- ↑ «Large-scale analysis of tandem repeat variability in the human genome». Nucleic Acids Research 42 (9): 5728–41. May 2014. doi:. PMID 24682812.
- ↑ Pierce BA (2012). Genetics : a conceptual approach (4th έκδοση). New York: W.H. Freeman. σελίδες 538–540. ISBN 978-1-4292-3250-0.
- ↑ «Active Alu retrotransposons in the human genome». Genome Research 18 (12): 1875–83. Dec 2008. doi:. PMID 18836035.
- ↑ «A gene expression restriction network mediated by sense and antisense Alu sequences located on protein-coding messenger RNAs». BMC Genomics 14: 325. 2013. doi:. PMID 23663499. PMC 3655826. http://www.biomedcentral.com/1471-2164/14/325.
- ↑ «Hot L1s account for the bulk of retrotransposition in the human population». Proceedings of the National Academy of Sciences of the United States of America 100 (9): 5280–5. Apr 2003. doi:. PMID 12682288. Bibcode: 2003PNAS..100.5280B.
- ↑ Barton NH, Briggs DE, Eisen JA, Goldstein DB, Patel NH (2007). Evolution. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-684-9.
- ↑ NCBI. «GRCh38 - hg38 - Genome - Assembly - NCBI». ncbi.nlm.nih.gov. Ανακτήθηκε στις 15 Μαρτίου 2019.
- ↑ «from Bill Clinton's 2000 State of the Union address». Αρχειοθετήθηκε από το πρωτότυπο στις 21 Φεβρουαρίου 2017. Ανακτήθηκε στις 20 Ιουνίου 2019.
- ↑ Nature (2006). «Global variation in copy number in the human genome : Article : Nature». Nature 444 (7118): 444–454. doi:. PMID 17122850. PMC 2669898. Bibcode: 2006Natur.444..444R. http://www.nature.com/nature/journal/v444/n7118/full/nature05329.html. Ανακτήθηκε στις 2009-08-09.
- ↑ «What's a Genome?». Genomenewsnetwork.org. 15 Ιανουαρίου 2003. Ανακτήθηκε στις 31 Μαΐου 2009.
- ↑ NCBI_user_services (29 Μαρτίου 2004). «Mapping Factsheet». Ncbi.nlm.nih.gov. Αρχειοθετήθηκε από το πρωτότυπο στις 19 Ιουλίου 2010. Ανακτήθηκε στις 31 Μαΐου 2009.
- ↑ «About the Project». HapMap. Αρχειοθετήθηκε από το πρωτότυπο στις 15 Μαΐου 2008. Ανακτήθηκε στις 31 Μαΐου 2009.
- ↑ «2008 Release: Researchers Produce First Sequence Map of Large-Scale Structural Variation in the Human Genome». genome.gov. Ανακτήθηκε στις 31 Μαΐου 2009.
- ↑ «Mapping and sequencing of structural variation from eight human genomes». Nature 453 (7191): 56–64. May 2008. doi:. PMID 18451855. Bibcode: 2008Natur.453...56K.
- ↑ «Single nucleotide polymorphisms as tools in human genetics». Hum. Mol. Genet. 9 (16): 2403–8. 2000. doi:. PMID 11005795.
- ↑ Lai, Eric (2001-06-01). «Application of SNP Technologies in Medicine: Lessons Learned and Future Challenges» (στα αγγλικά). Genome Research 11 (6): 927–929. doi:. ISSN 1088-9051. PMID 11381021. http://genome.cshlp.org/content/11/6/927.
- ↑ «Human Genome Project Completion: Frequently Asked Questions». genome.gov. Ανακτήθηκε στις 31 Μαΐου 2009.
- ↑ Singer, Emily (September 4, 2007). «Technology Review». Technology review. http://www.technologyreview.com/biomedicine/19328/?a=f. Ανακτήθηκε στις May 25, 2010.
- ↑ «Complete Genomics Adds 29 High-Coverage, Complete Human Genome Sequencing Datasets to Its Public Genomic Repository».
- ↑ Ian Sample (17 Φεβρουαρίου 2010). «Desmond Tutu's genome sequenced as part of genetic diversity study». The Guardian.
- ↑ «Complete Khoisan and Bantu genomes from southern Africa». Nature 463 (7283): 943–7. 2010. doi:. PMID 20164927. Bibcode: 2010Natur.463..943S.
- ↑ «Ancient human genome sequence of an extinct Palaeo-Eskimo». Nature 463 (7282): 757–62. Feb 2010. doi:. PMID 20148029. Bibcode: 2010Natur.463..757R.
- ↑ Corpas M, Cariaso M, Coletta A, Weiss D, Harrison AP, Moran F, Yang H (12 Νοεμβρίου 2013). «A Complete Public Domain Family Genomics Dataset τη». bioRxiv 000216
 .
.
- ↑ «Human genome sequencing in health and disease». Annual Review of Medicine 63: 35–61. 2012. doi:. PMID 22248320.
- ↑ «Genetic diagnosis by whole exome capture and massively parallel DNA sequencing». Proceedings of the National Academy of Sciences of the United States of America 106 (45): 19096–101. Nov 2009. doi:. PMID 19861545. Bibcode: 2009PNAS..10619096C.
- ↑ «Human Knockout Carriers: Dead, Diseased, Healthy, or Improved?». Trends in Molecular Medicine 22 (4): 341–351. April 2016. doi:. PMID 26988438.
- ↑ «Human Knockout Carriers: Dead, Diseased, Healthy, or Improved?». Trends in Molecular Medicine 22 (4): 341–351. April 2016. doi:. PMID 26988438.
- ↑ «Human knockouts and phenotypic analysis in a cohort with a high rate of consanguinity». Nature 544 (7649): 235–239. April 2017. doi:. PMID 28406212. Bibcode: 2017Natur.544..235S.
- ↑ 84,0 84,1 Online Mendelian Inheritance in Man (OMIM)
- ↑ «Sickle-cell anaemia – Report by the Secretariat» (pdf). Fifty-ninth World Health Assembly. World Health Organization. 24 Απριλίου 2006.
- ↑ «Initial sequencing and comparative analysis of the mouse genome». Nature 420 (6915): 520–62. Dec 2002. doi:. PMID 12466850. Bibcode: 2002Natur.420..520W. «the proportion of small (50–100 bp) segments in the mammalian genome that is under (purifying) selection can be estimated to be about 5%. This proportion is much higher than can be explained by protein-coding sequences alone, implying that the genome contains many additional features (such as untranslated regions, regulatory elements, non-protein-coding genes, and chromosomal structural elements) under selection for biological function.».
- ↑ «Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project». Nature 447 (7146): 799–816. Jun 2007. doi:. PMID 17571346. Bibcode: 2007Natur.447..799B.
- ↑ The Chimpanzee Sequencing; Analysis Consortium (Sep 2005). «Initial sequence of the chimpanzee genome and comparison with the human genome». Nature 437 (7055): 69–87. doi:. PMID 16136131. Bibcode: 2005Natur.437...69.. http://www.nature.com/nature/journal/v437/n7055/pdf/nature04072.pdf. «We calculate the genome-wide nucleotide divergence between human and chimpanzee to be 1.23%, confirming recent results from more limited studies.».
- ↑ The Chimpanzee Sequencing; Analysis Consortium (Sep 2005). «Initial sequence of the chimpanzee genome and comparison with the human genome». Nature 437 (7055): 69–87. doi:. PMID 16136131. Bibcode: 2005Natur.437...69.. «we estimate that polymorphism accounts for 14–22% of the observed divergence rate and thus that the fixed divergence is ~1.06% or less».
- ↑ «The evolution of mammalian gene families». PLOS ONE 1 (1): e85. 2006. doi:. PMID 17183716. Bibcode: 2006PLoSO...1...85D. «Our results imply that humans and chimpanzees differ by at least 6% (1,418 of 22,000 genes) in their complement of genes, which stands in stark contrast to the oft-cited 1.5% difference between orthologous nucleotide sequences».
- ↑ The Chimpanzee Sequencing; Analysis Consortium (Sep 2005). «Initial sequence of the chimpanzee genome and comparison with the human genome». Nature 437 (7055): 69–87. doi:. PMID 16136131. Bibcode: 2005Natur.437...69.. «Human chromosome 2 resulted from a fusion of two ancestral chromosomes that remained separate in the chimpanzee lineage».
«Sequencing the chimpanzee genome: insights into human evolution and disease». Nature Reviews Genetics 4 (1): 20–8. Jan 2003. doi:. PMID 12509750. «Large-scale sequencing of the chimpanzee genome is now imminent.». - ↑ «Loss of olfactory receptor genes coincides with the acquisition of full trichromatic vision in primates». PLoS Biology 2 (1): E5. Jan 2004. doi:. PMID 14737185. «Our findings suggest that the deterioration of the olfactory repertoire occurred concomitant with the acquisition of full trichromatic color vision in primates.».
- ↑ Zimmer, Carl (September 21, 2016). «How We Got Here: DNA Points to a Single Migration From Africa». New York Times. https://www.nytimes.com/2016/09/22/science/ancient-dna-human-history.html. Ανακτήθηκε στις September 22, 2016.
- ↑ Sykes, Bryan (9 Οκτωβρίου 2003). «Mitochondrial DNA and human history». The Human Genome. Αρχειοθετήθηκε από το πρωτότυπο στις 7 Σεπτεμβρίου 2015. Ανακτήθηκε στις 19 Σεπτεμβρίου 2006.
- ↑ «Beyond the sequence: cellular organization of genome function». Cell 128 (4): 787–800. Feb 2007. doi:. PMID 17320514.
- ↑ «The mammalian epigenome». Cell 128 (4): 669–81. Feb 2007. doi:. PMID 17320505.
- ↑ «[Epigenetics, interface between environment and genes: role in complex diseases]». Revue Médicale de Liège 67 (5–6): 250–7. May–Jun 2012. PMID 22891475.
Εξωτερικοί σύνδεσμοι[Επεξεργασία | επεξεργασία κώδικα]
- Ensembl The Ensembl Genome Browser Project
- National Library of Medicine human genome viewer
- UCSC Genome Browser.
- Human Genome Project.

