Αλληλουχία νουκλεϊκών οξέων
Μια αλληλουχία νουκλεϊκών οξέων είναι μια διαδοχή βάσεων που υποδηλώνεται από μια σειρά από πέντε διαφορετικά γράμματα (G, A, C, T και U) που υποδεικνύουν τη σειρά των νουκλεοτιδίων που σχηματίζουν ένα μόριο DNA (χρησιμοποιώντας τα G, A, C και T) ή ένα μόριο RNA (χρησιμοποιώντας τα G, A, C και U). Συνήθως, οι αλληλουχίες παρουσιάζονται συνήθως από το 5' άκρο στο 3' άκρο. Για το DNA, χρησιμοποιείται η κωδική αλυσίδα. Επειδή τα νουκλεϊκά οξέα είναι συνήθως γραμμικά (μη διακλαδισμένα) πολυμερή, ο καθορισμός της αλληλουχίας ισοδυναμεί με τον ορισμό της ομοιοπολικής δομής ολόκληρου του μορίου. Για το λόγο αυτό, η αλληλουχία νουκλεϊκών οξέων ονομάζεται επίσης πρωτογενής δομή.
Η ακολουθία έχει την ικανότητα να αντιπροσωπεύει πληροφορίες. Το βιολογικό δεοξυριβονουκλεϊκό οξύ αντιπροσωπεύει την πληροφορία που κατευθύνει τις λειτουργίες ενός ζωντανού όντος.
Τα νουκλεϊκά οξέα έχουν επίσης δευτεροταγή και τριτοταγή δομή. Η πρωτοταγής δομή αναφέρεται μερικές φορές κατά λάθος ως πρωτοταγής ακολουθία.
Νουκλεοτίδια
[Επεξεργασία | επεξεργασία κώδικα]

Τα νουκλεϊκά οξέα αποτελούνται από μια αλυσίδα συνδεδεμένων μονάδων που ονομάζονται νουκλεοτίδια. Κάθε νουκλεοτίδιο αποτελείται από τρεις υπομονάδες: μια φωσφορική ομάδα και ένα σάκχαρο (ριβόζη στην περίπτωση του RNA, δεοξυριβόζη στο DNA) αποτελούν τον σκελετό του κλώνου του νουκλεϊκού οξέος και προσκολλημένο στο σάκχαρο είναι ένα από ένα σύνολο νουκλεοβάσεων. Οι νουκλεοβάσεις είναι σημαντικές για τη σύζευξη των κλώνων των βάσεων για να σχηματίσουν υψηλότερο επίπεδο δευτεροταγούς και τριτοταγούς δομής όπως η φημισμένη διπλή έλικα.
Τα πιθανά γράμματα είναι A , C , G και T , που αντιπροσωπεύουν τις τέσσερις βάσεις των νουκλεοτιδίων ενός κλώνου DNA - αδενίνη, κυτοσίνη, γουανίνη, θυμίνη - που συνδέονται ομοιοπολικά με τον σκελετό ενός φωσφοδιεστερικού δεσμού. Στην τυπική περίπτωση, οι αλληλουχίες εκτυπώνονται η μία δίπλα στην άλλη χωρίς κενά, όπως στην αλληλουχία AAAGTCTGAC, που διαβάζεται από τα αριστερά προς τα δεξιά στην κατεύθυνση 5' προς 3'. Όσον αφορά τη μεταγραφή, μια αλληλουχία βρίσκεται στην κωδική αλυσίδα εάν έχει την ίδια σειρά με το μεταγραφόμενο RNA.
Μία αλληλουχία μπορεί να είναι συμπληρωματική σε μια άλλη αλληλουχία, που σημαίνει ότι έχουν τη βάση σε κάθε θέση στη συμπληρωματική (δηλαδή Α στη Τ, C στη G) και στην αντίστροφη σειρά. Για παράδειγμα, η συμπληρωματική αλληλουχία του TTAC είναι GTAA. Εάν μια αλυσίδα του διπλού κλώνου DNA θεωρείται η κωδική αλυσίδα, τότε η άλλη αλυσίδα, η οποία θεωρείται ο μη κωδική αλυσίδα, θα έχει τη συμπληρωματική αλληλουχία της αλυσίδας.
Σημειογραφία
[Επεξεργασία | επεξεργασία κώδικα]Σύγκριση και προσδιορισμός% διαφοράς μεταξύ δύο αλληλουχιών νουκλεοτιδίων.
- AATCCGCTAG
- AAACCCTTAG
- Δεδομένων των δύο αλληλουχιών 10-νουκλεοτιδίων, ευθυγραμμίστε τις και συγκρίνετε τις διαφορές μεταξύ τους. Υπολογίστε το ποσοστό ομοιότητας λαμβάνοντας τον αριθμό διαφορετικών βάσεων DNA δια του συνολικού αριθμού νουκλεοτιδίων Στην παραπάνω περίπτωση, υπάρχουν τρεις διαφορές στην αλληλουχία 10 νουκλεοτιδίων. Επομένως, διαιρέστε το 7/10 για να λάβετε την ομοιότητα 70% και αφαιρέστε το από 100% για να πάρετε μια διαφορά 30%.
Ενώ τα A, T, C και G αντιπροσωπεύουν ένα συγκεκριμένο νουκλεοτίδιο σε μια θέση, υπάρχουν επίσης γράμματα που αντιπροσωπεύουν αμφισημία που χρησιμοποιούνται όταν περισσότερα από ένα είδη νουκλεοτιδίων θα μπορούσαν να εμφανιστούν σε αυτήν τη θέση. Οι κανόνες της Διεθνούς Ένωσης Καθαράς και Εφαρμοσμένης Χημείας (IUPAC) είναι οι εξής: [1]
| Σύμβολο[2] | Περιγραφή | Εμφανιζόμενες βάσεις | Συμπλήρωμα | ||||
|---|---|---|---|---|---|---|---|
| A | Αδενίνη | A | 1 | T | |||
| C | Κυτοσίνη | C | G | ||||
| G | Γουανίνη | G | C | ||||
| T | Θυμίνη | T | A | ||||
| U | Ουρακίλη | U | A | ||||
| W | Ασθενής | A | T | 2 | W | ||
| S | Ισχυρή | C | G | S | |||
| M | Αμίνη | A | C | K | |||
| K | Κετόνη | G | T | M | |||
| R | Πουρίνη | A | G | Y | |||
| Y | Πυριμιδίνη | C | T | R | |||
| B | όχι A (το B έρχεται μετά το A) | C | G | T | 3 | V | |
| D | όχι C (το D έρχεται μετά το C) | A | G | T | H | ||
| H | όχι G (H έρχεται μετά το G) | A | C | T | D | ||
| V | όχι T (V έρχεται μετά το T και το U) | A | C | G | B | ||
| N | οποιοδήποτε Νουκλεοτίδιο (όχι κενό) | A | C | G | T | 4 | N |
| Z | Μηδέν | 0 | Z | ||||
Αυτά τα σύμβολα ισχύουν επίσης για το RNA, εκτός από το U (ουρακίλη) που αντικαθιστά το Τ (θυμίνη).[1]
Εκτός από την αδενίνη (Α), την κυτοσίνη (C), τη γουανίνη (G), τη θυμίνη (Τ) και την ουρακίλη (U), το DNA και το RNA περιέχουν επίσης βάσεις που έχουν τροποποιηθεί μετά το σχηματισμό της αλυσίδας νουκλεϊκών οξέων. Στο DNA, η πιο κοινή τροποποιημένη βάση είναι 5-μεθυλκυτιδίνη (m5C). Στο RNA, υπάρχουν πολλές τροποποιημένες βάσεις, όπως η ψευδοουριδίνη (Ψ), η διυδροουριδίνη (D), η ινοσίνη (Ι), η ριβοθυμιδίνη (rT) και η 7-μεθυλογουανοσίνη (m7G).[3][4] Η υποξανθίνη και η ξανθίνη είναι δύο από τις πολλές βάσεις που δημιουργήθηκαν μέσω της παρουσίας μεταλλαξιογόνου και οι δύο μέσω απαμίνωσης (αντικατάσταση της αμινομάδας με καρβονυλομάδα). Η υποξανθίνη παράγεται από αδενίνη και η ξανθίνη παράγεται από γουανίνη.[5] Ομοίως, η απαμίνωση της κυτοσίνης οδηγεί σε ουρακίλη.
Βιολογική σημασία
[Επεξεργασία | επεξεργασία κώδικα]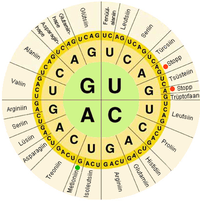
Στα βιολογικά συστήματα, τα νουκλεϊκά οξέα περιέχουν πληροφορίες που χρησιμοποιούνται από ένα ζωντανό κύτταρο για την κατασκευή συγκεκριμένων πρωτεϊνών. Η αλληλουχία της νουκλεοβάσης σε έναν κλώνο νουκλεϊκού οξέος μεταφράζεται από κυτταρικούς μηχανισμούς σε μια αλληλουχία αμινοξέος που αποτελεί έναν κλώνο πρωτεΐνης. Κάθε ομάδα τριών βάσεων, που ονομάζεται κωδικόνιο, αντιστοιχεί σε ένα μόνο αμινοξύ και υπάρχει ένας συγκεκριμένος γενετικός κώδικας με τον οποίο κάθε πιθανός συνδυασμός τριών βάσεων αντιστοιχεί σε ένα συγκεκριμένο αμινοξύ.
Το κεντρικό δόγμα της μοριακής βιολογίας περιγράφει τον μηχανισμό με τον οποίο οι πρωτεΐνες κατασκευάζονται χρησιμοποιώντας πληροφορίες που περιέχονται στα νουκλεϊκά οξέα. Το DNA είναι μεταγράφεται σε mRNA μόρια, τα οποία ταξιδεύουν στο ριβόσωμα όπου το mRNA χρησιμοποιείται ως πρότυπο για την κατασκευή της αλυσίδας της πρωτεΐνης. Δεδομένου ότι τα νουκλεϊκά οξέα μπορούν να συνδεθούν με μόρια με συμπληρωματικές αλληλουχίες, υπάρχει διάκριση μεταξύ κωδικών αλληλουχιών που κωδικοποιούν πρωτεΐνες και της συμπληρωματικής μη κωδικής αλληλουχίας η οποία από μόνη της δεν είναι λειτουργική, αλλά μπορεί να δεσμευτεί με την κωδική αλυσίδα.
Προσδιορισμός αλληλουχίας (ακολουθίας)
[Επεξεργασία | επεξεργασία κώδικα]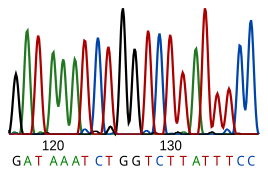
Αλληλούχιση του DNA είναι η διαδικασία προσδιορισμού της αλληλουχίας νουκλεοτιδίων ενός δεδομένου θραύσματος DNA. Η αλληλουχία του DNA ενός έμβιου όντος κωδικοποιεί τις απαραίτητες πληροφορίες ώστε αυτό να επιβιώσει και να αναπαραχθεί. Επομένως, ο καθορισμός της αλληλουχίας είναι χρήσιμος στη βασική έρευνα για το γιατί και πώς ζουν οι οργανισμοί, καθώς και σε εφαρμοσμένα θέματα. Λόγω της σημασίας του DNA για τα έμβια όντα, η γνώση μιας αλληλουχίας DNA μπορεί να είναι χρήσιμη σε σχεδόν κάθε βιολογική έρευνα. Για παράδειγμα, στην ιατρική μπορεί να χρησιμοποιηθεί για την αναγνώριση, διάγνωση και ενδεχομένως στην ανάπτυξη θεραπειών για γενετικές ασθένειες. Ομοίως, η έρευνα για παθογόνα μπορεί να οδηγήσει σε θεραπείες για μεταδοτικές ασθένειες. Η βιοτεχνολογία είναι μια αναπτυσσόμενος κλάδος, με τη δυνατότητα για πολλά χρήσιμα προϊόντα και υπηρεσίες.
Το RNA δεν αλληλουχίζεται άμεσα. Αντίθετα, αντιγράφεται σε ένα DNA από αντίστροφη μεταγραφάση και αυτό το DNA στη συνέχεια αλληλουχίζεται.
Οι τρέχουσες μέθοδοι αλληλούχισης βασίζονται στην ικανότητα διάκρισης των πολυμερασών DNA και ως εκ τούτου μπορούν να διακρίνουν μόνο τις τέσσερις βάσεις. Μια ινοσίνη (που δημιουργήθηκε από αδενοσίνη κατά τη διάρκεια της επεξεργασίας RNA) διαβάζεται ως G και η 5-μεθυλο-κυτοσίνη (δημιουργήθηκε από κυτοσίνη με μεθυλίωση DNA) διαβάζεται ως C. Με την τρέχουσα τεχνολογία, είναι δύσκολη η αλληλουχία μικρών ποσοτήτων DNA, καθώς το σήμα είναι πολύ αδύναμο για μέτρηση. Αυτό ξεπερνιέται με ενίσχυση μέσω αλυσιδωτής αντίδρασης πολυμεράσης (PCR).
Ψηφιακή αναπαράσταση
[Επεξεργασία | επεξεργασία κώδικα]
Μόλις ληφθεί μια αλληλουχία νουκλεϊκών οξέων από έναν οργανισμό, αποθηκεύεται "μέσω υπολογιστή" σε ψηφιακή μορφή. Οι ψηφιακές γενετικές αλληλουχίες μπορούν να αποθηκευτούν σε βάσεις δεδομένων αλληλουχιών, να αναλυθούν (βλέπε "Ανάλυση αλληλουχίας" παρακάτω), να τροποποιηθούν ψηφιακά και να χρησιμοποιηθούν ως πρότυπα για τη δημιουργία νέου πραγματικού DNA χρησιμοποιώντας σύνθεση τεχνητού γονιδίου.
Ανάλυση αλληλουχίας (ακολουθίας)
[Επεξεργασία | επεξεργασία κώδικα]Οι ψηφιακές γενετικές αλληλουχίες μπορούν να αναλυθούν χρησιμοποιώντας τα εργαλεία της βιοπληροφορικής για να προσπαθήσουμε να προσδιορίσουμε τη λειτουργία τους.
Γενετικός έλεγχος
[Επεξεργασία | επεξεργασία κώδικα]Το DNA στο γονιδίωμα ενός οργανισμού μπορεί να αναλυθεί για ευπάθειες στην ιατρική διάγνωση σε κληρονομικές ασθένειες και μπορεί επίσης να χρησιμοποιηθεί για τον προσδιορισμό της πατρότητας ενός παιδιού (γενετικός πατέρας) ή την καταγωγή ενός ατόμου. Κανονικά, κάθε άτομο φέρνει δύο παραλλαγές σε κάθε γονίδιο, η μία κληρονομείται από τη μητέρα του, η άλλη κληρονομείται από τον πατέρα του. Το ανθρώπινο γονιδίωμα πιστεύεται ότι περιέχει περίπου 20.000-25.000 γονίδια. Εκτός από τη μελέτη των χρωμοσωμάτων στο επίπεδο των μεμονωμένων γονιδίων, οι γενετικές εξετάσεις με την ευρύτερη έννοια περιλαμβάνουν τις βιοχημικές εξετάσεις για την πιθανή παρουσία γενετικής νόσου ή μεταλλαγμένων μορφών γονιδίων που σχετίζονται με αυξημένος κίνδυνος ανάπτυξης γενετικών διαταραχών.
Ο γενετικός έλεγχος προσδιορίζει τις αλλαγές στα χρωμοσώματα, τα γονίδια ή τις πρωτεΐνες.[6] Συνήθως, ο έλεγχος χρησιμοποιείται για τον εντοπισμό αλλαγών που σχετίζονται με κληρονομικές διαταραχές. Τα αποτελέσματα μιας γενετικής εξέτασης μπορούν να επιβεβαιώσουν ή να αποκλείσουν μια υποψία γενετικής κατάστασης ή να βοηθήσουν στον προσδιορισμό της πιθανότητας ενός ατόμου να αναπτύξει ή να μεταδώσει μια γενετική διαταραχή. Επί του παρόντος χρησιμοποιούνται αρκετές εκατοντάδες γενετικές εξετάσεις και αναπτύσσονται περισσότερες. [7][8]
Στοίχιση αλληλουχίας (ακολουθίας)
[Επεξεργασία | επεξεργασία κώδικα]Στη βιοπληροφορική, η στοίχιση αλληλουχίας είναι ένας τρόπος τακτοποίησης των αλληλουχιών DNA, RNA ή πρωτεΐνης για την ταυτοποίηση περιοχών ομοιότητας που μπορεί να οφείλονται σε λειτουργικές, δομικές ή εξελικτικές σχέσεις μεταξύ των αλληλουχιών. [9] Εάν δύο αλληλουχίες σε μια στοίχιση μοιράζονται έναν κοινό πρόγονο, οι αναντιστοιχίες μπορούν να ερμηνευτούν ως μεταλλάξεις σημείου και τα κενά ως μεταλλάξεις εισαγωγής ή διαγραφής (indels) που εισήχθησαν σε μία ή και στις δύο γραμμές στο χρόνο από τότε που απέκλιναν η μία από την άλλη. Σε στοιχίσεις αλληλουχιών πρωτεϊνών, ο βαθμός ομοιότητας μεταξύ του αμινοξέος που καταλαμβάνει μια συγκεκριμένη θέση στην αλληλουχία μπορεί να ερμηνευθεί ως ένα χοντρικό μέτρο διατήρησης μιας συγκεκριμένης περιοχής ή μοτίβο αλληλουχίας μεταξύ των γραμμών. Η απουσία υποκαταστάσεων ή η παρουσία μόνο πολύ συντηρητικών υποκαταστάσεων (δηλαδή, η υποκατάσταση αμινοξέων των οποίων οι πλευρικές αλυσίδες έχουν παρόμοιες βιοχημικές ιδιότητες) σε μια συγκεκριμένη περιοχή της αλληλουχίας, προτείνουν[10] ότι αυτή η περιοχή έχει δομική ή λειτουργική σημασία. Αν και οι βάσεις νουκλεοτιδίων DNA και RNA είναι πιο παρόμοιες μεταξύ τους από τα αμινοξέα, η διατήρηση των ζευγών βάσεων μπορεί να υποδηλώνει παρόμοιο λειτουργικό ή δομικό ρόλο.[11]
Η υπολογιστική φυλογενετική κάνει εκτενή χρήση των στοιχίσεων αλληλουχίας στην κατασκευή και ερμηνεία των φυλογενετικών δέντρων, οι οποίες χρησιμοποιούνται για την ταξινόμηση των εξελικτικών σχέσεων μεταξύ ομόλογων γονιδίων που εκπροσωπούνται στα γονιδιώματα αποκλινόντων ειδών. Ο βαθμός στον οποίο διαφέρουν οι αλληλουχίες σε ένα σύνολο ερωτημάτων σχετίζεται ποιοτικά με την εξελικτική απόσταση των αλληλουχιών μεταξύ τους. Σε γενικές γραμμές, η ταυτοποίηση υψηλής αλληλουχίας υποδηλώνει ότι οι εν λόγω αλληλουχίες έχουν ένα σχετικά νέο πιο πρόσφατο κοινό πρόγονο, ενώ η χαμηλή ταυτοποίηση δείχνει ότι η απόκλιση είναι πιο αρχαία. Αυτή η προσέγγιση, που αντικατοπτρίζει την υπόθεση "μοριακού ρολογιού" ότι ένας περίπου σταθερός ρυθμός εξελικτικής αλλαγής μπορεί να χρησιμοποιηθεί για να παρεκτείνει τον χρόνο που έχει παρέλθει από τότε που δύο γονίδια αποκλίνουν για πρώτη φορά (δηλαδή, τον χρόνο συγχώνευσης), υποθέτει ότι τα αποτελέσματα της μετάλλαξης και η επιλογή είναι σταθερά μεταξύ των γραμμών αλληλουχίας. Επομένως, δεν λαμβάνει υπόψη πιθανή διαφορά μεταξύ οργανισμών ή ειδών στα ποσοστά επιδιόρθωσης του DNA ή την πιθανή λειτουργική διατήρηση συγκεκριμένων περιοχών σε μια αλληλουχία. (Στην περίπτωση των νουκλεοτιδικών αλληλουχιών, η υπόθεση του μοριακού ρολογιού στην πιο βασική του μορφή μειώνει επίσης τη διαφορά στα ποσοστά αποδοχής μεταξύ σιωπηλής μετάλλαξης που δεν αλλάζουν την έννοια ενός δεδομένου κωδικονίου και άλλων μεταλλάξεων που καταλήγουν στην ενσωμάτωση ενός διαφορετικού αμινοξέος στην πρωτεΐνη.) Οι πιο στατιστικά ακριβείς μέθοδοι επιτρέπουν τον εξελικτικό ρυθμό σε κάθε κλάδο του φυλογενετικού δέντρου να διαφέρει, παράγοντας έτσι καλύτερες εκτιμήσεις των χρόνων συνένωσης για τα γονίδια.
Μοτίβα αλληλουχίας
[Επεξεργασία | επεξεργασία κώδικα]Συχνά η πρωτογενής δομή κωδικοποιεί μοτίβα λειτουργικής σημασίας. Μερικά παραδείγματα μοτίβων ακολουθίας είναι: το C / D [12] και τα πλαίσια H/ACA[13] των snoRNA s, στη θέση σύνδεσης Sm που βρέθηκε σε σωμάτια συναρμογής RNA όπως U1, U2, U4, U5, U6, U12 και U3, στην αλληλουχία Shine-Dalgarno,[14] στην αλληλουχία συναίνεσης του Κοζάκ[15] και στον τερματιστή ΙΙΙ πολυμεράσης RNA.[16]
Συσχετισμοί μεγάλης εμβέλειας
[Επεξεργασία | επεξεργασία κώδικα]Οι Peng et al.[17][18] βρήκαν την ύπαρξη συσχετίσεων μεγάλης εμβέλειας στις αλληλουχίες DNA που δεν κωδικοποιούν ζεύγη βάσεων. Αντιθέτως, τέτοιοι συσχετισμοί φαίνεται να μην εμφανίζονται σε κωδικοποιητικές αλληλουχίες DNA. Αυτό το εύρημα έχει εξηγηθεί από τους Grosberg et al.[19] από τη γενική χωρική δομή του DNA.
Εντροπία αλληλουχίας (ακολουθίας)
[Επεξεργασία | επεξεργασία κώδικα]Στη βιοπληροφορική, εντροπία αλληλουχίας, γνωστή επίσης ως πολυπλοκότητα αλληλουχίας ή κατατομής (προφίλ) πληροφοριών,[20] είναι μια αριθμητική αλληλουχία που παρέχει ένα ποσοτικό μέτρο της τοπικής πολυπλοκότητας μιας ακολουθίας DNA, ανεξάρτητα από την κατεύθυνση της στοίχιση. Οι χειρισμοί των κατατομών (προφίλ) πληροφοριών καθιστούν δυνατή την ανάλυση των αλληλουχιών χρησιμοποιώντας τεχνικές χωρίς ευθυγράμμιση, όπως για παράδειγμα στην ανίχνευση μοτίβου και αναδιατάξεων.[20][21] [22]
Δείτε επίσης
[Επεξεργασία | επεξεργασία κώδικα]Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ 1,0 1,1 Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences, NC-IUB, 1984.
- ↑ Nomenclature Committee of the International Union of Biochemistry (NC-IUB) (1984). «Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences». Ανακτήθηκε στις 4 Φεβρουαρίου 2008.
- ↑ «BIOL2060: Translation». mun.ca. Αρχειοθετήθηκε από το πρωτότυπο στις 5 Αυγούστου 2020. Ανακτήθηκε στις 18 Ιουνίου 2021.
- ↑ «Research». uw.edu.pl.
- ↑ Nguyen, T; Brunson, D; Crespi, C L; Penman, B W; Wishnok, J S; Tannenbaum, S R (April 1992). «DNA damage and mutation in human cells exposed to nitric oxide in vitro». Proc Natl Acad Sci USA 89 (7): 3030–034. doi:. PMID 1557408. Bibcode: 1992PNAS...89.3030N.
- ↑ «What is genetic testing?». Genetics Home Reference. 16 Μαρτίου 2015. Αρχειοθετήθηκε από το πρωτότυπο στις 29 Μαΐου 2006. Ανακτήθηκε στις 19 Μαΐου 2010.
- ↑ «Genetic Testing». nih.gov.
- ↑ «Definitions of Genetic Testing». Definitions of Genetic Testing (Jorge Sequeiros and Bárbara Guimarães). EuroGentest Network of Excellence Project. 11 Σεπτεμβρίου 2008. Αρχειοθετήθηκε από το πρωτότυπο στις 4 Φεβρουαρίου 2009. Ανακτήθηκε στις 10 Αυγούστου 2008.
- ↑ Mount DM. (2004). Bioinformatics: Sequence and Genome Analysis (2nd έκδοση). Cold Spring Harbor Laboratory Press: Cold Spring Harbor, NY. ISBN 0-87969-608-7.
- ↑ Ng, P. C.; Henikoff, S. (2001). «Predicting Deleterious Amino Acid Substitutions». Genome Research 11 (5): 863–74. doi:. PMID 11337480.
- ↑ Witzany, G (2016). «Crucial steps to life: From chemical reactions to code using agents». Biosystems 140: 49–57. doi:. PMID 26723230. https://philpapers.org/rec/GUECST-2.
- ↑ Samarsky, DA; Fournier MJ; Singer RH; Bertrand E (1998). «The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization». The EMBO Journal 17 (13): 3747–57. doi:. PMID 9649444.
- ↑ Ganot, Philippe; Caizergues-Ferrer, Michèle; Kiss, Tamás (1 April 1997). «The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation». Genes & Development 11 (7): 941–56. doi:. PMID 9106664. https://archive.org/details/sim_genes-development_1997-04-01_11_7/page/941.
- ↑ Shine J, Dalgarno L (1975). «Determinant of cistron specificity in bacterial ribosomes». Nature 254 (5495): 34–38. doi:. PMID 803646. Bibcode: 1975Natur.254...34S.
- ↑ Kozak M (October 1987). «An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs». Nucleic Acids Res. 15 (20): 8125–48. doi:. PMID 3313277.
- ↑ Bogenhagen DF, Brown DD (1981). «Nucleotide sequences in Xenopus 5S DNA required for transcription termination.». Cell 24 (1): 261–70. doi:. PMID 6263489.
- ↑ Peng, C.-K.; Buldyrev, S. V.; Goldberger, A. L.; Havlin, S.; Sciortino, F.; Simons, M.; Stanley, H. E. (1992). «Long-range correlations in nucleotide sequences». Nature 356 (6365): 168–70. doi:. ISSN 0028-0836. PMID 1301010. Bibcode: 1992Natur.356..168P.
- ↑ Peng, C.-K.; Buldyrev, S. V.; Havlin, S.; Simons, M.; Stanley, H. E.; Goldberger, A. L. (1994). «Mosaic organization of DNA nucleotides». Physical Review E 49 (2): 1685–89. doi:. ISSN 1063-651X. PMID 9961383. Bibcode: 1994PhRvE..49.1685P.
- ↑ Grosberg, A; Rabin, Y; Havlin, S; Neer, A (1993). «Crumpled globule model of the three-dimensional structure of DNA». Europhysics Letters 23 (5): 373–78. doi:. Bibcode: 1993EL.....23..373G.
- ↑ 20,0 20,1 Pinho, A; Garcia, S; Pratas, D; Ferreira, P (Nov 21, 2013). «DNA Sequences at a Glance.». PLOS ONE 8 (11): e79922. doi:. PMID 24278218. Bibcode: 2013PLoSO...879922P.
- ↑ Pratas, D; Silva, R; Pinho, A; Ferreira, P (May 18, 2015). «An alignment-free method to find and visualise rearrangements between pairs of DNA sequences.». Scientific Reports 5: 10203. doi:. PMID 25984837. Bibcode: 2015NatSR...510203P.
- ↑ Troyanskaya, O; Arbell, O; Koren, Y; Landau, G; Bolshoy, A (2002). «Sequence complexity profiles of prokaryotic genomic sequences: A fast algorithm for calculating linguistic complexity.». Bioinformatics 18 (5): 679–88. doi:. PMID 12050064. https://archive.org/details/sim_bioinformatics_2002-05_18_5/page/679.

