Γονιδίωμα: Διαφορά μεταξύ των αναθεωρήσεων
Διάσωση 1 πηγών και υποβολή 0 για αρχειοθέτηση.) #IABot (v2.0.8 |
Μετάφραση από την αγγλική wikipedia |
||
| Γραμμή 1: | Γραμμή 1: | ||
[[File:Prokaryotic Cell Diagram.jpg|thumb| Ένα διάγραμμα ετικετών που εξηγεί τα διάφορα μέρη ενός προκαρυωτικού γονιδιώματος]] |
|||
{{Πηγές|20|05|2010}}<br /> |
|||
[[Αρχείο:Genome gradient.jpg|thumb|220px|Βαθμωτή μεταβολή γονιδιώματος εξ αλληλουχίας [[χρωμόσωμα|χρωμοσωμάτων]]]] |
|||
[[File:UCSC human chromosome colours.png|thumb| Μια εικόνα των 46 χρωμοσωμάτων που αποτελούν το διπλοειδές γονιδίωμα ενός άνδρα. (Το μιτοχονδριακό χρωμόσωμα δεν εμφανίζεται.]] |
|||
Τόσο στη [[μοριακή βιολογία]], όσο και στη [[γενετική]], με τον όρο '''γονιδίωμα''' ή '''γένωμα''' (genome) χαρακτηρίζεται το σύνολο του γενετικού υλικού που βρίσκεται σ΄ ένα [[κύτταρο]] ή φέρεται σ΄ ένα άτομο. |
|||
Στα πεδία [[μοριακή βιολογία]] και [[γενετική]], '''γονιδίωμα''' είναι όλο το [[νουκλεϊκά οξέα|γενετικό υλικό]] ενός οργανισμού.<ref name="Roth p. ">{{cite journal | last=Roth | first=Stephanie Clare | title=What is genomic medicine? | journal=Journal of the Medical Library Association | publisher=University Library System, University of Pittsburgh | volume=107 | issue=3 | date=2019-07-01 | issn=1558-9439 | pmid=31258451 | pmc=6579593 | doi=10.5195/jmla.2019.604 }}</ref> Αποτελείται από [[DNA]] (ή [[RNA]] σε ιούς RNA). Το γονιδίωμα περιλαμβάνει τόσο τα [[γονίδιο|γονίδια]] (τις κωδικοποιητικές περιοχές) όσο και το μη κωδικοποιητικό DNA,<ref>{{citation |author=Brosius, J |year=2009 |title=The Fragmented Gene |journal=Annals of the New York Academy of Sciences |doi=10.1111/j.1749-6632.2009.05004.x |pmid=19845638 |volume=1178 |issue=1 |pages=186–93|bibcode=2009NYASA1178..186B |s2cid=8279434 }}</ref> καθώς και το μιτοχονδριακό DNA<ref>{{cite book | url = http://bioinformaticsinstitute.ru/sites/default/files/genome_the_autobiography_of_a_species_in_23_chapters_-_matt_ridley.pdf | vauthors = Ridley M | author-link = Matt Ridley | year = 2006 | title = Genome: the autobiography of a species in 23 chapters | location = New York | publisher = Harper Perennial | isbn = 978-0-06-019497-0 | access-date = 11 May 2016 | archive-url = https://web.archive.org/web/20181024231945/http://bioinformaticsinstitute.ru/sites/default/files/genome_the_autobiography_of_a_species_in_23_chapters_-_matt_ridley.pdf | archive-date = 24 October 2018 | url-status = dead }}</ref> και DNA των χλωροπλαστών. Η μελέτη του γονιδιώματος ονομάζεται [[γονιδιωματική]]. |
|||
== Προέλευση του όρου == |
|||
Ο όρος ''γονιδίωμα'' δημιουργήθηκε το 1920 από τον Hans Winkler, |
|||
<ref>{{cite book | vauthors = Winkler HL | title = Verbreitung und Ursache der Parthenogenesis im Pflanzen- und Tierreiche| url = https://archive.org/details/verbreitungundur00wink |date=1920|publisher=Verlag Fischer|location=Jena}}</ref> καθηγητή της [[βοτανική]]ς στο [[Πανεπιστήμιο του Αμβούργου]], Γερμανία. Το λεξικό Όξφορντ προτείνει το όνομα ως μείξη των λέξεων ''[[γονίδιο]]'' και ''[[χρωμόσωμα]]''<ref>{{cite web|title=definition of Genome in Oxford dictionary|url=http://www.oxforddictionaries.com/us/definition/american_english/genome|access-date=25 March 2014}}</ref> |
|||
== Αλληλούχιση και χαρτογράφηση == |
|||
Η αλληλουχία του γονιδιώματος είναι ο πλήρης κατάλογος των [[νουκλεοτίδιο|νουκλεοτιδίων]] (A, C, G και T για τα γονιδιώματα DNA) που αποτελούν όλα τα [[χρωμόσωμα|χρωμοσώματα]] ενός ατόμου ή ενός είδους. Σε ένα είδος, η συντριπτική πλειονότητα των νουκλεοτιδίων είναι ταυτόσημη μεταξύ των ατόμων, αλλά η αλληλούχιση πολλών ατόμων είναι απαραίτητη για την κατανόηση της γενετικής ποικιλομορφίας. |
|||
[[File:Part of DNA sequence prototypification of complete genome of virus 5418 nucleotides.gif|thumb|right|350 px|Μέρος της αλληλουχίας DNA - πρωτοτυποποίηση του πλήρους γονιδιώματος του ιού]] |
|||
Το 1976, ο Walter Fiers στο [[Πανεπιστήμιο της Γάνδης]] (Βέλγιο) ήταν ο πρώτος που καθιέρωσε την πλήρη νουκλεοτιδική αλληλουχία ενός ιικού γονιδιώματος RNA ([[βακτηριοφάγος]] MS2). Το επόμενο έτος, ο Fred Sanger ολοκλήρωσε την πρώτη αλληλουχία γονιδιώματος DNA : Φάγος Φ-Χ174, από 5386 ζεύγη βάσεων.<ref>{{cite web|url=http://www.beowulf.org.uk/|title=All about genes|website=www.beowulf.org.uk}}</ref> |
|||
Οι πρώτες πλήρεις αλληλουχίες γονιδιώματος μεταξύ των τριών επικρατειών της ζωής κυκλοφόρησαν σε σύντομο χρονικό διάστημα κατά τα μέσα της δεκαετίας του 1990: Το πρώτο βακτηριακό γονιδίωμα το οποίο αλληλουχήθηκε ήταν εκείνο της Haemophilus influenzae, που ολοκληρώθηκε από μια ομάδα στο "The Institute for Genomic Research" το 1995. Λίγους μήνες αργότερα, ολοκληρώθηκε το πρώτο ευκαρυωτικό γονιδίωμα, με αλληλουχίες των 16 χρωμοσωμάτων της εκβλάστησης ζύμης ''Saccharomyces cerevisiae'' που δημοσιεύθηκε ως αποτέλεσμα μιας ευρωπαϊκής προσπάθειας που ξεκίνησε το στα μέσα της δεκαετίας του 1980. Η πρώτη ακολουθία γονιδιώματος για ένα [[αρχαία|αρχαίο]], το ''Methanococcus jannaschii'', ολοκληρώθηκε το 1996, ξανά από το Ινστιτούτο Γονιδιωματικής Έρευνας. |
|||
Η ανάπτυξη νέων τεχνολογιών έχει κάνει την αλληλούχιση γονιδιώματος πολύ πιο φτηνή και πιο εύκολη και ο αριθμός των πλήρων αλληλουχιών γονιδιώματος αυξάνεται ραγδαία. Τα [[Εθνικά Ινστιτούτα Υγείας (ΗΠΑ)]] διατηρούν μία από τις πολλές ολοκληρωμένες βάσεις δεδομένων γονιδιωματικών πληροφοριών.<ref>{{cite web|url=https://www.ncbi.nlm.nih.gov/sites/entrez?db=Genome&itool=toolbar |title=Genome Home |date=2010-12-08 |access-date=27 January 2011}}</ref> |
|||
Μεταξύ των χιλιάδων ολοκληρωμένων έργων αλληλούχισης του γονιδιώματος περιλαμβάνονται εκείνα για το [[ρύζι]], το ποντίκι, το φυτό ''Arabidopsis thaliana'', τις τετραοδοντίδες και τα βακτήρια [[Εσερίχια κόλι]]. Τον Δεκέμβριο του 2013, οι επιστήμονες αλληλούχισαν για πρώτη φορά ολόκληρο το ''γονιδίωμα'' ενός [[Νεάντερταλ]], ενός εξαφανισμένου είδους [[Αρχαϊκοί Homo sapiens|ανθρώπων]]. Το γονιδίωμα εξήχθη από το οστό δακτύλου ενός Νεάντερταλ περίπου 130.000 ετών που βρέθηκε σε Σπήλαιο της Σιβηρίας.<ref name="NYT-20131218">{{cite news |last=Zimmer |first=Carl | name-list-style = vanc |author-link=Carl Zimmer |title=Toe Fossil Provides Complete Neanderthal Genome |url=https://www.nytimes.com/2013/12/19/science/toe-fossil-provides-complete-neanderthal-genome.html |date=December 18, 2013 |work=[[The New York Times]] |access-date=18 December 2013}}</ref><ref name="NAT-20131218">{{cite journal | vauthors = Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, Heinze A, Renaud G, Sudmant PH, de Filippo C, Li H, Mallick S, Dannemann M, Fu Q, Kircher M, Kuhlwilm M, Lachmann M, Meyer M, Ongyerth M, Siebauer M, Theunert C, Tandon A, Moorjani P, Pickrell J, Mullikin JC, Vohr SH, Green RE, Hellmann I, Johnson PL, Blanche H, Cann H, Kitzman JO, Shendure J, Eichler EE, Lein ES, Bakken TE, Golovanova LV, Doronichev VB, Shunkov MV, Derevianko AP, Viola B, Slatkin M, Reich D, Kelso J, Pääbo S | display-authors = 6 | title = The complete genome sequence of a Neanderthal from the Altai Mountains | journal = Nature | volume = 505 | issue = 7481 | pages = 43–49 | date = January 2014 | pmid = 24352235 | pmc = 4031459 | doi = 10.1038/nature12886 | bibcode = 2014Natur.505...43P }}</ref> |
|||
Νέες τεχνολογίες αλληλούχισης, όπως μαζική παράλληλη αλληλούχιση έχουν επίσης ανοίξει την προοπτική της αλληλούχισης του προσωπικού γονιδιώματος ως διαγνωστικού εργαλείου, όπου πρωτοστάτησε η Manteia Predictive Medicine. Ένα σημαντικό βήμα προς αυτόν τον στόχο ήταν η ολοκλήρωση το 2007 του [[ανθρώπινο γονιδίωμα|πλήρους γονιδιώματος]] του James D. Watson, ενός από τα άτομα που ανακάλυψαν τη δομή του DNA.<ref>{{cite news| url=https://www.nytimes.com/2007/05/31/science/31cnd-gene.html | work=The New York Times | title=Genome of DNA Pioneer Is Deciphered | first=Nicholas | last=Wade | name-list-style = vanc | date=2007-05-31 | access-date=2 April 2010}}</ref> |
|||
Ενώ μια αλληλουχία γονιδιώματος παραθέτει τη σειρά κάθε βάσης DNA σε ένα γονιδίωμα, ένας χάρτης γονιδιώματος προσδιορίζει τα σημεία αναφοράς. Ένας χάρτης γονιδιώματος είναι λιγότερο λεπτομερής από μια αλληλουχία γονιδιώματος και βοηθά στην πλοήγηση γύρω από το γονιδίωμα. Το έργο του ανθρωπίνου γονιδιώματος οργανώθηκε σε χάρτη και σε αλληλούχιση του ανθρωπίνου γονιδιώματος. Ένα βασικό βήμα στο έργο ήταν η έκδοση ενός λεπτομερούς γονιδιωματικού χάρτη από τον Jean Weissenbach και την ομάδα του στο Genoscope στο Παρίσι.<ref>{{cite web|url=http://www.genomenewsnetwork.org/resources/whats_a_genome/Chp3_1.shtml |title=What's a Genome? |publisher=Genomenewsnetwork.org |date=2003-01-15 |access-date=27 January 2011}}</ref><ref>{{cite web|author=NCBI_user_services |url=https://www.ncbi.nlm.nih.gov/About/primer/mapping.html |title=Mapping Factsheet |date=2004-03-29 |access-date=27 January 2011 |url-status=dead |archive-url=https://web.archive.org/web/20100719235548/http://www.ncbi.nlm.nih.gov/About/primer/mapping.html |archive-date=19 July 2010 |df=dmy }}</ref> |
|||
Οι αλληλουχίες του γονιδιώματος αναφοράς και οι χάρτες συνεχίζουν να ενημερώνονται, αφαιρώντας τα λάθη και αποσαφηνίζοντας περιοχές με υψηλή πολυπλοκότητα αλληλομόρφων.<ref>{{cite web|author=Genome Reference Consortium |url=https://www.ncbi.nlm.nih.gov/projects/genome/assembly/grc/info/ |title=Assembling the Genome |access-date=23 August 2016}}</ref> Το μειωμένο κόστος της γονιδιωματικής χαρτογράφησης επέτρεψε στους [[γενεαλογία|γενεαλογικούς]] ιστότοπους να την προσφέρουν ως υπηρεσία,<ref>{{cite news | url=https://www.washingtonpost.com/news/speaking-of-science/wp/2016/05/17/how-do-your-20000-genes-determine-so-many-wildly-different-traits-they-multitask/ | title=How do your 20,000 genes determine so many wildly different traits? They multitask. | newspaper=The Washington Post | date=2016-04-17 | access-date=2016-08-27 | author=Kaplan, Sarah}}</ref> στο βαθμό που κάποιος μπορεί να υποβάλει το γονιδίωμα κάποιου σε πληθοπορισμικά εγχειρήματα όπως DNA.LAND στο Κέντρο Γονιδιώματος της Νέας Υόρκης,<ref name="Hayden">{{cite journal|last1=Check Hayden|first1=Erika|title=Scientists hope to attract millions to 'DNA.LAND'|url=https://www.nature.com/news/scientists-hope-to-attract-millions-to-dna-land-1.18514|journal=Nature|language=en|doi=10.1038/nature.2015.18514|year=2015}}</ref> ένα παράδειγμα τόσο [[οικονομίες κλίμακας|οικονομιών κλίμακας]] όσο και επιστήμης των πολιτών.<ref name="DNA.land">{{cite web | url=https://www.statnews.com/feature/game-of-genomes/season-three/ | title=Game of Genomes, Episode 13: Answers and Questions | publisher=STAT | access-date=2016-08-27 | author=Zimmer, Carl}}</ref> |
|||
== Ιικά γονιδιώματα == |
|||
Τα ιικά γονιδιώματα μπορεί να αποτελούνται είτε από RNA ή από DNA. Τα γονιδιώματα των ιών RNA μπορεί να είναι είτε μονόκλωνα, είτε δίκλωνα RNA και μπορεί να περιέχουν ένα ή περισσότερα ξεχωριστά μόρια RNA (τμήματα: μονομερή ή πολυμερή γονιδιώματος). Οι ιοί DNA μπορούν να έχουν είτε μονόκλωνα είτε δίκλωνα γονιδιώματα. Τα περισσότερα γονιδιώματα ιών DNA αποτελούνται από ένα μόνο γραμμικό μόριο DNA, αλλά μερικά αποτελούνται από ένα μόριο κυκλικού DNA.<ref>{{cite book|last1=Gelderblom|first1=Hans R.|title=Medical Microbiology|date=1996|publisher=The University of Texas Medical Branch at Galveston|location=Galveston, TX|edition=4th|url=https://www.ncbi.nlm.nih.gov/books/NBK8174/}}</ref> Υπάρχουν επίσης ιικά RNA που είναι μονόκλωνα RNA και χρησιμεύουν ως πρότυπο για τη σύνθεση mRNA<ref>{{Cite book|last=Urry|first=Lisa A.|title=Campbell Biology: seventh edition|publisher=Hoboken: Pearson Higher Education|year=2016|isbn=0134093410|location=New York|pages=403–404}}</ref> και μονόκλωνα RNA που χρησιμεύουν ως πρότυπα για τη σύνθεση DNA. |
|||
Iικός φάκελος<ref>{{Cite book|last=Urry|first=Lisa A|title=Campbell Biology: seventh edition|publisher=Hoboken: Pearson Higher Education|year=2016|isbn=0134093410|location=New York|pages=403–404}}</ref> |
|||
είναι ένα εξωτερικό στρώμα μεμβράνης που χρησιμοποιούν τα ιικά γονιδιώματα για να εισέλθουν στο κύτταρο του ξενιστή. Ορισμένες από τις κατηγορίες ιικού DNA και RNA έχουν ιικό φάκελο, ενώ άλλες όχι. |
|||
== Προκαρυωτικά γονιδιώματα == |
|||
Τα προκαρυωτικά και τα ευκαρυωτικά κύτταρα έχουν γονιδιώματα DNA. Τα αρχαία και τα περισσότερα βακτήρια έχουν ένα μοναδικό κυκλικό χρωμόσωμα.<ref>{{cite journal | vauthors = Samson RY, Bell SD | title = Archaeal chromosome biology | journal = Journal of Molecular Microbiology and Biotechnology | volume = 24 | issue = 5–6 | pages = 420–27 | date = 2014 | pmid = 25732343 | pmc = 5175462 | doi = 10.1159/000368854 }}</ref> Ωστόσο, ορισμένα βακτηριακά είδη έχουν γραμμικά ή πολλαπλά χρωμοσώματα.<ref>{{cite journal|title=Replication of Linear Bacterial Chromosomes: No Longer Going Around in Circles|journal=The Bacterial Chromosome|date=2005|pages=525–540|doi=10.1128/9781555817640.ch29|url=http://www.asmscience.org/content/book/10.1128/9781555817640.chap29 | last1 = Chaconas | first1 = George | last2 = Chen | first2 = Carton W. | name-list-style = vanc |isbn=9781555812324}}</ref><ref>{{cite web|url=http://www.sci.sdsu.edu/~smaloy/MicrobialGenetics/topics/chroms-genes-prots/chromosomes.html|title=Bacterial Chromosomes|website=Microbial Genetics|year=2002}}</ref> |
|||
Εάν το DNA αντιγράφεται γρηγορότερα από ό,τι διαιρούνται τα βακτηριακά κύτταρα, μπορούν να υπάρχουν πολλαπλά αντίγραφα του χρωμοσώματος σε ένα μοναδικό κύτταρο και εάν τα κύτταρα διαιρούνται γρηγορότερα από ό,τι μπορεί να αναπαραχθεί το DNA, ξεκινά πολλαπλή αναπαραγωγή του χρωμοσώματος πριν συμβεί η διαίρεση, επιτρέποντας στα θυγατρικά κύτταρα να κληρονομήσουν πλήρη γονιδιώματα και ήδη μερικώς αναπαραγόμενα χρωμοσώματα. Τα περισσότερα προκαρυωτικά κύτταρα έχουν πολύ λίγο επαναλαμβανόμενο DNA στα γονιδιώματά τους.<ref name="constraints and plasticity in genome and molecular">{{cite journal | vauthors = Koonin EV, Wolf YI | title = Constraints and plasticity in genome and molecular-phenome evolution | journal = Nature Reviews. Genetics | volume = 11 | issue = 7 | pages = 487–98 | date = July 2010 | pmid = 20548290 | pmc = 3273317 | doi = 10.1038/nrg2810 }}</ref> Ωστόσο, ορισμένα συμβιωτικά βακτήρια (π.χ. ''Serratia symbiotica'') έχουν μειωμένα γονιδιώματα και ένα υψηλό κλάσμα ψευδογονιδίων: μόνο το περίπου 40% του DNA τους κωδικοποιεί πρωτεΐνες<ref>{{cite journal | vauthors = McCutcheon JP, Moran NA | title = Extreme genome reduction in symbiotic bacteria | journal = Nature Reviews. Microbiology | volume = 10 | issue = 1 | pages = 13–26 | date = November 2011 | pmid = 22064560 | doi = 10.1038/nrmicro2670 | s2cid = 7175976 }}</ref><ref>{{cite journal | vauthors = Land M, Hauser L, Jun SR, Nookaew I, Leuze MR, Ahn TH, Karpinets T, Lund O, Kora G, Wassenaar T, Poudel S, Ussery DW | title = Insights from 20 years of bacterial genome sequencing | journal = Functional & Integrative Genomics | volume = 15 | issue = 2 | pages = 141–61 | date = March 2015 | pmid = 25722247 | pmc = 4361730 | doi = 10.1007/s10142-015-0433-4 }}</ref> |
|||
Μερικά βακτήρια έχουν βοηθητικό γενετικό υλικό, επίσης μέρος του γονιδιώματός τους, το οποίο μεταφέρεται σε [[πλασμίδιο|πλασμίδια]]. Για αυτό, η λέξη ''γονιδίωμα'' δεν πρέπει να χρησιμοποιείται ως συνώνυμο του ''χρωμοσώματος''. |
|||
== Ευκαρυωτικά γονιδιώματα == |
|||
Τα ευκαρυωτικά γονιδιώματα αποτελούνται από ένα ή περισσότερα γραμμικά χρωμοσώματα DNA. Ο αριθμός των χρωμοσωμάτων ποικίλλει σημαντικά από ''Myrmecia pilosula'' και ''Diploscapter pachys (asexual nemotode)'', |
|||
<ref>{{cite web|title=Scientists sequence asexual tiny worm whose lineage stretches back 18 million years|url=https://www.sciencedaily.com/releases/2017/09/170921141303.htm|website=ScienceDaily|access-date=7 November 2017}}</ref> που το καθένα έχει μόνο ένα ζεύγος, σε ''Ophioglossum (είδος φτέρης)'' που έχει 720 ζεύγη. |
|||
<ref>{{cite journal|last1=Khandelwal|first1=Sharda |title=Chromosome evolution in the genus Ophioglossum L.|journal=Botanical Journal of the Linnean Society|date=March 1990|volume=102|issue=3|pages=205–17|doi=10.1111/j.1095-8339.1990.tb01876.x }}</ref> Είναι εκπληκτικό το ποσό του DNA που περιέχουν τα ευκαρυωτικά γονιδιώματα σε σύγκριση με άλλα γονιδιώματα. Η ποσότητα είναι ακόμη μεγαλύτερη από ό,τι είναι απαραίτητη για κωδικοποιητικά γονίδια πρωτεϊνών DNA και μη κωδικοποιητικά γονίδια λόγω του γεγονότος ότι τα ευκαρυωτικά γονιδιώματα εμφανίζουν παραλλαγές έως και 64.000 φορές στα μεγέθη τους.<ref name=":0"/> Ωστόσο, αυτό το ιδιαίτερο χαρακτηριστικό προκαλείται από την παρουσία επαναλαμβανόμενου DNA και μεταθετών στοιχείων (TEs). |
|||
Ένα τυπικό ανθρώπινο κύτταρο έχει δύο αντίγραφα καθένα από τα 22 ζεύγη [[αυτοσωμικό χρωμόσωμα|αυτοσωμάτων]], που κανονικά το καθένα κληρονομείται από κάθε γονέα, συν δύο [[φυλετικό χρωμόσωμα|φυλετικά χρωμοσώματα]], καθιστώντας το διπλοειδές. Οι [[γαμέτης|γαμέτες]], όπως τα ωάρια, το σπέρμα, τα σπόρια και η γύρη, είναι απλοειδή, που σημαίνει ότι φέρουν μόνο ένα αντίγραφο κάθε χρωμοσώματος. Εκτός από τα χρωμοσώματα στον πυρήνα, οργανίδια όπως οι [[χλωροπλάστης|χλωροπλάστες]] και τα [[μιτοχόνδριο|μιτοχόνδρια]] έχουν το δικό τους DNA. Τα μιτοχόνδρια μερικές φορές λέγεται ότι έχουν το δικό τους γονιδίωμα που συχνά αναφέρεται ως "μιτοχονδριακό γονιδίωμα". Το DNA που βρίσκεται μέσα στον χλωροπλάστη μπορεί να αναφέρεται ως "πλαστωμα". Όπως τα βακτήρια από τα οποία προέρχονται, τα μιτοχόνδρια και οι χλωροπλάστες έχουν κυκλικό χρωμόσωμα. |
|||
Σε αντίθεση με τα προκαρυωτικά, τα ευκαρυωτικά έχουν οργάνωση γονιδίων εξωνίων-εσωνίων που κωδικοποιούν πρωτεΐνες και μεταβλητές ποσότητες επαναλαμβανόμενου DNA. Στα θηλαστικά και τα φυτά, η πλειονότητα του γονιδιώματος αποτελείται από επαναλαμβανόμενο DNA.<ref name="Lewin 2004">{{cite book | last = Lewin | first = Benjamin | title=Genes VIII|date=2004|publisher=Pearson/Prentice Hall|location=Upper Saddle River, NJ|isbn=978-0-13-143981-8|edition=8th}}</ref> |
|||
Τα γονίδια στα ευκαρυωτικά γονιδιώματα μπορούν να περιγραφούν χρησιμοποιώντας το FINDER.<ref name="pmid33879057">{{cite journal | vauthors = Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM | title = FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences | journal = BMC Bioinformatics | volume = 44 | issue = 9 | pages = e89 | date = Apr 2021 | pmid = 33879057 | doi = 10.1186/s12859-021-04120-9 | doi-access = free }}</ref> |
|||
=== Αλληλουχίες κωδικοποίησης === |
|||
Οι αλληλουχίες DNA που φέρουν τις οδηγίες για την παραγωγή πρωτεϊνών αναφέρονται ως κωδικοποιητικές αλληλουχίες. Η αναλογία του γονιδιώματος που καταλαμβάνεται από κωδικοποιητικές αλληλουχίες ποικίλλει ευρέως. Ένα μεγαλύτερο γονιδίωμα δεν περιέχει απαραίτητα περισσότερα γονίδια και η αναλογία του μη επαναλαμβανόμενου DNA μειώνεται μαζί με την αύξηση του μεγέθους του γονιδιώματος σε πολύπλοκους ευκαρυώτες.<ref name="Lewin 2004"/>[[File:Components of the human genome.png|thumb| Σύνθεση του ανθρωπίνου γονιδιώματος.]] |
|||
=== Αλληλουχίες χωρίς κωδικοποίηση === |
|||
Οι μη κωδικοποιητικές αλληλουχίες περιλαμβάνουν [[ιντρόνιο|εσώνια]], αλληλουχίες για μη κωδικοποιητικά RNA, ρυθμιστικές περιοχές και επαναλαμβανόμενο DNA. Οι μη κωδικοποιητικές αλληλουχίες αποτελούν το 98% του ανθρώπινου γονιδιώματος. Υπάρχουν δύο κατηγορίες επαναλαμβανόμενων DNA στο γονιδίωμα: διαδοχικές επαναλήψεις και διάσπαρτες επαναλήψεις.<ref>{{cite book|editor-last=Stojanovic|editor-first=Nikola|title=Computational genomics : current methods|date=2007|publisher=Horizon Bioscience|location=Wymondham|isbn=978-1-904933-30-4}}</ref> |
|||
==== Διαδοχικές επαναλήψεις ==== |
|||
Σύντομες, μη κωδικοποιητικές αλληλουχίες που επαναλαμβάνονται από την αρχή έως το τέλος καλούνται διαδοχικές επαναλήψεις. Μικροδορυφόροι αποτελούμενοι από 2-5 επαναλήψεις ζεύγους βάσεων, ενώ οι επαναλήψεις μινιδορυφόρων είναι 30-35 bp. Οι διαδοχικές επαναλήψεις αποτελούν περίπου το 4% του ανθρώπινου γονιδιώματος και το 9% του γονιδιώματος της μύγας των φρούτων.<ref name="Padeken">{{cite journal | vauthors = Padeken J, Zeller P, Gasser SM | title = Repeat DNA in genome organization and stability | journal = Current Opinion in Genetics & Development | volume = 31 | pages = 12–19 | date = April 2015 | pmid = 25917896 | doi = 10.1016/j.gde.2015.03.009 }}</ref> Οι διαδοχικές επαναλήψεις μπορούν να είναι λειτουργικές. Για παράδειγμα, τα [[τελομερές|τελομερή]] αποτελούνται από την διαδοχική επανάληψη TTAGGG στα θηλαστικά και παίζουν σημαντικό ρόλο στην προστασία των άκρων του χρωμοσώματος. |
|||
Σε άλλες περιπτώσεις, οι επεκτάσεις του αριθμού των διαδοχικών επαναλήψεων σε εξώνια ή εσώνια μπορεί να προκαλέσει την ασθένεια της τρινουκλεοτιδικής επαναλαμβανόμενης διαταραχής.<ref name="Usdin">{{cite journal | vauthors = Usdin K | title = The biological effects of simple tandem repeats: lessons from the repeat expansion diseases | journal = Genome Research | volume = 18 | issue = 7 | pages = 1011–19 | date = July 2008 | pmid = 18593815 | pmc = 3960014 | doi = 10.1101/gr.070409.107 }}</ref> Για παράδειγμα, το ανθρώπινο γονίδιο χαντιγκτίνη περιέχει συνήθως 6-29 διαδοχικές επαναλήψεις των νουκλεοτιδίων CAG (κωδικοποιώντας μια πολυγλουταμίνη). Η επέκταση σε πάνω από 36 επαναλήψεις οδηγεί στη [[νόσος του Χάντινγκτον|νόσο του Χάντινγκτον]], μια νευροεκφυλιστική ασθένεια. Είκοσι ανθρώπινες διαταραχές είναι γνωστό ότι προκύπτουν από παρόμοιες διαδοχικές επαναλαμβανόμενες επεκτάσεις σε διάφορα γονίδια. Ο μηχανισμός με τον οποίο οι πρωτεΐνες με διευρυμένες οδούς πολυγλουταμίνης προκαλούν θάνατο των νευρώνων δεν είναι πλήρως κατανοητός. Μία πιθανότητα είναι ότι οι πρωτεΐνες αποτυγχάνουν να διπλώνονται σωστά και να αποφεύγουν την αποικοδόμηση, αντί να συσσωρεύονται σε συσσωματώματα που επίσης δεσμεύουν σημαντικούς παράγοντες μεταγραφής, μεταβάλλοντας έτσι τη γονιδιακή έκφραση.<ref name="Usdin"/> |
|||
Οι διαδοχικές επαναλήψεις προκαλούνται συνήθως από ολίσθηση κατά την αναπαραγωγή, άνισο επιχιασμό και μετατροπή γονιδίων.<ref>{{cite journal | vauthors = Li YC, Korol AB, Fahima T, Beiles A, Nevo E | title = Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review | journal = Molecular Ecology | volume = 11 | issue = 12 | pages = 2453–65 | date = December 2002 | pmid = 12453231 | doi = 10.1046/j.1365-294X.2002.01643.x | s2cid = 23606208 | doi-access = free }}</ref> |
|||
==== Μεταθετά στοιχεία ==== |
|||
Τα μεταθετά στοιχεία (ΤΕ) είναι αλληλουχίες DNA με καθορισμένη δομή που μπορούν να αλλάξουν τη θέση τους στο γονιδίωμα.<ref name="Padeken" /><ref name="constraints and plasticity in genome and molecular" /><ref name="Wessler 17600–17601">{{cite journal | vauthors = Wessler SR | title = Transposable elements and the evolution of eukaryotic genomes | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 103 | issue = 47 | pages = 17600–01 | date = November 2006 | pmid = 17101965 | doi = 10.1073/pnas.0607612103 | bibcode = 2006PNAS..10317600W | pmc = 1693792 }}</ref> Τα TE κατηγοριοποιούνται είτε ως ένας μηχανισμός που αναπαράγεται με αντιγραφή και επικόλληση, είτε ως μηχανισμός που μπορεί να αποκοπεί από το γονιδίωμα και να εισαχθεί σε μια νέα θέση. Στο ανθρώπινο γονιδίωμα, υπάρχουν τρεις σημαντικές τάξεις ΤΕ που αποτελούν περισσότερο από το 45% του ανθρώπινου DNA. Αυτές οι τάξεις είναι τα μακρά διασπαρμένα πυρηνικά στοιχεία (long interspersed nuclear elements ή LINEs), τα διασπαρμένα πυρηνικά στοιχεία (interspersed nuclear elements ή SINEs) και οι ενδογενείς ρετροϊοί. Αυτά τα στοιχεία έχουν μεγάλες δυνατότητες τροποποίησης του γενετικού ελέγχου σε έναν οργανισμό ξενιστή.<ref name=":0">{{Cite journal|last=Zhou|first=Wanding|last2=Liang|first2=Gangning|last3=Molloy|first3=Peter L.|last4=Jones|first4=Peter A.|date=11 August 2020|title=DNA methylation enables transposable element-driven genome expansion|url=https://pubmed.ncbi.nlm.nih.gov/32719115/|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=117|issue=32|pages=19359–19366|doi=10.1073/pnas.1921719117|issn=1091-6490|pmc=7431005|pmid=32719115}}</ref> |
|||
Η κίνηση των TEs είναι η κινητήρια δύναμη της εξέλιξης του γονιδιώματος στους ευκαρυώτες, επειδή η εισαγωγή τους μπορεί να διαταράξει τις γονιδιακές λειτουργίες, ο ομόλογος ανασυνδυασμός μεταξύ των TE μπορεί να προκαλέσει διπλασιασμούς και το TE μπορεί να ανακατέψει τα εξώνια και τις ρυθμιστικές αλληλουχίες σε νέες τοποθεσίες.<ref name="Kazazian 1626–1632">{{cite journal | vauthors = Kazazian HH | s2cid = 1956932 | title = Mobile elements: drivers of genome evolution | journal = Science | volume = 303 | issue = 5664 | pages = 1626–32 | date = March 2004 | pmid = 15016989 | doi = 10.1126/science.1089670 | bibcode = 2004Sci...303.1626K }}</ref> |
|||
===== Ρετρομεταθετόνια (Retrotransposons) ===== |
|||
Τα ρετρομεταθετόνια<ref>{{Cite web|title=Transposon {{!}} genetics|url=https://www.britannica.com/science/transposon|access-date=2020-12-05|website=Encyclopedia Britannica|language=en}}</ref> βρίσκονται κυρίως σε ευκαρυώτες αλλά δεν βρίσκονται σε προκαρυώτες και τα ρετρομεταθετόνια σχηματίζουν μεγάλο μέρος των γονιδιωμάτων πολλών ευκαρυωτικών. Το ρετρομεταθετόνιο είναι ένα μεταθετό στοιχείο που μεταφέρεται μέσω ενός ενδιάμεσου [[RNA]]. Τα ρετρομεταθετόνια<ref>{{Cite book|last=Sanders|first=Mark Frederick|title=Genetic Analysis: an integrated approach third edition|publisher=Pearson, always learning, and mastering|year=2019|isbn=9780134605173|location=New York|pages=425}}</ref> αποτελούνται από [[DNA]], αλλά μεταγράφονται σε RNA για τη μεταφορά, έπειτα το μεταγράφημα RNA αντιγράφεται πίσω στον σχηματισμό DNA με τη βοήθεια ενός συγκεκριμένου ενζύμου που ονομάζεται αντίστροφη μεταγραφάση. Τα ρετρομεταθετόνια που φέρουν αντίστροφη μεταγραφάση στο γονίδιο τους μπορούν να προκαλέσουν τη δική της μεταφορά, αλλά τα γονίδια που δεν έχουν την αντίστροφη μεταγραφάση πρέπει να χρησιμοποιησουν την αντίστροφη μεταγραφάση που συντίθεται από άλλο ρετρομεταθετόνιο. Τα ρετρομεταθετόνια μπορούν να μεταγραφούν σε RNA, που στη συνέχεια διπλασιάζονται σε άλλη θέση στο γονιδίωμα.<ref>{{cite journal | vauthors = Deininger PL, Moran JV, Batzer MA, Kazazian HH | title = Mobile elements and mammalian genome evolution | journal = Current Opinion in Genetics & Development | volume = 13 | issue = 6 | pages = 651–58 | date = December 2003 | pmid = 14638329 | doi = 10.1016/j.gde.2003.10.013 }}</ref> Τα ρετρομεταθετόνια μπορούν να χωριστούν σε μακριές ακραίες επαναλήψεις (long terminal repeats ή LTRs) και σε μη μακριές ακραίες επαναλήψεις (Non-LTRs).<ref name="Kazazian 1626–1632"/> |
|||
Οι '''μακριές ακραίες επαναλήψεις Long terminal repeats (LTRs)''' προέρχονται από αρχαίες ρετροϊικές λοιμώξεις, επομένως κωδικοποιούν πρωτεΐνες που σχετίζονται με ρετροϊικές πρωτεΐνες, συμπεριλαμβανομένων ομαδοειδικών αντιγόνων (group-specific antigen ή gag), γλυκοζαμινογλυκάνης, pol (πολυμερασών - αντίστροφη μεταγραφάση και ιντεγκράση), pro (πρωτεάση) και σε μερικές περιπτώσεις φακέλους (env) γονιδίων.<ref name="Wessler 17600–17601"/> Αυτά τα γονίδια πλαισιώνονται από μεγάλες επαναλήψεις και στα δύο άκρα 5' και 3'. Έχει αναφερθεί ότι τα LTRs αποτελούν το μεγαλύτερο κλάσμα στα περισσότερα φυτικά γονιδιώματα και ενδέχεται να αντιπροσωπεύουν την τεράστια διακύμανση στο μέγεθος του γονιδιώματος.<ref>{{cite journal | vauthors = Kidwell MG, Lisch DR | title = Transposable elements and host genome evolution | journal = Trends in Ecology & Evolution | volume = 15 | issue = 3 | pages = 95–99 | date = March 2000 | pmid = 10675923 | doi = 10.1016/S0169-5347(99)01817-0 }}</ref> |
|||
'''Οι μη μακριές ακραίες επαναλήψεις Non-long terminal repeats (Non-LTR)''' ταξινομούνται ως μακρά διάσπαρτα πυρηνικά στοιχεία (long interspersed nuclear elements (LINEs)), βραχέα διάσπαρτα πυρηνικά στοιχεία (short interspersed nuclear elements (SINEs)) και στοιχεία τύπου Penelope (Penelope-like elements (PLE)). Στο "Dictyostelium discoideum", υπάρχουν και άλλα στοιχεία τύπου DIRS που ανήκουν σε μη LTR. Τα μη LTRs εξαπλώνονται ευρέως στα ευκαρυωτικά γονιδιώματα.<ref>{{cite journal | vauthors = Richard GF, Kerrest A, Dujon B | title = Comparative genomics and molecular dynamics of DNA repeats in eukaryotes | journal = Microbiology and Molecular Biology Reviews | volume = 72 | issue = 4 | pages = 686–727 | date = December 2008 | pmid = 19052325 | pmc = 2593564 | doi = 10.1128/MMBR.00011-08 }}</ref> |
|||
Τα μακρά διάσπαρτα στοιχεία (Long interspersed elements LINEs) κωδικοποιούν γονίδια για την αντίστροφη μεταγραφάση και την ενδονουκλεάση, καθιστώντας τα αυτόνομα μεταθετά στοιχεία. Το ανθρώπινο γονιδίωμα έχει περίπου 500.000 LINEs, καταλαμβάνοντας περίπου το 17% του γονιδιώματος.<ref>{{cite journal | vauthors = Cordaux R, Batzer MA | title = The impact of retrotransposons on human genome evolution | journal = Nature Reviews. Genetics | volume = 10 | issue = 10 | pages = 691–703 | date = October 2009 | pmid = 19763152 | pmc = 2884099 | doi = 10.1038/nrg2640 }}</ref> |
|||
Τα βραχέα διάσπαρτα στοιχεία (Short interspersed elements (SINEs)) είναι συνήθως μικρότερα από 500 ζεύγη βάσεων και δεν είναι αυτόνομα, επομένως βασίζονται στις πρωτεΐνες που κωδικοποιούνται από τις LINE για μετάθεση.<ref>{{cite journal | vauthors = Han JS, Boeke JD | title = LINE-1 retrotransposons: modulators of quantity and quality of mammalian gene expression? | journal = BioEssays | volume = 27 | issue = 8 | pages = 775–84 | date = August 2005 | pmid = 16015595 | doi = 10.1002/bies.20257 | s2cid = 26424042 }}</ref> Το στοιχείο Alu είναι το πιο κοινό SINE που βρίσκεται στα πρωτεύοντα. Είναι περίπου 350 ζεύγη βάσεων και καταλαμβάνει περίπου το 11% του ανθρώπινου γονιδιώματος με περίπου 1.500.000 αντίγραφα.<ref name="Kazazian 1626–1632"/> |
|||
===== Μεταθετόνια DNA ===== |
|||
Τα μεταθετόνια DNA κωδικοποιούν ένα ένζυμο μεταθετάσης μεταξύ ανεστραμμένων τελικών επαναλήψεων. Όταν εκφράζεται, η μεταθετάση αναγνωρίζει τις ακραίες ανεστραμμένες επαναλήψεις που πλαισιώνουν το μεταθετόνιο και καταλύει την αποκοπή και την επανεισαγωγή του σε μια νέα θέση.<ref name="Padeken" /> Αυτός ο μηχανισμός αποκοπής και επικόλλησης επανασυνδέει συνήθως τα μεταθετόνια κοντά στην αρχική τους θέση (εντός 100kb).<ref name="Kazazian 1626–1632"/> Τα μεταθετόνια DNA βρίσκονται σε βακτήρια και αποτελούν το 3% του ανθρώπινου γονιδιώματος και το 12% του γονιδιώματος του στρογγυλού σκουληκιού "Caenorhabditis elegans".<ref name="Kazazian 1626–1632"/> |
|||
== Μέγεθος γονιδιώματος == |
|||
[[File:Genome_size_vs_protein_count.svg|thumbnail|Λογαριθμική ως προς λογαριθμική γραφική παράσταση του συνολικού αριθμού σχολιασμένων πρωτεϊνών σε γονιδιώματα που υποβλήθηκαν στη ως συνάρτηση του μεγέθους του γονιδιώματος.]] |
|||
Μέγεθος γονιδιώματος είναι ο συνολικός αριθμός των ζευγών βάσεων DNA σε ένα αντίγραφο ενός απλοειδούς γονιδιώματος. Το μέγεθος του γονιδιώματος ποικίλλει ευρέως μεταξύ των ειδών. Τα ασπόνδυλα έχουν μικρά γονιδιώματα, αυτό συσχετίζεται επίσης με μικρό αριθμό μεταθετών στοιχείων. Τα ψάρια και τα αμφίβια έχουν γονιδιώματα μεσαίου μεγέθους και τα πουλιά έχουν σχετικά μικρά γονιδιώματα, αλλά έχει προταθεί ότι τα πουλιά έχασαν σημαντικό μέρος των γονιδιωμάτων τους κατά τη φάση της μετάβασης στην πτήση. Πριν από αυτήν την απώλεια, η μεθυλίωση του DNA επέτρεπε την επαρκή επέκταση του γονιδιώματος.<ref name=":0" /> |
|||
Στον άνθρωπο, το πυρηνικό γονιδίωμα περιλαμβάνει περίπου 3,2 δισεκατομμύρια νουκλεοτίδια DNA, χωρισμένα σε 24 γραμμικά μόρια, τα βραχύτερα 50.000.000 νουκλεοτίδια σε μήκος και τα μακρύτερα 260.000.000 νουκλεοτίδια, που το καθένα περιέχεται σε ένα διαφορετικό χρωμόσωμα.<ref>{{cite web|title=Human genome|url=http://www.whatisdna.net/wiki/genetic-genealogy-understanding-ancestry-dna/|access-date=19 August 2016}}</ref> |
|||
Δεν υπάρχει σαφής και συνεπής συσχέτιση μεταξύ μορφολογικής πολυπλοκότητας και μεγέθους γονιδιώματος είτε σε προκαρυωτικά είτε σε ευκαρυωτικά κύτταρα.<ref name="Lewin 2004" /><ref>{{cite journal | vauthors = Gregory TR, Nicol JA, Tamm H, Kullman B, Kullman K, Leitch IJ, Murray BG, Kapraun DF, Greilhuber J, Bennett MD | title = Eukaryotic genome size databases | journal = Nucleic Acids Research | volume = 35 | issue = Database issue | pages = D332–38 | date = January 2007 | pmid = 17090588 | pmc = 1669731 | doi = 10.1093/nar/gkl828 }}</ref> Το μέγεθος του γονιδιώματος είναι σε μεγάλο βαθμό συνάρτηση της επέκτασης και της συστολής των επαναλαμβανόμενων στοιχείων DNA. |
|||
Δεδομένου ότι τα γονιδιώματα είναι πολύ περίπλοκα, μια ερευνητική στρατηγική είναι να μειωθεί ο αριθμός των γονιδίων σε ένα γονιδίωμα στο απολύτως ελάχιστο και να διατηρηθεί ο εν λόγω οργανισμός. Γίνεται πειραματική εργασία σε ελάχιστα γονιδιώματα για μονοκύτταρους οργανισμούς, καθώς και ελάχιστα γονιδιώματα για πολυκυτταρικούς οργανισμούς. Το έργο είναι τόσο '' [[in vivo|ζωντανά]] '' όσο και '' [[in silico|υπολογιστικά]] ''.<ref>{{cite journal | vauthors = Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC | title = Essential genes of a minimal bacterium | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 103 | issue = 2 | pages = 425–30 | date = January 2006 | pmid = 16407165 | pmc = 1324956 | doi = 10.1073/pnas.0510013103 | bibcode = 2006PNAS..103..425G }} |
|||
</ref><ref>{{cite journal | vauthors = Forster AC, Church GM | title = Towards synthesis of a minimal cell | journal = Molecular Systems Biology | volume = 2 | issue = 1 | pages = 45 | date = 2006 | pmid = 16924266 | pmc = 1681520 | doi = 10.1038/msb4100090 }}</ref> |
|||
=== Μέγεθος γονιδιώματος λόγω μεταθετών === |
|||
Υπάρχουν πολλές τεράστιες διαφορές στο μέγεθος στα γονιδιώματα, που αναφέρθηκαν προηγουμένως στα πολυκύτταρα ευκαρυωτικά γονιδιώματα. Ο κύριος λόγος για τον οποίο υπάρχει τόσο μεγάλη ποικιλία μεγεθών οφείλεται στην παρουσία μεταθετών στοιχείων. Τα TE (μεταθετά στοιχεία "transposable elements") είναι γνωστό ότι συμβάλλουν σε μια σημαντική αλλαγή σε μάζα κυττάρου του DNA.<ref name=":0"/> Αυτή η διαδικασία σχετίζεται με τη μακροπρόθεσμη προσαρμογή τους στο γονιδίωμα του ξενιστή, και συνεπώς, με την επέκταση του μεγέθους του γονιδιώματος. |
|||
Εδώ είναι ένας πίνακας ορισμένων σημαντικών ή αντιπροσωπευτικών γονιδιωμάτων. |
|||
{| class="wikitable sortable"<!--Let's keep this table ordered by type and within that by size.--> |
|||
|- |
|||
!Τύπος οργανισμού |
|||
!Οργανισμός |
|||
!colspan="2"|Μέγεθος γονιδιώματος <br>(Ζεύγη βάσεων) |
|||
!Προσεγγιστικός αριθμός γονιδίων |
|||
!class="unsortable"|Σημείωση |
|||
|- |
|||
|[[Ιός]] |
|||
|Κυκλοϊός χοίρων τύπος 1 |
|||
|align="right"|1.759 |
|||
|1,8 kB |
|||
| |
|||
| Οι μικρότεροι ιοί που αντιγράφονται αυτόνομα σε [[ευκαρυωτικά κύτταρα]].<ref name=Equinexus>{{cite book |chapter-url=http://www.horizonpress.com/avir|author=Mankertz P|date=2008|chapter=Molecular Biology of Porcine Circoviruses|title=Animal Viruses: Molecular Biology|publisher=Caister Academic Press |isbn = 978-1-904455-22-6 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|[[Βακτηριοφάγος]] MS2 |
|||
|align="right"|3.569 |
|||
|3,5 kB |
|||
| |
|||
| Πρώτο αλληλουχημένο γονιδίωμα RNA<ref name=Fiers1976>{{cite journal | vauthors = Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, Min Jou W, Molemans F, Raeymaekers A, Van den Berghe A, Volckaert G, Ysebaert M | title = Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene | journal = Nature | volume = 260 | issue = 5551 | pages = 500–07 | date = April 1976 | pmid = 1264203 | doi = 10.1038/260500a0 | bibcode = 1976Natur.260..500F | s2cid = 4289674 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|SV40 |
|||
|align="right"|5.224 |
|||
|5,2 kB |
|||
| |
|||
|<ref name=Fiers1978>{{cite journal | vauthors = Fiers W, Contreras R, Haegemann G, Rogiers R, Van de Voorde A, Van Heuverswyn H, Van Herreweghe J, Volckaert G, Ysebaert M | title = Complete nucleotide sequence of SV40 DNA | journal = Nature | volume = 273 | issue = 5658 | pages = 113–20 | date = May 1978 | pmid = 205802 | doi = 10.1038/273113a0 | bibcode = 1978Natur.273..113F | s2cid = 1634424 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|Φάγος Φ-X174 |
|||
|align="right"|5.386 |
|||
|5,4 kB |
|||
| |
|||
| Πρώτο αλληλουχημένο γονιδίωμα DNA <ref name=Sanger1977>{{cite journal | vauthors = Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, Hutchison CA, Slocombe PM, Smith M | title = Nucleotide sequence of bacteriophage phi X174 DNA | journal = Nature | volume = 265 | issue = 5596 | pages = 687–95 | date = February 1977 | pmid = 870828 | doi = 10.1038/265687a0 | bibcode = 1977Natur.265..687S | s2cid = 4206886 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|[[ Ιός ανθρώπινης ανοσοανεπάρκειας|HIV]] |
|||
|align="right"|9.749 |
|||
|9,7 kB |
|||
| |
|||
|<ref>{{cite web|url=http://pathmicro.med.sc.edu/lecture/hiv9.htm |title=Virology – Human Immunodeficiency Virus And Aids, Structure: The Genome And Proteins Of HIV |publisher=Pathmicro.med.sc.edu |date=2010-07-01 |access-date=27 January 2011}}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|Φάγος λ |
|||
|align="right"|48.502 |
|||
|48,5 kB |
|||
| |
|||
| Συχνά χρησιμοποιείται ως φορέας για την κλωνοποίηση ανασυνδυασμένου DNA. |
|||
<ref>{{cite journal | vauthors = Thomason L, Court DL, Bubunenko M, Costantino N, Wilson H, Datta S, Oppenheim A | s2cid = 490362 | title = Recombineering: genetic engineering in bacteria using homologous recombination | journal = Current Protocols in Molecular Biology | volume = Chapter 1 | page = Unit 1.16 | date = April 2007 | pmid = 18265390 | doi = 10.1002/0471142727.mb0116s78 | isbn = 978-0-471-14272-0 }}</ref> |
|||
<ref>{{cite journal | vauthors = Court DL, Oppenheim AB, Adhya SL | title = A new look at bacteriophage lambda genetic networks | journal = Journal of Bacteriology | volume = 189 | issue = 2 | pages = 298–304 | date = January 2007 | pmid = 17085553 | pmc = 1797383 | doi = 10.1128/JB.01215-06 }}</ref> |
|||
<ref>{{cite journal | vauthors = Sanger F, Coulson AR, Hong GF, Hill DF, Petersen GB | title = Nucleotide sequence of bacteriophage lambda DNA | journal = Journal of Molecular Biology | volume = 162 | issue = 4 | pages = 729–73 | date = December 1982 | pmid = 6221115 | doi = 10.1016/0022-2836(82)90546-0 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|Μεγαϊός Megavirus |
|||
|align="right"|1.259.197 |
|||
|1,3 MB |
|||
| |
|||
| Μέχρι το 2013 το μεγαλύτερο γνωστό ιικό γονιδίωμα.<ref>{{cite journal | vauthors = Legendre M, Arslan D, Abergel C, Claverie JM | title = Genomics of Megavirus and the elusive fourth domain of Life | journal = Communicative & Integrative Biology | volume = 5 | issue = 1 | pages = 102–06 | date = January 2012 | pmid = 22482024 | pmc = 3291303 | doi = 10.4161/cib.18624 }}</ref> |
|||
|- |
|||
|[[Ιός]] |
|||
|''Pandoravirus salinus'' |
|||
| style="text-align:right;"|2.470.000 |
|||
|2,47 MB |
|||
| |
|||
| Το μεγαλύτερο γνωστό ιικό γονιδίωμα.<ref>{{cite journal | vauthors = Philippe N, Legendre M, Doutre G, Couté Y, Poirot O, Lescot M, Arslan D, Seltzer V, Bertaux L, Bruley C, Garin J, Claverie JM, Abergel C | s2cid = 16877147 | title = Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes | journal = Science | volume = 341 | issue = 6143 | pages = 281–86 | date = July 2013 | pmid = 23869018 | doi = 10.1126/science.1239181 | bibcode = 2013Sci...341..281P | url = https://hal-cea.archives-ouvertes.fr/cea-00862677/file/phi.pdf }}</ref> |
|||
|- |
|||
|[[Ευκαρυωτικά κύτταρα|Ευκαρυωτικό]] [[οργανίδιο]] |
|||
|Ανθρώπινο [[μιτοχόνδριο]] |
|||
|align="right"|16.569 |
|||
|16,6 kB |
|||
| |
|||
| <ref>{{cite journal | vauthors = Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG | title = Sequence and organization of the human mitochondrial genome | journal = Nature | volume = 290 | issue = 5806 | pages = 457–65 | date = April 1981 | pmid = 7219534 | doi = 10.1038/290457a0 | bibcode = 1981Natur.290..457A | s2cid = 4355527 }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Nasuia deltocephalinicola'' (strain NAS-ALF) |
|||
|align="right"|112.091 |
|||
|112 kB |
|||
|137 |
|||
| Το μικρότερο γνωστό μη-ιικό γονιδίωμα. Συμβιώτης των τζιτζικιών<ref>{{cite journal | vauthors = Bennett GM, Moran NA | title = Small, smaller, smallest: the origins and evolution of ancient dual symbioses in a Phloem-feeding insect | journal = Genome Biology and Evolution | volume = 5 | issue = 9 | pages = 1675–88 | date = 5 August 2013 | pmid = 23918810 | pmc = 3787670 | doi = 10.1093/gbe/evt118 }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Carsonella ruddii'' |
|||
|align="right"|159.662 |
|||
|160 kB |
|||
| |
|||
|Ενδοσυμβιώτης των εντόμων ψυλίδες |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Buchnera aphidicola'' |
|||
|align="right"|600.000 |
|||
|600 kB |
|||
| |
|||
|Ενδοσυμβιώτης των [[αφίδα|αφιδών]]<ref>{{cite journal | vauthors = Shigenobu S, Watanabe H, Hattori M, Sakaki Y, Ishikawa H | title = Genome sequence of the endocellular bacterial symbiont of aphids Buchnera sp. APS | journal = Nature | volume = 407 | issue = 6800 | pages = 81–86 | date = September 2000 | pmid = 10993077 | doi = 10.1038/35024074 | bibcode = 2000Natur.407...81S | doi-access = free }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Wigglesworthia glossinidia'' |
|||
|align="right"|700.000 |
|||
|700Kb |
|||
| |
|||
|Συμβιώτης στο έντερο των [[μύγα τσε-τσε|μυγών τσε-τσε]] |
|||
|- |
|||
|[[Βακτήριο]] – [[κυανοβακτήρια|κυανοβακτήριο]] |
|||
|''Prochlorococcus'' spp. (1,7 Mb) |
|||
|align="right"|1.700.000 |
|||
|1,7 MB |
|||
|1.884 |
|||
| Το μικρότερο γνωστό γονιδίωμα κυανοβακτηρίου. Ένας από τους κύριους φωτοσυνθέτες στη Γη. <ref> |
|||
{{cite journal | vauthors = Rocap G, Larimer FW, Lamerdin J, Malfatti S, Chain P, Ahlgren NA, Arellano A, Coleman M, Hauser L, Hess WR, Johnson ZI, Land M, Lindell D, Post AF, Regala W, Shah M, Shaw SL, Steglich C, Sullivan MB, Ting CS, Tolonen A, Webb EA, Zinser ER, Chisholm SW | display-authors = 6 | title = Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation | journal = Nature | volume = 424 | issue = 6952 | pages = 1042–47 | date = August 2003 | pmid = 12917642 | doi = 10.1038/nature01947 | bibcode = 2003Natur.424.1042R | s2cid = 4344597 }}</ref><ref> |
|||
{{cite journal | vauthors = Dufresne A, Salanoubat M, Partensky F, Artiguenave F, Axmann IM, Barbe V, Duprat S, Galperin MY, Koonin EV, Le Gall F, Makarova KS, Ostrowski M, Oztas S, Robert C, Rogozin IB, Scanlan DJ, Tandeau de Marsac N, Weissenbach J, Wincker P, Wolf YI, Hess WR | display-authors = 6 | title = Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 100 | issue = 17 | pages = 10020–25 | date = August 2003 | pmid = 12917486 | pmc = 187748 | doi = 10.1073/pnas.1733211100 | bibcode = 2003PNAS..10010020D }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Haemophilus influenzae'' |
|||
|align="right"| 1.830.000 |
|||
|1,8 MB |
|||
| |
|||
| Πρώτο γονιδίωμα ενός ζωντανού οργανισμού με αλληλουχία, Ιούλιος 1995<ref name=Fleichmann_1995>{{cite journal | vauthors = Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, Bult CJ, Tomb JF, Dougherty BA, Merrick JM | s2cid = 10423613 | title = Whole-genome random sequencing and assembly of Haemophilus influenzae Rd | journal = Science | volume = 269 | issue = 5223 | pages = 496–512 | date = July 1995 | pmid = 7542800 | doi = 10.1126/science.7542800 | bibcode = 1995Sci...269..496F }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''[[Εσερίχια κόλι]]'' |
|||
|align="right"|4.600.000 |
|||
|4,6 MB |
|||
|4,288 |
|||
|<ref>{{cite journal | vauthors = Blattner FR, Plunkett G, Bloch CA, Perna NT, Burland V, Riley M, Collado-Vides J, Glasner JD, Rode CK, Mayhew GF, Gregor J, Davis NW, Kirkpatrick HA, Goeden MA, Rose DJ, Mau B, Shao Y | display-authors = 6 | title = The complete genome sequence of Escherichia coli K-12 | journal = Science | volume = 277 | issue = 5331 | pages = 1453–62 | date = September 1997 | pmid = 9278503 | doi = 10.1126/science.277.5331.1453 | doi-access = free }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] – κυανοβακτήριο |
|||
|''Nostoc punctiforme'' |
|||
|align="right"|9.000.000 |
|||
|9 MB |
|||
|7.432 |
|||
|7432 ανοικτά πλαίσια ανάγνωσης<ref> |
|||
{{cite journal | vauthors = Meeks JC, Elhai J, Thiel T, Potts M, Larimer F, Lamerdin J, Predki P, Atlas R | title = An overview of the genome of Nostoc punctiforme, a multicellular, symbiotic cyanobacterium | journal = Photosynthesis Research | volume = 70 | issue = 1 | pages = 85–106 | year = 2001 | pmid = 16228364 | doi = 10.1023/A:1013840025518 | s2cid = 8752382 }}</ref> |
|||
|- |
|||
|[[Βακτήριο]] |
|||
|''Solibacter usitatus'' (στέλεχος Ellin 6076) |
|||
|align="right"|9.970.000 |
|||
|10 MB |
|||
| |
|||
|<ref>{{cite journal | vauthors = Challacombe JF, Eichorst SA, Hauser L, Land M, Xie G, Kuske CR | title = Biological consequences of ancient gene acquisition and duplication in the large genome of Candidatus Solibacter usitatus Ellin6076 | journal = PLOS ONE | volume = 6 | issue = 9 | page = e24882 | date = 15 September 2011 | pmid = 21949776 | pmc = 3174227 | doi = 10.1371/journal.pone.0024882 | editor1-last = Steinke | bibcode = 2011PLoSO...624882C | editor1-first = Dirk }}</ref> |
|||
|- |
|||
|Αμοιβαδοειδής |
|||
|''Polychaos dubium'' (''moeba" dubia'') |
|||
|align="right"|670.000.000.000 |
|||
|670 GB |
|||
| |
|||
| Μεγαλύτερο γνωστό γονιδίωμα.<ref name=Parfrey2008>{{cite journal | vauthors = Parfrey LW, Lahr DJ, Katz LA | title = The dynamic nature of eukaryotic genomes | journal = Molecular Biology and Evolution | volume = 25 | issue = 4 | pages = 787–94 | date = April 2008 | pmid = 18258610 | pmc = 2933061 | doi = 10.1093/molbev/msn032 }}</ref> (Αμφισβητούμενο)<ref name="ScienceShot: Biggest Genome Ever">[http://news.sciencemag.org/sciencenow/2010/10/scienceshot-biggest-genome-ever.html ScienceShot: Biggest Genome Ever] {{webarchive|url=https://web.archive.org/web/20101011155609/http://news.sciencemag.org/sciencenow/2010/10/scienceshot-biggest-genome-ever.html |date=11 October 2010 }}, comments: "The measurement for Amoeba dubia and other protozoa which have been reported to have very large genomes were made in the 1960s using a rough biochemical approach which is now considered to be an unreliable method for accurate genome size determinations."</ref> |
|||
|- |
|||
|[[Φυτό]] |
|||
|''Genlisea tuberosa'' |
|||
|align="right"|61.000.000 |
|||
|61 MB |
|||
| |
|||
|Το μικρότερο κατεγραμμένο γονιδίωμα ανθόφυτου, 2014.<ref name="Fleischmann 2014">{{cite journal | vauthors = Fleischmann A, Michael TP, Rivadavia F, Sousa A, Wang W, Temsch EM, Greilhuber J, Müller KF, Heubl G | title = Evolution of genome size and chromosome number in the carnivorous plant genus Genlisea (Lentibulariaceae), with a new estimate of the minimum genome size in angiosperms | journal = Annals of Botany | volume = 114 | issue = 8 | pages = 1651–63 | date = December 2014 | pmid = 25274549 | pmc = 4649684 | doi = 10.1093/aob/mcu189 }}</ref> |
|||
|- |
|||
|[[Φυτό]] |
|||
|''Arabidopsis thaliana'' |
|||
|align="right"|135.000.000<ref>{{cite web | title = Genome Assembly | url = https://www.arabidopsis.org/portals/genAnnotation/gene_structural_annotation/agicomplete.jsp | work = The Arabidopsis Information Resource (TAIR) }}</ref> |
|||
|135 MB |
|||
|27.655<ref>{{cite web|url=http://plants.ensembl.org/Arabidopsis_thaliana/Info/Annotation/|title=Details - Arabidopsis thaliana - Ensembl Genomes 40|website=plants.ensembl.org}}</ref> |
|||
|First plant genome sequenced, December 2000.<ref name="Greilhuber">{{cite journal | vauthors = Greilhuber J, Borsch T, Müller K, Worberg A, Porembski S, Barthlott W | title = Smallest angiosperm genomes found in lentibulariaceae, with chromosomes of bacterial size | journal = Plant Biology | volume = 8 | issue = 6 | pages = 770–77 | date = November 2006 | pmid = 17203433 | doi = 10.1055/s-2006-924101 }}</ref> |
|||
|- |
|||
|[[Φυτό]] |
|||
|''Populus|Populus trichocarpa'' |
|||
|align="right"|480.000.000 |
|||
|480 MB |
|||
|73.013 |
|||
|Τα πρώτα τρία γονιδιώματα που αλληλουχήθηκαν, Σεπτέμβριος 2006<ref>{{cite journal | vauthors = Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, Putnam N, Ralph S, Rombauts S, Salamov A, Schein J, Sterck L, Aerts A, Bhalerao RR, Bhalerao RP, Blaudez D, Boerjan W, Brun A, Brunner A, Busov V, Campbell M, Carlson J, Chalot M, Chapman J, Chen GL, Cooper D, Coutinho PM, Couturier J, Covert S, Cronk Q, Cunningham R, Davis J, Degroeve S, Déjardin A, Depamphilis C, Detter J, Dirks B, Dubchak I, Duplessis S, Ehlting J, Ellis B, Gendler K, Goodstein D, Gribskov M, Grimwood J, Groover A, Gunter L, Hamberger B, Heinze B, Helariutta Y, Henrissat B, Holligan D, Holt R, Huang W, Islam-Faridi N, Jones S, Jones-Rhoades M, Jorgensen R, Joshi C, Kangasjärvi J, Karlsson J, Kelleher C, Kirkpatrick R, Kirst M, Kohler A, Kalluri U, Larimer F, Leebens-Mack J, Leplé JC, Locascio P, Lou Y, Lucas S, Martin F, Montanini B, Napoli C, Nelson DR, Nelson C, Nieminen K, Nilsson O, Pereda V, Peter G, Philippe R, Pilate G, Poliakov A, Razumovskaya J, Richardson P, Rinaldi C, Ritland K, Rouzé P, Ryaboy D, Schmutz J, Schrader J, Segerman B, Shin H, Siddiqui A, Sterky F, Terry A, Tsai CJ, Uberbacher E, Unneberg P, Vahala J, Wall K, Wessler S, Yang G, Yin T, Douglas C, Marra M, Sandberg G, Van de Peer Y, Rokhsar D | display-authors = 6 | title = The genome of black cottonwood, Populus trichocarpa (Torr. & Gray) | journal = Science | volume = 313 | issue = 5793 | pages = 1596–604 | date = September 2006 | pmid = 16973872 | doi = 10.1126/science.1128691 | bibcode = 2006Sci...313.1596T | s2cid = 7717980 | url = https://digital.library.unt.edu/ark:/67531/metadc883930/m2/1/high_res_d/901819.pdf }}</ref> |
|||
|- |
|||
|[[Φυτό]] |
|||
|''Fritillaria assyriaca'' |
|||
|align="right"|130.000.000.000 |
|||
|130 GB |
|||
| |
|||
| |
|||
|- |
|||
|[[ Φυτό]] |
|||
|''Paris japonica'' (ιθαγενές της Ιαπωνίας, ωχρό πέταλο) |
|||
|align="right"|150.000.000.000 |
|||
|150 GB |
|||
| |
|||
|Το μεγαλύτερο γνωστό γονιδίωμα φυτού <ref>{{cite journal | last1 = Pellicer | first1 = Jaume | last2 = Fay | first2 = Michael F. | last3 = Leitch | first3 = Ilia J. | name-list-style = vanc |title=The largest eukaryotic genome of them all?|journal=Botanical Journal of the Linnean Society|date=15 September 2010|volume=164|issue=1|pages=10–15|doi=10.1111/j.1095-8339.2010.01072.x|doi-access=free}}</ref> |
|||
|- |
|||
|[[Φυτό]] |
|||
|''Physcomitrella patens'' |
|||
|align="right"|480.000.000 |
|||
|480 MB |
|||
| |
|||
|Πρώτο γονιδίωμα βρυοφύτου που αλληλουχήθηκε, Ιανουάριος 2008.<ref name="pmid18762443">{{cite journal | vauthors = Lang D, Zimmer AD, Rensing SA, Reski R | title = Exploring plant biodiversity: the Physcomitrella genome and beyond | journal = Trends in Plant Science | volume = 13 | issue = 10 | pages = 542–49 | date = October 2008 | pmid = 18762443 | doi = 10.1016/j.tplants.2008.07.002 }}</ref> |
|||
|- |
|||
|[[Μύκητας]] – [[ζυμομύκητες]] |
|||
|''Saccharomyces cerevisiae'' |
|||
|align="right"|12.100.000 |
|||
|12,1 MB |
|||
|6.294 |
|||
|Το πρώτο ευκαρυωτικό γονιδίωμα που αλληλουχήθηκε, 1996<ref>{{cite web|url=http://www.yeastgenome.org/ |title=Saccharomyces Genome Database |publisher=Yeastgenome.org |access-date=27 January 2011}}</ref> |
|||
|- |
|||
|[[Μύκητας]] |
|||
|''Aspergillus nidulans'' |
|||
|align="right"|30.000.000 |
|||
|30 MB |
|||
|9.541 |
|||
|<ref name = "Galagan_2005">{{cite journal | vauthors = Galagan JE, Calvo SE, Cuomo C, Ma LJ, Wortman JR, Batzoglou S, Lee SI, Baştürkmen M, Spevak CC, Clutterbuck J, Kapitonov V, Jurka J, Scazzocchio C, Farman M, Butler J, Purcell S, Harris S, Braus GH, Draht O, Busch S, D'Enfert C, Bouchier C, Goldman GH, Bell-Pedersen D, Griffiths-Jones S, Doonan JH, Yu J, Vienken K, Pain A, Freitag M, Selker EU, Archer DB, Peñalva MA, Oakley BR, Momany M, Tanaka T, Kumagai T, Asai K, Machida M, Nierman WC, Denning DW, Caddick M, Hynes M, Paoletti M, Fischer R, Miller B, Dyer P, Sachs MS, Osmani SA, Birren BW | display-authors = 6 | title = Sequencing of Aspergillus nidulans and comparative analysis with A. fumigatus and A. oryzae | journal = Nature | volume = 438 | issue = 7071 | pages = 1105–15 | date = December 2005 | pmid = 16372000 | doi = 10.1038/nature04341 | bibcode = 2005Natur.438.1105G | doi-access = free }}</ref> |
|||
|- |
|||
|[[Νηματώδη|Νηματώδη]] |
|||
|''Pratylenchus coffeae'' |
|||
|align="right"|20.000.000 |
|||
|20 MB |
|||
| |
|||
|<ref>{{cite journal | vauthors = Leroy S, Bouamer S, Morand S, Fargette M | year = 2007 | title = Genome size of plant-parasitic nematodes | journal = Nematology | volume = 9 | issue = 3| pages = 449–50 | doi=10.1163/156854107781352089}}</ref> Ελάχιστο γνωστό γονιδίωμα ζώου<ref>{{cite web | vauthors = Gregory TR |date=2005 |title=Animal Genome Size Database |publisher= Gregory, T.R. (2016). Animal Genome Size Database. | url=http://www.genomesize.com/statistics.php?stats=entire#stats_top}}</ref> |
|||
|- |
|||
|[[Νηματώδη|Νηματώδες]] |
|||
|''Caenorhabditis elegans'' |
|||
|align="right"|100.300.000 |
|||
|100 MB |
|||
|19.000 |
|||
|Πρώτο πολυκύτταρο γονιδίωμα ζώου που αλληλουχήθηκε, Δεκέμβριος 1998<ref>{{cite journal | author = The ''C. elegans'' Sequencing Consortium | s2cid = 16873716 | title = Genome sequence of the nematode C. elegans: a platform for investigating biology | journal = Science | volume = 282 | issue = 5396 | pages = 2012–18 | date = December 1998 | pmid = 9851916 | doi = 10.1126/science.282.5396.2012 | bibcode = 1998Sci...282.2012. }}</ref> |
|||
|- |
|||
|[[Έντομα|Έντομο]] |
|||
|''Drosophila melanogaster'' (δροσόφιλα) |
|||
|align="right"|175.000.000 |
|||
|175 MB |
|||
|13.600 |
|||
|Διακύμανση μεγέθους με βάση το στέλεχος (175-180Mb; το τυπικό ''y w'' στέλεχος είναι 175Mb)<ref name="Adams_2000">{{cite journal | vauthors = Ellis LL, Huang W, Quinn AM, Ahuja A, Alfrejd B, Gomez FE, Hjelmen CE, Moore KL, Mackay TF, Johnston JS, Tarone AM | title = Intrapopulation genome size variation in D. melanogaster reflects life history variation and plasticity | journal = PLOS Genetics | volume = 10 | issue = 7 | page = e1004522 | date = July 2014 | pmid = 25057905 | pmc = 4109859 | doi = 10.1371/journal.pgen.1004522 }}</ref> |
|||
|- |
|||
|[[Έντομα|Έντομο]] |
|||
|''Apis mellifera'' (μέλισσα) |
|||
|align="right"|236.000.000 |
|||
|236 MB |
|||
|10.157 |
|||
|<ref>{{cite journal | author = Honeybee Genome Sequencing Consortium | title = Insights into social insects from the genome of the honeybee Apis mellifera | journal = Nature | volume = 443 | issue = 7114 | pages = 931–49 | date = October 2006 | pmid = 17073008 | pmc = 2048586 | doi = 10.1038/nature05260 | bibcode = 2006Natur.443..931T }}</ref> |
|||
|- |
|||
|[[Έντομα|Έντομο]] |
|||
|''Bombyx mori'' (μεταξοσκώληκας) |
|||
|align="right"|432.000.000 |
|||
|432 MB |
|||
|14.623 |
|||
|14.623 προβλεπόμενα γονίδια<ref name="Bombyxgenome">{{cite journal | title = The genome of a lepidopteran model insect, the silkworm Bombyx mori | journal = Insect Biochemistry and Molecular Biology | volume = 38 | issue = 12 | pages = 1036–45 | date = December 2008 | pmid = 19121390 | doi = 10.1016/j.ibmb.2008.11.004 | last1 = The International Silkworm Genome }}</ref> |
|||
|- |
|||
|[[Έντομα|Έντομο]] |
|||
|''Solenopsis invicta'' (μηρμύγκι της φωτιάς) |
|||
|align="right"|480.000.000 |
|||
|480 MB |
|||
|16.569 |
|||
|<ref name=Wurm_2011>{{cite journal | vauthors = Wurm Y, Wang J, Riba-Grognuz O, Corona M, Nygaard S, Hunt BG, Ingram KK, Falquet L, Nipitwattanaphon M, Gotzek D, Dijkstra MB, Oettler J, Comtesse F, Shih CJ, Wu WJ, Yang CC, Thomas J, Beaudoing E, Pradervand S, Flegel V, Cook ED, Fabbretti R, Stockinger H, Long L, Farmerie WG, Oakey J, Boomsma JJ, Pamilo P, Yi SV, Heinze J, Goodisman MA, Farinelli L, Harshman K, Hulo N, Cerutti L, Xenarios I, Shoemaker D, Keller L | display-authors = 6 | title = The genome of the fire ant Solenopsis invicta | journal = Proceedings of the National Academy of Sciences of the United States of America | volume = 108 | issue = 14 | pages = 5679–84 | date = April 2011 | pmid = 21282665 | pmc = 3078418 | doi = 10.1073/pnas.1009690108 | bibcode = 2011PNAS..108.5679W }}</ref> |
|||
|- |
|||
|[[Θηλαστικό]] |
|||
|''Mus musculus'' |
|||
|align="right"|2.700.000.000 |
|||
|2,7 GB |
|||
|20.210 |
|||
|<ref>{{cite journal | vauthors = Church DM, Goodstadt L, Hillier LW, Zody MC, Goldstein S, She X, Bult CJ, Agarwala R, Cherry JL, DiCuccio M, Hlavina W, Kapustin Y, Meric P, Maglott D, Birtle Z, Marques AC, Graves T, Zhou S, Teague B, Potamousis K, Churas C, Place M, Herschleb J, Runnheim R, Forrest D, Amos-Landgraf J, Schwartz DC, Cheng Z, Lindblad-Toh K, Eichler EE, Ponting CP | display-authors = 6 | title = Lineage-specific biology revealed by a finished genome assembly of the mouse | journal = PLOS Biology | volume = 7 | issue = 5 | page = e1000112 | date = May 2009 | pmid = 19468303 | pmc = 2680341 | doi = 10.1371/journal.pbio.1000112 | editor1-last = Roberts | editor1-first = Richard J }}</ref> |
|||
|- |
|||
|[[Θηλαστικό]] |
|||
|''Pan paniscus'' |
|||
|align="right"|3.286.640.000 |
|||
|3.3 GB |
|||
|20.000 |
|||
|Μπονόμπο – εκτιμώμενο μέγεθος γονιδιώματος 3,29 δισεκατομμύρια ζεύγη βάσεων<ref name="Bonobo">{{cite web|url=https://www.ncbi.nlm.nih.gov/genome/10729 |title=Pan paniscus (pygmy chimpanzee) |publisher=nih.gov |access-date=30 June 2016 }}</ref> |
|||
|- |
|||
|[[Θηλαστικό]] |
|||
|''Homo sapiens'' |
|||
|align="right"|3.000.000.000 |
|||
|3 GB |
|||
|20.000 |
|||
|Εκτιμώμενο μέγεθος γονιδιώματος του ''Homo sapiens'' στα 3,2 τρισεκατομμύρια ζεύγη βάσεων το 2001<ref>{{cite journal |display-authors=etal|last1=Eric Lander |title=Initial sequencing and analysis of the human genome |journal=Nature |date=Feb 15, 2001 |volume=409 |issue=6822 |pages=860–921 |doi=10.1038/35057062|pmid=11237011 |doi-access=free }} Table 8.</ref><ref name="Homo sapien genome size">{{cite web |url=http://www.ornl.gov/sci/techresources/Human_Genome/faq/compgen.shtml#genomesize |title=Functional and Comparative Genomics Fact Sheet|publisher=Ornl.gov |archive-url=https://web.archive.org/web/20080920064848/http://www.ornl.gov/sci/techresources/Human_Genome/faq/compgen.shtml#genomesize |archive-date=2008-09-20}}</ref> |
|||
Αρχική αλληλούχιση και ανάλυση του ανθρώπινου γονιδιώματος<ref name="Analysis of human genome">{{cite journal | vauthors = Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, Smith HO, Yandell M, Evans CA, Holt RA, Gocayne JD, Amanatides P, Ballew RM, Huson DH, Wortman JR, Zhang Q, Kodira CD, Zheng XH, Chen L, Skupski M, Subramanian G, Thomas PD, Zhang J, Gabor Miklos GL, Nelson C, Broder S, Clark AG, Nadeau J, McKusick VA, Zinder N, Levine AJ, Roberts RJ, Simon M, Slayman C, Hunkapiller M, Bolanos R, Delcher A, Dew I, Fasulo D, Flanigan M, Florea L, Halpern A, Hannenhalli S, Kravitz S, Levy S, Mobarry C, Reinert K, Remington K, Abu-Threideh J, Beasley E, Biddick K, Bonazzi V, Brandon R, Cargill M, Chandramouliswaran I, Charlab R, Chaturvedi K, Deng Z, Di Francesco V, Dunn P, Eilbeck K, Evangelista C, Gabrielian AE, Gan W, Ge W, Gong F, Gu Z, Guan P, Heiman TJ, Higgins ME, Ji RR, Ke Z, Ketchum KA, Lai Z, Lei Y, Li Z, Li J, Liang Y, Lin X, Lu F, Merkulov GV, Milshina N, Moore HM, Naik AK, Narayan VA, Neelam B, Nusskern D, Rusch DB, Salzberg S, Shao W, Shue B, Sun J, Wang Z, Wang A, Wang X, Wang J, Wei M, Wides R, Xiao C, Yan C, Yao A, Ye J, Zhan M, Zhang W, Zhang H, Zhao Q, Zheng L, Zhong F, Zhong W, Zhu S, Zhao S, Gilbert D, Baumhueter S, Spier G, Carter C, Cravchik A, Woodage T, Ali F, An H, Awe A, Baldwin D, Baden H, Barnstead M, Barrow I, Beeson K, Busam D, Carver A, Center A, Cheng ML, Curry L, Danaher S, Davenport L, Desilets R, Dietz S, Dodson K, Doup L, Ferriera S, Garg N, Gluecksmann A, Hart B, Haynes J, Haynes C, Heiner C, Hladun S, Hostin D, Houck J, Howland T, Ibegwam C, Johnson J, Kalush F, Kline L, Koduru S, Love A, Mann F, May D, McCawley S, McIntosh T, McMullen I, Moy M, Moy L, Murphy B, Nelson K, Pfannkoch C, Pratts E, Puri V, Qureshi H, Reardon M, Rodriguez R, Rogers YH, Romblad D, Ruhfel B, Scott R, Sitter C, Smallwood M, Stewart E, Strong R, Suh E, Thomas R, Tint NN, Tse S, Vech C, Wang G, Wetter J, Williams S, Williams M, Windsor S, Winn-Deen E, Wolfe K, Zaveri J, Zaveri K, Abril JF, Guigó R, Campbell MJ, Sjolander KV, Karlak B, Kejariwal A, Mi H, Lazareva B, Hatton T, Narechania A, Diemer K, Muruganujan A, Guo N, Sato S, Bafna V, Istrail S, Lippert R, Schwartz R, Walenz B, Yooseph S, Allen D, Basu A, Baxendale J, Blick L, Caminha M, Carnes-Stine J, Caulk P, Chiang YH, Coyne M, Dahlke C, Mays A, Dombroski M, Donnelly M, Ely D, Esparham S, Fosler C, Gire H, Glanowski S, Glasser K, Glodek A, Gorokhov M, Graham K, Gropman B, Harris M, Heil J, Henderson S, Hoover J, Jennings D, Jordan C, Jordan J, Kasha J, Kagan L, Kraft C, Levitsky A, Lewis M, Liu X, Lopez J, Ma D, Majoros W, McDaniel J, Murphy S, Newman M, Nguyen T, Nguyen N, Nodell M, Pan S, Peck J, Peterson M, Rowe W, Sanders R, Scott J, Simpson M, Smith T, Sprague A, Stockwell T, Turner R, Venter E, Wang M, Wen M, Wu D, Wu M, Xia A, Zandieh A, Zhu X | display-authors = 6 | title = The sequence of the human genome | journal = Science | volume = 291 | issue = 5507 | pages = 1304–51 | date = February 2001 | pmid = 11181995 | doi = 10.1126/science.1058040 | bibcode = 2001Sci...291.1304V | author-link1 = Craig Venter | doi-access = free }}</ref> |
|||
|- |
|||
|[[Πτηνά|Πτηνό]] |
|||
|''Gallus gallus'' |
|||
|align="right"|1.043.000.000 |
|||
|1,0 GB |
|||
|20.000 |
|||
|<ref>{{Cite journal|date=December 2004|title=Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution|journal=Nature|language=En|volume=432|issue=7018|pages=695–716|doi=10.1038/nature03154|pmid=15592404|issn=0028-0836|author1=International Chicken Genome Sequencing Consortium|bibcode=2004Natur.432..695C|doi-access=free}}</ref> |
|||
|- |
|||
|[[Ψάρι]] |
|||
|''Tetraodon nigroviridis'' (τύπος τετραοδοντιδών) |
|||
|align="right"|385.000.000 |
|||
|390 MB |
|||
| |
|||
|Το μικρότερο γνωστό γονιδίωμα σπονδυλωτού εκτιμώμενο σε 340 Mb<ref>{{cite journal | vauthors = Roest Crollius H, Jaillon O, Dasilva C, Ozouf-Costaz C, Fizames C, Fischer C, Bouneau L, Billault A, Quetier F, Saurin W, Bernot A, Weissenbach J | title = Characterization and repeat analysis of the compact genome of the freshwater pufferfish Tetraodon nigroviridis | journal = Genome Research | volume = 10 | issue = 7 | pages = 939–49 | date = July 2000 | pmid = 10899143 | pmc = 310905 | doi = 10.1101/gr.10.7.939 }}</ref><ref>{{cite journal | vauthors = Jaillon O, Aury JM, Brunet F, Petit JL, Stange-Thomann N, Mauceli E, Bouneau L, Fischer C, Ozouf-Costaz C, Bernot A, Nicaud S, Jaffe D, Fisher S, Lutfalla G, Dossat C, Segurens B, Dasilva C, Salanoubat M, Levy M, Boudet N, Castellano S, Anthouard V, Jubin C, Castelli V, Katinka M, Vacherie B, Biémont C, Skalli Z, Cattolico L, Poulain J, De Berardinis V, Cruaud C, Duprat S, Brottier P, Coutanceau JP, Gouzy J, Parra G, Lardier G, Chapple C, McKernan KJ, McEwan P, Bosak S, Kellis M, Volff JN, Guigó R, Zody MC, Mesirov J, Lindblad-Toh K, Birren B, Nusbaum C, Kahn D, Robinson-Rechavi M, Laudet V, Schachter V, Quétier F, Saurin W, Scarpelli C, Wincker P, Lander ES, Weissenbach J, Roest Crollius H | display-authors = 6 | title = Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype | journal = Nature | volume = 431 | issue = 7011 | pages = 946–57 | date = October 2004 | pmid = 15496914 | doi = 10.1038/nature03025 | bibcode = 2004Natur.431..946J | doi-access = free }}</ref> – 385 Mb.<ref>{{cite web|title=Tetraodon Project Information|url=http://www.broadinstitute.org/annotation/tetraodon/background.html|access-date=17 October 2012|url-status=dead|archive-url=https://web.archive.org/web/20120926160058/http://www.broadinstitute.org/annotation/tetraodon/background.html|archive-date=26 September 2012|df=dmy-all}}</ref> |
|||
|- |
|||
|[[Ψάρι]] |
|||
|''Protopterus aethiopicus'' (πνευμονόψαρο) |
|||
|align="right"|130.000.000.000 |
|||
|130 GB |
|||
| |
|||
|Το μεγαλύτερο γνωστό γονιδίωμα σπονδυλωτού |
|||
|} |
|||
== Γονιδιωματικές αλλοιώσεις == |
|||
Όλα τα κύτταρα ενός οργανισμού προκύπτουν από ένα μοναδικό κύτταρο, έτσι αναμένεται να έχουν ταυτόσημα γονιδιώματα. Ωστόσο, σε κάποιες περιπτώσεις, προκύπτουν διαφορές. Τόσο η διαδικασία αντιγραφής του DNA κατά τη διαίρεση του κυττάρου όσο και η έκθεση σε περιβαλλοντικά μεταλλαξιογόνα μπορούν να έχουν ως αποτέλεσμα μεταλλάξεις σε σωματικά κύτταρα. Σε κάποιες περιπτώσεις, τέτοιες μεταλλάξεις οδηγούν σε καρκίνο, επειδή προκαλούν διαίρεση των κυττάρων και εισβάλλουν στους περιβάλλοντες ιστούς.<ref>{{cite journal | vauthors = Martincorena I, Campbell PJ | title = Somatic mutation in cancer and normal cells | journal = Science | volume = 349 | issue = 6255 | pages = 1483–89 | date = September 2015 | pmid = 26404825 | doi = 10.1126/science.aab4082 | bibcode = 2015Sci...349.1483M | s2cid = 13945473 }}</ref> Σε συγκεκριμένα λεμφοκύτταρα στο ανθρώπινο ανοσοποιητικό σύστημα, ο [[ανασυνδυασμός V(D)J]] δημιουργεί διαφορετικές γονιδιωματικές αλληλουχίες τέτοιες που κάθε κύτταρο παράγει ένα μοναδικό αντίσωμα ή υποδοχείς κυττάρων Τ. |
|||
Κατά τη [[μείωση]], τα διπλοειδή κύτταρα διαιρούνται δύο φορές για να παράξουν απλοειδή γαμετικά κύτταρα. Κατά τη διάρκεια αυτής της διαδικασίας, ο ανασυνδυασμός καταλήγει σε αναδιάταξη του γενετικού υλικού από τα ομόλογα χρωμοσώματα, έτσι ώστε κάθε γαμέτης να έχει μοναδικό γονιδίωμα. |
|||
===Επαναπρογραμματισμός ολόκληρου γονιδιώματος=== |
|||
Ο επαναπρογραμματισμός ολόκληρου του γονιδιώματος σε γεννητικά κύτταρα ποντικιών περιλαμβάνει σβήσιμο [[επιγενετική|επιγενετικού]] αποτυπώματος που οδηγεί σε παντοδυναμία. Ο επαναπρογραμματισμός διευκολύνεται από ενεργή απομεθυλίωση του DNA, μια διαδικασίας που απαιτεί την επιδιόρθωση με εκτομή βάσης της οδού του DNA.<ref name="pmid20595612">{{cite journal |vauthors=Hajkova P, Jeffries SJ, Lee C, Miller N, Jackson SP, Surani MA |title=Genome-wide reprogramming in the mouse germ line entails the base excision repair pathway |journal=Science |volume=329 |issue=5987 |pages=78–82 |date=July 2010 |pmid=20595612 |pmc=3863715 |doi=10.1126/science.1187945 |bibcode=2010Sci...329...78H }}</ref> Αυτή η οδός χρησιμοποιείται στο σβήσιμο της μεθυλίωσης CpG (5-μεθυλοκυτοσίνη 5mC) σε αρχέγονα γεννητικά κύτταρα. Το σβήσιμο της 5mC συμβαίνει μέσω της μετατροπής σε 5-υδροξυλμεθυλοκυτοσίνης (5hmC) που καθοδηγείται από υψηλά επίπεδα ενζύμων της δέκα-ένδεκα διοξυγονάσης TET1 και TET2.<ref name="pmid23223451">{{cite journal |vauthors=Hackett JA, Sengupta R, Zylicz JJ, Murakami K, Lee C, Down TA, Surani MA |title=Germline DNA demethylation dynamics and imprint erasure through 5-hydroxymethylcytosine |journal=Science |volume=339 |issue=6118 |pages=448–52 |date=January 2013 |pmid=23223451 |pmc=3847602 |doi=10.1126/science.1229277 |bibcode=2013Sci...339..448H }}</ref> |
|||
== Εξέλιξη γονιδιώματος == |
|||
Τα γονιδιώματα είναι περισσότερα από το άθροισμα [[γονίδιο|γονιδίων]] ενός οργανισμού και έχουν χαρακτηριστικά που μπορούν να [[μέτρηση|μετρηθούν]] και να μελετηθούν χωρίς παραπομπή σε λεπτομέρειες οποιονδήποτε συγκεκριμένων γονιδίων και των προϊόντων τους. Οι ερευνητές συγκρίνουν χαρακτηριστικά όπως [[καρυότυπος]] (αριθμός χρωμοσωμάτων)), μέγεθος γονιδίου, σειρά γονιδίου, προτίμηση χρήσης κωδικονίων (codon usage bias), και περιεχόμενο γουανίνης-κυτοσίνης (GC-content) για να προσδιορίσουν ποιοι μηχανισμοί θα μπορούσαν να έχουν παράξει τη μεγάλη ποικιλία των γονιδιωμάτων που υπάρχουν σήμερα. |
|||
Ο διπλασιασμός γονιδίου παίζουν σημαντικό ρόλο στη διαμόρφωση του γονιδιώματος. Ο διπλασιασμός μπορεί να εκτείνεται από την επέκταση των βραχέων διαδοχοκών επαναλήψεων (short tandem repeats), τον διπλασιασμό συστοιχίας γονιδίων και μέχρι τον διπλασιασμό ολόκληρων χρωμοσωμάτων ή ακόμα ολόκληρων γονιδιωμάτων. Τέτοιοι διπλασιασμοί είναι προφανώς βασικοί στη δημιουργία καινοτομιών. |
|||
H Οριζόντια μεταφορά γονιδίου επικαλείται για να εξηγήσει πώς υπάρχει συχνά μια ακραία ομοιότητα μεταξύ μικρών τμημάτων των γονιδιωμάτων δύο οργανισμών που κατά τα άλλα έχουν πολύ μακρινή συγγένεια. Η οριζόντια μεταφορά γονιδίων φαίνεται να είναι κοινή μεταξύ πολλών [[μικροοργανισμός|μικροοργανισμών]]. Επίσης, τα [[ευκαρυωτικά κύτταρα]] φαίνεται να έχουν βιώσει μεταφορά κάποιου γενετικού υλικού από τα [[χλωροπλάστης|χλωροπλαστικά]] και τα [[μιτοχόνδριο|μιτοχονδριακά]] γονιδιώματά τους στα πυρηνικά χρωμοσώματά τους. Τα πρόσφατα εμπειρικά δεδομένα προτείνουν ότι ένας σημαντικός ρόλος ιών και υποιικών δικτύων RNA αντιπροσωπεύει έναν κύριο οδηγό ρόλο στη δημιουργία γενετικής καινοτομίας και φυσικής επεξεργασίας γονιδιώματος. |
|||
Ειδικότερα, χρήση του όρου αυτού γίνεται σχεδόν σε όλες τις περιπτώσεις αναφορών επί των [[γονίδιο|γονιδίων]] και των μηχανισμών αυτών. |
|||
Ενώ συγκεκριμένα για τους ευκαρυωτικούς οργανισμούς, ο όρος αναφέρεται στο γενετικό υλικό του πυρήνα. |
|||
<ref>{{cite web |
|||
|author = Δρ Βασιλική Αλεπόρου-Μαρίνου, Δρ Αλέξανδρος Αργυροκαστρίτης, Δρ Αικατερίνη Κομητοπούλου, Δρ Περικλής Πιαλόγλου, Βασιλική Σγουρίτσα. |
|||
|title = ΒΙΟΛΟΓΙΑ θετικής κατεύθυνσης Γ' τάξης Γενικού Λυκείου 1ο Κεφάλαιο |
|||
|url = http://ebooks.edu.gr/modules/ebook/show.php/DSGL-C112/52/390,1500/ |
|||
|publisher = Οργανισμός Εκδόσεων Διδακτικών Βιβλίων. |
|||
|date = |
|||
|accessdate = 8 Νοε. 2015 |
|||
|archive-date = 2016-03-05 |
|||
|archive-url = https://web.archive.org/web/20160305230033/http://ebooks.edu.gr/modules/ebook/show.php/DSGL-C112/52/390,1500/ |
|||
|url-status = dead |
|||
}}</ref> |
|||
== Παραπομπές == |
== Παραπομπές == |
||
{{reflist}} |
|||
<references /> |
|||
== Παραπέρα μελέτη == |
|||
==Βιβλιογραφία== |
|||
* {{cite book | vauthors = Benfey P, Protopapas AD|title=Essentials of Genomics|publisher=Prentice Hall|date=2004}} |
|||
* Βουτσινάς, Γεράσιμος: «Η πολυπλοκότητα του γονιδιώματος», ''[[Περισκόπιο της Επιστήμης]]'', Φεβρουάριος 1997, σελ. 12 |
|||
* {{cite book | last1=Brown|first1=Terence A. |title=Genomes 2|publisher=Bios Scientific Publishers|location=Oxford|date=2002|isbn=978-1-85996-029-5 }} |
|||
* Benfey, P., Protopapas, A.D.: ''Essentials of Genomics'', Prentice Hall, 2004. |
|||
* {{cite book|last1=Gibson|first1=Greg|last2=Muse|first2=Spencer V.|title=A Primer of Genome Science|edition=Second|publisher=Sinauer Assoc|location=Sunderland, Mass|date=2004|isbn=978-0-87893-234-4|url-access=registration|url=https://archive.org/details/primerofgenomesc00greg}} |
|||
* {{cite book|last=Gregory|first=T. Ryan |title=The Evolution of the Genome|publisher=Elsevier|date=2005|isbn=978-0-12-301463-4|title-link=The Evolution of the Genome }} |
|||
* {{cite book|last=Reece|first=Richard J.|title=Analysis of Genes and Genomes|publisher=John Wiley & Sons|location=Chichester|date=2004|isbn=978-0-470-84379-6}} |
|||
* {{cite book|last1=Saccone|first1=Cecilia|last2=Pesole | first2 = Graziano|title=Handbook of Comparative Genomics|publisher=John Wiley & Sons|location=Chichester|date=2003|isbn=978-0-471-39128-9 }} |
|||
* {{cite journal | vauthors = Werner E | title = In silico multicellular systems biology and minimal genomes | journal = Drug Discovery Today | volume = 8 | issue = 24 | pages = 1121–27 | date = December 2003 | pmid = 14678738 | doi = 10.1016/S1359-6446(03)02918-0 }} |
|||
== Εξωτερικοί σύνδεσμοι == |
|||
{{wikiquote}} |
|||
* [http://genome.ucsc.edu UCSC Genome Browser] – view the genome and annotations for more than 80 organisms. |
|||
* [https://web.archive.org/web/20130809102814/http://www.genomecenter.howard.edu/ genomecenter.howard.edu] |
|||
* [https://web.archive.org/web/20100609223959/http://learn.genetics.utah.edu/content/begin/dna/builddna/ Build a DNA Molecule] |
|||
* [http://www.genomenewsnetwork.org/articles/02_01/Sizing_genomes.shtml Some comparative genome sizes] |
|||
* [http://www.dnai.org/ DNA Interactive: The History of DNA Science] |
|||
* [http://www.dnaftb.org/ DNA From The Beginning] |
|||
* [http://www.genome.gov/10001772 All About The Human Genome Project]—from Genome.gov |
|||
* [http://www.genomesize.com/ Animal genome size database] |
|||
* [https://web.archive.org/web/20050901105257/http://www.rbgkew.org.uk/cval/homepage.html Plant genome size database] |
|||
* [http://www.genomesonline.org/ GOLD:Genomes OnLine Database] |
|||
* [http://www.genomenewsnetwork.org/ The Genome News Network] |
|||
* [https://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=genomeprj NCBI Entrez Genome Project database] |
|||
* [https://www.ncbi.nlm.nih.gov/About/primer/genetics_genome.html NCBI Genome Primer] |
|||
* [https://www.genecards.org/ GeneCards]—an integrated database of human genes |
|||
* [http://news.bbc.co.uk/1/hi/sci/tech/4994088.stm BBC News – Final genome 'chapter' published] |
|||
* [http://img.jgi.doe.gov/ IMG] (The Integrated Microbial Genomes system)—for genome analysis by the DOE-JGI |
|||
* [https://web.archive.org/web/20120303111440/http://www.geknome.com/ GeKnome Technologies Next-Gen Sequencing Data Analysis]—next-generation sequencing data analysis for [[Illumina (company)|Illumina]] and [[454 Life Sciences|454]] Service from GeKnome Technologies. |
|||
{{Authority control}} |
{{Authority control}} |
||
{{Γενετική-επέκταση}} |
|||
[[Κατηγορία: |
[[Κατηγορία:Γενετική]] |
||
Έκδοση από την 19:18, 28 Ιουλίου 2021
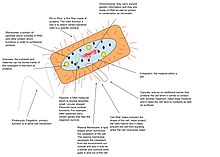

Στα πεδία μοριακή βιολογία και γενετική, γονιδίωμα είναι όλο το γενετικό υλικό ενός οργανισμού.[1] Αποτελείται από DNA (ή RNA σε ιούς RNA). Το γονιδίωμα περιλαμβάνει τόσο τα γονίδια (τις κωδικοποιητικές περιοχές) όσο και το μη κωδικοποιητικό DNA,[2] καθώς και το μιτοχονδριακό DNA[3] και DNA των χλωροπλαστών. Η μελέτη του γονιδιώματος ονομάζεται γονιδιωματική.
Προέλευση του όρου
Ο όρος γονιδίωμα δημιουργήθηκε το 1920 από τον Hans Winkler, [4] καθηγητή της βοτανικής στο Πανεπιστήμιο του Αμβούργου, Γερμανία. Το λεξικό Όξφορντ προτείνει το όνομα ως μείξη των λέξεων γονίδιο και χρωμόσωμα[5]
Αλληλούχιση και χαρτογράφηση
Η αλληλουχία του γονιδιώματος είναι ο πλήρης κατάλογος των νουκλεοτιδίων (A, C, G και T για τα γονιδιώματα DNA) που αποτελούν όλα τα χρωμοσώματα ενός ατόμου ή ενός είδους. Σε ένα είδος, η συντριπτική πλειονότητα των νουκλεοτιδίων είναι ταυτόσημη μεταξύ των ατόμων, αλλά η αλληλούχιση πολλών ατόμων είναι απαραίτητη για την κατανόηση της γενετικής ποικιλομορφίας.

Το 1976, ο Walter Fiers στο Πανεπιστήμιο της Γάνδης (Βέλγιο) ήταν ο πρώτος που καθιέρωσε την πλήρη νουκλεοτιδική αλληλουχία ενός ιικού γονιδιώματος RNA (βακτηριοφάγος MS2). Το επόμενο έτος, ο Fred Sanger ολοκλήρωσε την πρώτη αλληλουχία γονιδιώματος DNA : Φάγος Φ-Χ174, από 5386 ζεύγη βάσεων.[6] Οι πρώτες πλήρεις αλληλουχίες γονιδιώματος μεταξύ των τριών επικρατειών της ζωής κυκλοφόρησαν σε σύντομο χρονικό διάστημα κατά τα μέσα της δεκαετίας του 1990: Το πρώτο βακτηριακό γονιδίωμα το οποίο αλληλουχήθηκε ήταν εκείνο της Haemophilus influenzae, που ολοκληρώθηκε από μια ομάδα στο "The Institute for Genomic Research" το 1995. Λίγους μήνες αργότερα, ολοκληρώθηκε το πρώτο ευκαρυωτικό γονιδίωμα, με αλληλουχίες των 16 χρωμοσωμάτων της εκβλάστησης ζύμης Saccharomyces cerevisiae που δημοσιεύθηκε ως αποτέλεσμα μιας ευρωπαϊκής προσπάθειας που ξεκίνησε το στα μέσα της δεκαετίας του 1980. Η πρώτη ακολουθία γονιδιώματος για ένα αρχαίο, το Methanococcus jannaschii, ολοκληρώθηκε το 1996, ξανά από το Ινστιτούτο Γονιδιωματικής Έρευνας. Η ανάπτυξη νέων τεχνολογιών έχει κάνει την αλληλούχιση γονιδιώματος πολύ πιο φτηνή και πιο εύκολη και ο αριθμός των πλήρων αλληλουχιών γονιδιώματος αυξάνεται ραγδαία. Τα Εθνικά Ινστιτούτα Υγείας (ΗΠΑ) διατηρούν μία από τις πολλές ολοκληρωμένες βάσεις δεδομένων γονιδιωματικών πληροφοριών.[7]
Μεταξύ των χιλιάδων ολοκληρωμένων έργων αλληλούχισης του γονιδιώματος περιλαμβάνονται εκείνα για το ρύζι, το ποντίκι, το φυτό Arabidopsis thaliana, τις τετραοδοντίδες και τα βακτήρια Εσερίχια κόλι. Τον Δεκέμβριο του 2013, οι επιστήμονες αλληλούχισαν για πρώτη φορά ολόκληρο το γονιδίωμα ενός Νεάντερταλ, ενός εξαφανισμένου είδους ανθρώπων. Το γονιδίωμα εξήχθη από το οστό δακτύλου ενός Νεάντερταλ περίπου 130.000 ετών που βρέθηκε σε Σπήλαιο της Σιβηρίας.[8][9]
Νέες τεχνολογίες αλληλούχισης, όπως μαζική παράλληλη αλληλούχιση έχουν επίσης ανοίξει την προοπτική της αλληλούχισης του προσωπικού γονιδιώματος ως διαγνωστικού εργαλείου, όπου πρωτοστάτησε η Manteia Predictive Medicine. Ένα σημαντικό βήμα προς αυτόν τον στόχο ήταν η ολοκλήρωση το 2007 του πλήρους γονιδιώματος του James D. Watson, ενός από τα άτομα που ανακάλυψαν τη δομή του DNA.[10]
Ενώ μια αλληλουχία γονιδιώματος παραθέτει τη σειρά κάθε βάσης DNA σε ένα γονιδίωμα, ένας χάρτης γονιδιώματος προσδιορίζει τα σημεία αναφοράς. Ένας χάρτης γονιδιώματος είναι λιγότερο λεπτομερής από μια αλληλουχία γονιδιώματος και βοηθά στην πλοήγηση γύρω από το γονιδίωμα. Το έργο του ανθρωπίνου γονιδιώματος οργανώθηκε σε χάρτη και σε αλληλούχιση του ανθρωπίνου γονιδιώματος. Ένα βασικό βήμα στο έργο ήταν η έκδοση ενός λεπτομερούς γονιδιωματικού χάρτη από τον Jean Weissenbach και την ομάδα του στο Genoscope στο Παρίσι.[11][12]
Οι αλληλουχίες του γονιδιώματος αναφοράς και οι χάρτες συνεχίζουν να ενημερώνονται, αφαιρώντας τα λάθη και αποσαφηνίζοντας περιοχές με υψηλή πολυπλοκότητα αλληλομόρφων.[13] Το μειωμένο κόστος της γονιδιωματικής χαρτογράφησης επέτρεψε στους γενεαλογικούς ιστότοπους να την προσφέρουν ως υπηρεσία,[14] στο βαθμό που κάποιος μπορεί να υποβάλει το γονιδίωμα κάποιου σε πληθοπορισμικά εγχειρήματα όπως DNA.LAND στο Κέντρο Γονιδιώματος της Νέας Υόρκης,[15] ένα παράδειγμα τόσο οικονομιών κλίμακας όσο και επιστήμης των πολιτών.[16]
Ιικά γονιδιώματα
Τα ιικά γονιδιώματα μπορεί να αποτελούνται είτε από RNA ή από DNA. Τα γονιδιώματα των ιών RNA μπορεί να είναι είτε μονόκλωνα, είτε δίκλωνα RNA και μπορεί να περιέχουν ένα ή περισσότερα ξεχωριστά μόρια RNA (τμήματα: μονομερή ή πολυμερή γονιδιώματος). Οι ιοί DNA μπορούν να έχουν είτε μονόκλωνα είτε δίκλωνα γονιδιώματα. Τα περισσότερα γονιδιώματα ιών DNA αποτελούνται από ένα μόνο γραμμικό μόριο DNA, αλλά μερικά αποτελούνται από ένα μόριο κυκλικού DNA.[17] Υπάρχουν επίσης ιικά RNA που είναι μονόκλωνα RNA και χρησιμεύουν ως πρότυπο για τη σύνθεση mRNA[18] και μονόκλωνα RNA που χρησιμεύουν ως πρότυπα για τη σύνθεση DNA.
Iικός φάκελος[19] είναι ένα εξωτερικό στρώμα μεμβράνης που χρησιμοποιούν τα ιικά γονιδιώματα για να εισέλθουν στο κύτταρο του ξενιστή. Ορισμένες από τις κατηγορίες ιικού DNA και RNA έχουν ιικό φάκελο, ενώ άλλες όχι.
Προκαρυωτικά γονιδιώματα
Τα προκαρυωτικά και τα ευκαρυωτικά κύτταρα έχουν γονιδιώματα DNA. Τα αρχαία και τα περισσότερα βακτήρια έχουν ένα μοναδικό κυκλικό χρωμόσωμα.[20] Ωστόσο, ορισμένα βακτηριακά είδη έχουν γραμμικά ή πολλαπλά χρωμοσώματα.[21][22] Εάν το DNA αντιγράφεται γρηγορότερα από ό,τι διαιρούνται τα βακτηριακά κύτταρα, μπορούν να υπάρχουν πολλαπλά αντίγραφα του χρωμοσώματος σε ένα μοναδικό κύτταρο και εάν τα κύτταρα διαιρούνται γρηγορότερα από ό,τι μπορεί να αναπαραχθεί το DNA, ξεκινά πολλαπλή αναπαραγωγή του χρωμοσώματος πριν συμβεί η διαίρεση, επιτρέποντας στα θυγατρικά κύτταρα να κληρονομήσουν πλήρη γονιδιώματα και ήδη μερικώς αναπαραγόμενα χρωμοσώματα. Τα περισσότερα προκαρυωτικά κύτταρα έχουν πολύ λίγο επαναλαμβανόμενο DNA στα γονιδιώματά τους.[23] Ωστόσο, ορισμένα συμβιωτικά βακτήρια (π.χ. Serratia symbiotica) έχουν μειωμένα γονιδιώματα και ένα υψηλό κλάσμα ψευδογονιδίων: μόνο το περίπου 40% του DNA τους κωδικοποιεί πρωτεΐνες[24][25]
Μερικά βακτήρια έχουν βοηθητικό γενετικό υλικό, επίσης μέρος του γονιδιώματός τους, το οποίο μεταφέρεται σε πλασμίδια. Για αυτό, η λέξη γονιδίωμα δεν πρέπει να χρησιμοποιείται ως συνώνυμο του χρωμοσώματος.
Ευκαρυωτικά γονιδιώματα
Τα ευκαρυωτικά γονιδιώματα αποτελούνται από ένα ή περισσότερα γραμμικά χρωμοσώματα DNA. Ο αριθμός των χρωμοσωμάτων ποικίλλει σημαντικά από Myrmecia pilosula και Diploscapter pachys (asexual nemotode), [26] που το καθένα έχει μόνο ένα ζεύγος, σε Ophioglossum (είδος φτέρης) που έχει 720 ζεύγη. [27] Είναι εκπληκτικό το ποσό του DNA που περιέχουν τα ευκαρυωτικά γονιδιώματα σε σύγκριση με άλλα γονιδιώματα. Η ποσότητα είναι ακόμη μεγαλύτερη από ό,τι είναι απαραίτητη για κωδικοποιητικά γονίδια πρωτεϊνών DNA και μη κωδικοποιητικά γονίδια λόγω του γεγονότος ότι τα ευκαρυωτικά γονιδιώματα εμφανίζουν παραλλαγές έως και 64.000 φορές στα μεγέθη τους.[28] Ωστόσο, αυτό το ιδιαίτερο χαρακτηριστικό προκαλείται από την παρουσία επαναλαμβανόμενου DNA και μεταθετών στοιχείων (TEs).
Ένα τυπικό ανθρώπινο κύτταρο έχει δύο αντίγραφα καθένα από τα 22 ζεύγη αυτοσωμάτων, που κανονικά το καθένα κληρονομείται από κάθε γονέα, συν δύο φυλετικά χρωμοσώματα, καθιστώντας το διπλοειδές. Οι γαμέτες, όπως τα ωάρια, το σπέρμα, τα σπόρια και η γύρη, είναι απλοειδή, που σημαίνει ότι φέρουν μόνο ένα αντίγραφο κάθε χρωμοσώματος. Εκτός από τα χρωμοσώματα στον πυρήνα, οργανίδια όπως οι χλωροπλάστες και τα μιτοχόνδρια έχουν το δικό τους DNA. Τα μιτοχόνδρια μερικές φορές λέγεται ότι έχουν το δικό τους γονιδίωμα που συχνά αναφέρεται ως "μιτοχονδριακό γονιδίωμα". Το DNA που βρίσκεται μέσα στον χλωροπλάστη μπορεί να αναφέρεται ως "πλαστωμα". Όπως τα βακτήρια από τα οποία προέρχονται, τα μιτοχόνδρια και οι χλωροπλάστες έχουν κυκλικό χρωμόσωμα.
Σε αντίθεση με τα προκαρυωτικά, τα ευκαρυωτικά έχουν οργάνωση γονιδίων εξωνίων-εσωνίων που κωδικοποιούν πρωτεΐνες και μεταβλητές ποσότητες επαναλαμβανόμενου DNA. Στα θηλαστικά και τα φυτά, η πλειονότητα του γονιδιώματος αποτελείται από επαναλαμβανόμενο DNA.[29] Τα γονίδια στα ευκαρυωτικά γονιδιώματα μπορούν να περιγραφούν χρησιμοποιώντας το FINDER.[30]
Αλληλουχίες κωδικοποίησης
Οι αλληλουχίες DNA που φέρουν τις οδηγίες για την παραγωγή πρωτεϊνών αναφέρονται ως κωδικοποιητικές αλληλουχίες. Η αναλογία του γονιδιώματος που καταλαμβάνεται από κωδικοποιητικές αλληλουχίες ποικίλλει ευρέως. Ένα μεγαλύτερο γονιδίωμα δεν περιέχει απαραίτητα περισσότερα γονίδια και η αναλογία του μη επαναλαμβανόμενου DNA μειώνεται μαζί με την αύξηση του μεγέθους του γονιδιώματος σε πολύπλοκους ευκαρυώτες.[29]

Αλληλουχίες χωρίς κωδικοποίηση
Οι μη κωδικοποιητικές αλληλουχίες περιλαμβάνουν εσώνια, αλληλουχίες για μη κωδικοποιητικά RNA, ρυθμιστικές περιοχές και επαναλαμβανόμενο DNA. Οι μη κωδικοποιητικές αλληλουχίες αποτελούν το 98% του ανθρώπινου γονιδιώματος. Υπάρχουν δύο κατηγορίες επαναλαμβανόμενων DNA στο γονιδίωμα: διαδοχικές επαναλήψεις και διάσπαρτες επαναλήψεις.[31]
Διαδοχικές επαναλήψεις
Σύντομες, μη κωδικοποιητικές αλληλουχίες που επαναλαμβάνονται από την αρχή έως το τέλος καλούνται διαδοχικές επαναλήψεις. Μικροδορυφόροι αποτελούμενοι από 2-5 επαναλήψεις ζεύγους βάσεων, ενώ οι επαναλήψεις μινιδορυφόρων είναι 30-35 bp. Οι διαδοχικές επαναλήψεις αποτελούν περίπου το 4% του ανθρώπινου γονιδιώματος και το 9% του γονιδιώματος της μύγας των φρούτων.[32] Οι διαδοχικές επαναλήψεις μπορούν να είναι λειτουργικές. Για παράδειγμα, τα τελομερή αποτελούνται από την διαδοχική επανάληψη TTAGGG στα θηλαστικά και παίζουν σημαντικό ρόλο στην προστασία των άκρων του χρωμοσώματος.
Σε άλλες περιπτώσεις, οι επεκτάσεις του αριθμού των διαδοχικών επαναλήψεων σε εξώνια ή εσώνια μπορεί να προκαλέσει την ασθένεια της τρινουκλεοτιδικής επαναλαμβανόμενης διαταραχής.[33] Για παράδειγμα, το ανθρώπινο γονίδιο χαντιγκτίνη περιέχει συνήθως 6-29 διαδοχικές επαναλήψεις των νουκλεοτιδίων CAG (κωδικοποιώντας μια πολυγλουταμίνη). Η επέκταση σε πάνω από 36 επαναλήψεις οδηγεί στη νόσο του Χάντινγκτον, μια νευροεκφυλιστική ασθένεια. Είκοσι ανθρώπινες διαταραχές είναι γνωστό ότι προκύπτουν από παρόμοιες διαδοχικές επαναλαμβανόμενες επεκτάσεις σε διάφορα γονίδια. Ο μηχανισμός με τον οποίο οι πρωτεΐνες με διευρυμένες οδούς πολυγλουταμίνης προκαλούν θάνατο των νευρώνων δεν είναι πλήρως κατανοητός. Μία πιθανότητα είναι ότι οι πρωτεΐνες αποτυγχάνουν να διπλώνονται σωστά και να αποφεύγουν την αποικοδόμηση, αντί να συσσωρεύονται σε συσσωματώματα που επίσης δεσμεύουν σημαντικούς παράγοντες μεταγραφής, μεταβάλλοντας έτσι τη γονιδιακή έκφραση.[33]
Οι διαδοχικές επαναλήψεις προκαλούνται συνήθως από ολίσθηση κατά την αναπαραγωγή, άνισο επιχιασμό και μετατροπή γονιδίων.[34]
Μεταθετά στοιχεία
Τα μεταθετά στοιχεία (ΤΕ) είναι αλληλουχίες DNA με καθορισμένη δομή που μπορούν να αλλάξουν τη θέση τους στο γονιδίωμα.[32][23][35] Τα TE κατηγοριοποιούνται είτε ως ένας μηχανισμός που αναπαράγεται με αντιγραφή και επικόλληση, είτε ως μηχανισμός που μπορεί να αποκοπεί από το γονιδίωμα και να εισαχθεί σε μια νέα θέση. Στο ανθρώπινο γονιδίωμα, υπάρχουν τρεις σημαντικές τάξεις ΤΕ που αποτελούν περισσότερο από το 45% του ανθρώπινου DNA. Αυτές οι τάξεις είναι τα μακρά διασπαρμένα πυρηνικά στοιχεία (long interspersed nuclear elements ή LINEs), τα διασπαρμένα πυρηνικά στοιχεία (interspersed nuclear elements ή SINEs) και οι ενδογενείς ρετροϊοί. Αυτά τα στοιχεία έχουν μεγάλες δυνατότητες τροποποίησης του γενετικού ελέγχου σε έναν οργανισμό ξενιστή.[28]
Η κίνηση των TEs είναι η κινητήρια δύναμη της εξέλιξης του γονιδιώματος στους ευκαρυώτες, επειδή η εισαγωγή τους μπορεί να διαταράξει τις γονιδιακές λειτουργίες, ο ομόλογος ανασυνδυασμός μεταξύ των TE μπορεί να προκαλέσει διπλασιασμούς και το TE μπορεί να ανακατέψει τα εξώνια και τις ρυθμιστικές αλληλουχίες σε νέες τοποθεσίες.[36]
Ρετρομεταθετόνια (Retrotransposons)
Τα ρετρομεταθετόνια[37] βρίσκονται κυρίως σε ευκαρυώτες αλλά δεν βρίσκονται σε προκαρυώτες και τα ρετρομεταθετόνια σχηματίζουν μεγάλο μέρος των γονιδιωμάτων πολλών ευκαρυωτικών. Το ρετρομεταθετόνιο είναι ένα μεταθετό στοιχείο που μεταφέρεται μέσω ενός ενδιάμεσου RNA. Τα ρετρομεταθετόνια[38] αποτελούνται από DNA, αλλά μεταγράφονται σε RNA για τη μεταφορά, έπειτα το μεταγράφημα RNA αντιγράφεται πίσω στον σχηματισμό DNA με τη βοήθεια ενός συγκεκριμένου ενζύμου που ονομάζεται αντίστροφη μεταγραφάση. Τα ρετρομεταθετόνια που φέρουν αντίστροφη μεταγραφάση στο γονίδιο τους μπορούν να προκαλέσουν τη δική της μεταφορά, αλλά τα γονίδια που δεν έχουν την αντίστροφη μεταγραφάση πρέπει να χρησιμοποιησουν την αντίστροφη μεταγραφάση που συντίθεται από άλλο ρετρομεταθετόνιο. Τα ρετρομεταθετόνια μπορούν να μεταγραφούν σε RNA, που στη συνέχεια διπλασιάζονται σε άλλη θέση στο γονιδίωμα.[39] Τα ρετρομεταθετόνια μπορούν να χωριστούν σε μακριές ακραίες επαναλήψεις (long terminal repeats ή LTRs) και σε μη μακριές ακραίες επαναλήψεις (Non-LTRs).[36]
Οι μακριές ακραίες επαναλήψεις Long terminal repeats (LTRs) προέρχονται από αρχαίες ρετροϊικές λοιμώξεις, επομένως κωδικοποιούν πρωτεΐνες που σχετίζονται με ρετροϊικές πρωτεΐνες, συμπεριλαμβανομένων ομαδοειδικών αντιγόνων (group-specific antigen ή gag), γλυκοζαμινογλυκάνης, pol (πολυμερασών - αντίστροφη μεταγραφάση και ιντεγκράση), pro (πρωτεάση) και σε μερικές περιπτώσεις φακέλους (env) γονιδίων.[35] Αυτά τα γονίδια πλαισιώνονται από μεγάλες επαναλήψεις και στα δύο άκρα 5' και 3'. Έχει αναφερθεί ότι τα LTRs αποτελούν το μεγαλύτερο κλάσμα στα περισσότερα φυτικά γονιδιώματα και ενδέχεται να αντιπροσωπεύουν την τεράστια διακύμανση στο μέγεθος του γονιδιώματος.[40]
Οι μη μακριές ακραίες επαναλήψεις Non-long terminal repeats (Non-LTR) ταξινομούνται ως μακρά διάσπαρτα πυρηνικά στοιχεία (long interspersed nuclear elements (LINEs)), βραχέα διάσπαρτα πυρηνικά στοιχεία (short interspersed nuclear elements (SINEs)) και στοιχεία τύπου Penelope (Penelope-like elements (PLE)). Στο "Dictyostelium discoideum", υπάρχουν και άλλα στοιχεία τύπου DIRS που ανήκουν σε μη LTR. Τα μη LTRs εξαπλώνονται ευρέως στα ευκαρυωτικά γονιδιώματα.[41]
Τα μακρά διάσπαρτα στοιχεία (Long interspersed elements LINEs) κωδικοποιούν γονίδια για την αντίστροφη μεταγραφάση και την ενδονουκλεάση, καθιστώντας τα αυτόνομα μεταθετά στοιχεία. Το ανθρώπινο γονιδίωμα έχει περίπου 500.000 LINEs, καταλαμβάνοντας περίπου το 17% του γονιδιώματος.[42]
Τα βραχέα διάσπαρτα στοιχεία (Short interspersed elements (SINEs)) είναι συνήθως μικρότερα από 500 ζεύγη βάσεων και δεν είναι αυτόνομα, επομένως βασίζονται στις πρωτεΐνες που κωδικοποιούνται από τις LINE για μετάθεση.[43] Το στοιχείο Alu είναι το πιο κοινό SINE που βρίσκεται στα πρωτεύοντα. Είναι περίπου 350 ζεύγη βάσεων και καταλαμβάνει περίπου το 11% του ανθρώπινου γονιδιώματος με περίπου 1.500.000 αντίγραφα.[36]
Μεταθετόνια DNA
Τα μεταθετόνια DNA κωδικοποιούν ένα ένζυμο μεταθετάσης μεταξύ ανεστραμμένων τελικών επαναλήψεων. Όταν εκφράζεται, η μεταθετάση αναγνωρίζει τις ακραίες ανεστραμμένες επαναλήψεις που πλαισιώνουν το μεταθετόνιο και καταλύει την αποκοπή και την επανεισαγωγή του σε μια νέα θέση.[32] Αυτός ο μηχανισμός αποκοπής και επικόλλησης επανασυνδέει συνήθως τα μεταθετόνια κοντά στην αρχική τους θέση (εντός 100kb).[36] Τα μεταθετόνια DNA βρίσκονται σε βακτήρια και αποτελούν το 3% του ανθρώπινου γονιδιώματος και το 12% του γονιδιώματος του στρογγυλού σκουληκιού "Caenorhabditis elegans".[36]
Μέγεθος γονιδιώματος

Μέγεθος γονιδιώματος είναι ο συνολικός αριθμός των ζευγών βάσεων DNA σε ένα αντίγραφο ενός απλοειδούς γονιδιώματος. Το μέγεθος του γονιδιώματος ποικίλλει ευρέως μεταξύ των ειδών. Τα ασπόνδυλα έχουν μικρά γονιδιώματα, αυτό συσχετίζεται επίσης με μικρό αριθμό μεταθετών στοιχείων. Τα ψάρια και τα αμφίβια έχουν γονιδιώματα μεσαίου μεγέθους και τα πουλιά έχουν σχετικά μικρά γονιδιώματα, αλλά έχει προταθεί ότι τα πουλιά έχασαν σημαντικό μέρος των γονιδιωμάτων τους κατά τη φάση της μετάβασης στην πτήση. Πριν από αυτήν την απώλεια, η μεθυλίωση του DNA επέτρεπε την επαρκή επέκταση του γονιδιώματος.[28]
Στον άνθρωπο, το πυρηνικό γονιδίωμα περιλαμβάνει περίπου 3,2 δισεκατομμύρια νουκλεοτίδια DNA, χωρισμένα σε 24 γραμμικά μόρια, τα βραχύτερα 50.000.000 νουκλεοτίδια σε μήκος και τα μακρύτερα 260.000.000 νουκλεοτίδια, που το καθένα περιέχεται σε ένα διαφορετικό χρωμόσωμα.[44] Δεν υπάρχει σαφής και συνεπής συσχέτιση μεταξύ μορφολογικής πολυπλοκότητας και μεγέθους γονιδιώματος είτε σε προκαρυωτικά είτε σε ευκαρυωτικά κύτταρα.[29][45] Το μέγεθος του γονιδιώματος είναι σε μεγάλο βαθμό συνάρτηση της επέκτασης και της συστολής των επαναλαμβανόμενων στοιχείων DNA. Δεδομένου ότι τα γονιδιώματα είναι πολύ περίπλοκα, μια ερευνητική στρατηγική είναι να μειωθεί ο αριθμός των γονιδίων σε ένα γονιδίωμα στο απολύτως ελάχιστο και να διατηρηθεί ο εν λόγω οργανισμός. Γίνεται πειραματική εργασία σε ελάχιστα γονιδιώματα για μονοκύτταρους οργανισμούς, καθώς και ελάχιστα γονιδιώματα για πολυκυτταρικούς οργανισμούς. Το έργο είναι τόσο ζωντανά όσο και υπολογιστικά .[46][47]
Μέγεθος γονιδιώματος λόγω μεταθετών
Υπάρχουν πολλές τεράστιες διαφορές στο μέγεθος στα γονιδιώματα, που αναφέρθηκαν προηγουμένως στα πολυκύτταρα ευκαρυωτικά γονιδιώματα. Ο κύριος λόγος για τον οποίο υπάρχει τόσο μεγάλη ποικιλία μεγεθών οφείλεται στην παρουσία μεταθετών στοιχείων. Τα TE (μεταθετά στοιχεία "transposable elements") είναι γνωστό ότι συμβάλλουν σε μια σημαντική αλλαγή σε μάζα κυττάρου του DNA.[28] Αυτή η διαδικασία σχετίζεται με τη μακροπρόθεσμη προσαρμογή τους στο γονιδίωμα του ξενιστή, και συνεπώς, με την επέκταση του μεγέθους του γονιδιώματος.
Εδώ είναι ένας πίνακας ορισμένων σημαντικών ή αντιπροσωπευτικών γονιδιωμάτων.
| Τύπος οργανισμού | Οργανισμός | Μέγεθος γονιδιώματος (Ζεύγη βάσεων) |
Προσεγγιστικός αριθμός γονιδίων | Σημείωση | |
|---|---|---|---|---|---|
| Ιός | Κυκλοϊός χοίρων τύπος 1 | 1.759 | 1,8 kB | Οι μικρότεροι ιοί που αντιγράφονται αυτόνομα σε ευκαρυωτικά κύτταρα.[48] | |
| Ιός | Βακτηριοφάγος MS2 | 3.569 | 3,5 kB | Πρώτο αλληλουχημένο γονιδίωμα RNA[49] | |
| Ιός | SV40 | 5.224 | 5,2 kB | [50] | |
| Ιός | Φάγος Φ-X174 | 5.386 | 5,4 kB | Πρώτο αλληλουχημένο γονιδίωμα DNA [51] | |
| Ιός | HIV | 9.749 | 9,7 kB | [52] | |
| Ιός | Φάγος λ | 48.502 | 48,5 kB | Συχνά χρησιμοποιείται ως φορέας για την κλωνοποίηση ανασυνδυασμένου DNA. | |
| Ιός | Μεγαϊός Megavirus | 1.259.197 | 1,3 MB | Μέχρι το 2013 το μεγαλύτερο γνωστό ιικό γονιδίωμα.[56] | |
| Ιός | Pandoravirus salinus | 2.470.000 | 2,47 MB | Το μεγαλύτερο γνωστό ιικό γονιδίωμα.[57] | |
| Ευκαρυωτικό οργανίδιο | Ανθρώπινο μιτοχόνδριο | 16.569 | 16,6 kB | [58] | |
| Βακτήριο | Nasuia deltocephalinicola (strain NAS-ALF) | 112.091 | 112 kB | 137 | Το μικρότερο γνωστό μη-ιικό γονιδίωμα. Συμβιώτης των τζιτζικιών[59] |
| Βακτήριο | Carsonella ruddii | 159.662 | 160 kB | Ενδοσυμβιώτης των εντόμων ψυλίδες | |
| Βακτήριο | Buchnera aphidicola | 600.000 | 600 kB | Ενδοσυμβιώτης των αφιδών[60] | |
| Βακτήριο | Wigglesworthia glossinidia | 700.000 | 700Kb | Συμβιώτης στο έντερο των μυγών τσε-τσε | |
| Βακτήριο – κυανοβακτήριο | Prochlorococcus spp. (1,7 Mb) | 1.700.000 | 1,7 MB | 1.884 | Το μικρότερο γνωστό γονιδίωμα κυανοβακτηρίου. Ένας από τους κύριους φωτοσυνθέτες στη Γη. [61][62] |
| Βακτήριο | Haemophilus influenzae | 1.830.000 | 1,8 MB | Πρώτο γονιδίωμα ενός ζωντανού οργανισμού με αλληλουχία, Ιούλιος 1995[63] | |
| Βακτήριο | Εσερίχια κόλι | 4.600.000 | 4,6 MB | 4,288 | [64] |
| Βακτήριο – κυανοβακτήριο | Nostoc punctiforme | 9.000.000 | 9 MB | 7.432 | 7432 ανοικτά πλαίσια ανάγνωσης[65] |
| Βακτήριο | Solibacter usitatus (στέλεχος Ellin 6076) | 9.970.000 | 10 MB | [66] | |
| Αμοιβαδοειδής | Polychaos dubium (moeba" dubia) | 670.000.000.000 | 670 GB | Μεγαλύτερο γνωστό γονιδίωμα.[67] (Αμφισβητούμενο)[68] | |
| Φυτό | Genlisea tuberosa | 61.000.000 | 61 MB | Το μικρότερο κατεγραμμένο γονιδίωμα ανθόφυτου, 2014.[69] | |
| Φυτό | Arabidopsis thaliana | 135.000.000[70] | 135 MB | 27.655[71] | First plant genome sequenced, December 2000.[72] |
| Φυτό | Populus trichocarpa | 480.000.000 | 480 MB | 73.013 | Τα πρώτα τρία γονιδιώματα που αλληλουχήθηκαν, Σεπτέμβριος 2006[73] |
| Φυτό | Fritillaria assyriaca | 130.000.000.000 | 130 GB | ||
| Φυτό | Paris japonica (ιθαγενές της Ιαπωνίας, ωχρό πέταλο) | 150.000.000.000 | 150 GB | Το μεγαλύτερο γνωστό γονιδίωμα φυτού [74] | |
| Φυτό | Physcomitrella patens | 480.000.000 | 480 MB | Πρώτο γονιδίωμα βρυοφύτου που αλληλουχήθηκε, Ιανουάριος 2008.[75] | |
| Μύκητας – ζυμομύκητες | Saccharomyces cerevisiae | 12.100.000 | 12,1 MB | 6.294 | Το πρώτο ευκαρυωτικό γονιδίωμα που αλληλουχήθηκε, 1996[76] |
| Μύκητας | Aspergillus nidulans | 30.000.000 | 30 MB | 9.541 | [77] |
| Νηματώδη | Pratylenchus coffeae | 20.000.000 | 20 MB | [78] Ελάχιστο γνωστό γονιδίωμα ζώου[79] | |
| Νηματώδες | Caenorhabditis elegans | 100.300.000 | 100 MB | 19.000 | Πρώτο πολυκύτταρο γονιδίωμα ζώου που αλληλουχήθηκε, Δεκέμβριος 1998[80] |
| Έντομο | Drosophila melanogaster (δροσόφιλα) | 175.000.000 | 175 MB | 13.600 | Διακύμανση μεγέθους με βάση το στέλεχος (175-180Mb; το τυπικό y w στέλεχος είναι 175Mb)[81] |
| Έντομο | Apis mellifera (μέλισσα) | 236.000.000 | 236 MB | 10.157 | [82] |
| Έντομο | Bombyx mori (μεταξοσκώληκας) | 432.000.000 | 432 MB | 14.623 | 14.623 προβλεπόμενα γονίδια[83] |
| Έντομο | Solenopsis invicta (μηρμύγκι της φωτιάς) | 480.000.000 | 480 MB | 16.569 | [84] |
| Θηλαστικό | Mus musculus | 2.700.000.000 | 2,7 GB | 20.210 | [85] |
| Θηλαστικό | Pan paniscus | 3.286.640.000 | 3.3 GB | 20.000 | Μπονόμπο – εκτιμώμενο μέγεθος γονιδιώματος 3,29 δισεκατομμύρια ζεύγη βάσεων[86] |
| Θηλαστικό | Homo sapiens | 3.000.000.000 | 3 GB | 20.000 | Εκτιμώμενο μέγεθος γονιδιώματος του Homo sapiens στα 3,2 τρισεκατομμύρια ζεύγη βάσεων το 2001[87][88]
Αρχική αλληλούχιση και ανάλυση του ανθρώπινου γονιδιώματος[89] |
| Πτηνό | Gallus gallus | 1.043.000.000 | 1,0 GB | 20.000 | [90] |
| Ψάρι | Tetraodon nigroviridis (τύπος τετραοδοντιδών) | 385.000.000 | 390 MB | Το μικρότερο γνωστό γονιδίωμα σπονδυλωτού εκτιμώμενο σε 340 Mb[91][92] – 385 Mb.[93] | |
| Ψάρι | Protopterus aethiopicus (πνευμονόψαρο) | 130.000.000.000 | 130 GB | Το μεγαλύτερο γνωστό γονιδίωμα σπονδυλωτού | |
Γονιδιωματικές αλλοιώσεις
Όλα τα κύτταρα ενός οργανισμού προκύπτουν από ένα μοναδικό κύτταρο, έτσι αναμένεται να έχουν ταυτόσημα γονιδιώματα. Ωστόσο, σε κάποιες περιπτώσεις, προκύπτουν διαφορές. Τόσο η διαδικασία αντιγραφής του DNA κατά τη διαίρεση του κυττάρου όσο και η έκθεση σε περιβαλλοντικά μεταλλαξιογόνα μπορούν να έχουν ως αποτέλεσμα μεταλλάξεις σε σωματικά κύτταρα. Σε κάποιες περιπτώσεις, τέτοιες μεταλλάξεις οδηγούν σε καρκίνο, επειδή προκαλούν διαίρεση των κυττάρων και εισβάλλουν στους περιβάλλοντες ιστούς.[94] Σε συγκεκριμένα λεμφοκύτταρα στο ανθρώπινο ανοσοποιητικό σύστημα, ο ανασυνδυασμός V(D)J δημιουργεί διαφορετικές γονιδιωματικές αλληλουχίες τέτοιες που κάθε κύτταρο παράγει ένα μοναδικό αντίσωμα ή υποδοχείς κυττάρων Τ. Κατά τη μείωση, τα διπλοειδή κύτταρα διαιρούνται δύο φορές για να παράξουν απλοειδή γαμετικά κύτταρα. Κατά τη διάρκεια αυτής της διαδικασίας, ο ανασυνδυασμός καταλήγει σε αναδιάταξη του γενετικού υλικού από τα ομόλογα χρωμοσώματα, έτσι ώστε κάθε γαμέτης να έχει μοναδικό γονιδίωμα.
Επαναπρογραμματισμός ολόκληρου γονιδιώματος
Ο επαναπρογραμματισμός ολόκληρου του γονιδιώματος σε γεννητικά κύτταρα ποντικιών περιλαμβάνει σβήσιμο επιγενετικού αποτυπώματος που οδηγεί σε παντοδυναμία. Ο επαναπρογραμματισμός διευκολύνεται από ενεργή απομεθυλίωση του DNA, μια διαδικασίας που απαιτεί την επιδιόρθωση με εκτομή βάσης της οδού του DNA.[95] Αυτή η οδός χρησιμοποιείται στο σβήσιμο της μεθυλίωσης CpG (5-μεθυλοκυτοσίνη 5mC) σε αρχέγονα γεννητικά κύτταρα. Το σβήσιμο της 5mC συμβαίνει μέσω της μετατροπής σε 5-υδροξυλμεθυλοκυτοσίνης (5hmC) που καθοδηγείται από υψηλά επίπεδα ενζύμων της δέκα-ένδεκα διοξυγονάσης TET1 και TET2.[96]
Εξέλιξη γονιδιώματος
Τα γονιδιώματα είναι περισσότερα από το άθροισμα γονιδίων ενός οργανισμού και έχουν χαρακτηριστικά που μπορούν να μετρηθούν και να μελετηθούν χωρίς παραπομπή σε λεπτομέρειες οποιονδήποτε συγκεκριμένων γονιδίων και των προϊόντων τους. Οι ερευνητές συγκρίνουν χαρακτηριστικά όπως καρυότυπος (αριθμός χρωμοσωμάτων)), μέγεθος γονιδίου, σειρά γονιδίου, προτίμηση χρήσης κωδικονίων (codon usage bias), και περιεχόμενο γουανίνης-κυτοσίνης (GC-content) για να προσδιορίσουν ποιοι μηχανισμοί θα μπορούσαν να έχουν παράξει τη μεγάλη ποικιλία των γονιδιωμάτων που υπάρχουν σήμερα. Ο διπλασιασμός γονιδίου παίζουν σημαντικό ρόλο στη διαμόρφωση του γονιδιώματος. Ο διπλασιασμός μπορεί να εκτείνεται από την επέκταση των βραχέων διαδοχοκών επαναλήψεων (short tandem repeats), τον διπλασιασμό συστοιχίας γονιδίων και μέχρι τον διπλασιασμό ολόκληρων χρωμοσωμάτων ή ακόμα ολόκληρων γονιδιωμάτων. Τέτοιοι διπλασιασμοί είναι προφανώς βασικοί στη δημιουργία καινοτομιών. H Οριζόντια μεταφορά γονιδίου επικαλείται για να εξηγήσει πώς υπάρχει συχνά μια ακραία ομοιότητα μεταξύ μικρών τμημάτων των γονιδιωμάτων δύο οργανισμών που κατά τα άλλα έχουν πολύ μακρινή συγγένεια. Η οριζόντια μεταφορά γονιδίων φαίνεται να είναι κοινή μεταξύ πολλών μικροοργανισμών. Επίσης, τα ευκαρυωτικά κύτταρα φαίνεται να έχουν βιώσει μεταφορά κάποιου γενετικού υλικού από τα χλωροπλαστικά και τα μιτοχονδριακά γονιδιώματά τους στα πυρηνικά χρωμοσώματά τους. Τα πρόσφατα εμπειρικά δεδομένα προτείνουν ότι ένας σημαντικός ρόλος ιών και υποιικών δικτύων RNA αντιπροσωπεύει έναν κύριο οδηγό ρόλο στη δημιουργία γενετικής καινοτομίας και φυσικής επεξεργασίας γονιδιώματος.
Παραπομπές
- ↑ Roth, Stephanie Clare (2019-07-01). «What is genomic medicine?». Journal of the Medical Library Association (University Library System, University of Pittsburgh) 107 (3). doi:. ISSN 1558-9439. PMID 31258451.
- ↑ Brosius, J (2009), «The Fragmented Gene», Annals of the New York Academy of Sciences 1178 (1): 186–93, doi:, PMID 19845638
- ↑ Ridley M (2006). Genome: the autobiography of a species in 23 chapters (PDF). New York: Harper Perennial. ISBN 978-0-06-019497-0. Αρχειοθετήθηκε από το πρωτότυπο (PDF) στις 24 Οκτωβρίου 2018. Ανακτήθηκε στις 11 Μαΐου 2016.
- ↑ Winkler HL (1920). Verbreitung und Ursache der Parthenogenesis im Pflanzen- und Tierreiche. Jena: Verlag Fischer.
- ↑ «definition of Genome in Oxford dictionary». Ανακτήθηκε στις 25 Μαρτίου 2014.
- ↑ «All about genes». www.beowulf.org.uk.
- ↑ «Genome Home». 8 Δεκεμβρίου 2010. Ανακτήθηκε στις 27 Ιανουαρίου 2011.
- ↑ Zimmer, Carl (December 18, 2013). «Toe Fossil Provides Complete Neanderthal Genome». The New York Times. https://www.nytimes.com/2013/12/19/science/toe-fossil-provides-complete-neanderthal-genome.html. Ανακτήθηκε στις 18 December 2013.
- ↑ «The complete genome sequence of a Neanderthal from the Altai Mountains». Nature 505 (7481): 43–49. January 2014. doi:. PMID 24352235. Bibcode: 2014Natur.505...43P.
- ↑ Wade, Nicholas (2007-05-31). «Genome of DNA Pioneer Is Deciphered». The New York Times. https://www.nytimes.com/2007/05/31/science/31cnd-gene.html. Ανακτήθηκε στις 2 April 2010.
- ↑ «What's a Genome?». Genomenewsnetwork.org. 15 Ιανουαρίου 2003. Ανακτήθηκε στις 27 Ιανουαρίου 2011.
- ↑ NCBI_user_services (29 Μαρτίου 2004). «Mapping Factsheet». Αρχειοθετήθηκε από το πρωτότυπο στις 19 Ιουλίου 2010. Ανακτήθηκε στις 27 Ιανουαρίου 2011.
- ↑ Genome Reference Consortium. «Assembling the Genome». Ανακτήθηκε στις 23 Αυγούστου 2016.
- ↑ Kaplan, Sarah (2016-04-17). «How do your 20,000 genes determine so many wildly different traits? They multitask.». The Washington Post. https://www.washingtonpost.com/news/speaking-of-science/wp/2016/05/17/how-do-your-20000-genes-determine-so-many-wildly-different-traits-they-multitask/. Ανακτήθηκε στις 2016-08-27.
- ↑ Check Hayden, Erika (2015). «Scientists hope to attract millions to 'DNA.LAND'» (στα αγγλικά). Nature. doi:. https://www.nature.com/news/scientists-hope-to-attract-millions-to-dna-land-1.18514.
- ↑ Zimmer, Carl. «Game of Genomes, Episode 13: Answers and Questions». STAT. Ανακτήθηκε στις 27 Αυγούστου 2016.
- ↑ Gelderblom, Hans R. (1996). Medical Microbiology (4th έκδοση). Galveston, TX: The University of Texas Medical Branch at Galveston.
- ↑ Urry, Lisa A. (2016). Campbell Biology: seventh edition. New York: Hoboken: Pearson Higher Education. σελίδες 403–404. ISBN 0134093410.
- ↑ Urry, Lisa A (2016). Campbell Biology: seventh edition. New York: Hoboken: Pearson Higher Education. σελίδες 403–404. ISBN 0134093410.
- ↑ «Archaeal chromosome biology». Journal of Molecular Microbiology and Biotechnology 24 (5–6): 420–27. 2014. doi:. PMID 25732343.
- ↑ Chaconas, George; Chen, Carton W. (2005). «Replication of Linear Bacterial Chromosomes: No Longer Going Around in Circles». The Bacterial Chromosome: 525–540. doi:. ISBN 9781555812324. http://www.asmscience.org/content/book/10.1128/9781555817640.chap29.
- ↑ «Bacterial Chromosomes». Microbial Genetics. 2002.
- ↑ 23,0 23,1 «Constraints and plasticity in genome and molecular-phenome evolution». Nature Reviews. Genetics 11 (7): 487–98. July 2010. doi:. PMID 20548290.
- ↑ «Extreme genome reduction in symbiotic bacteria». Nature Reviews. Microbiology 10 (1): 13–26. November 2011. doi:. PMID 22064560.
- ↑ «Insights from 20 years of bacterial genome sequencing». Functional & Integrative Genomics 15 (2): 141–61. March 2015. doi:. PMID 25722247.
- ↑ «Scientists sequence asexual tiny worm whose lineage stretches back 18 million years». ScienceDaily. Ανακτήθηκε στις 7 Νοεμβρίου 2017.
- ↑ Khandelwal, Sharda (March 1990). «Chromosome evolution in the genus Ophioglossum L.». Botanical Journal of the Linnean Society 102 (3): 205–17. doi:.
- ↑ 28,0 28,1 28,2 28,3 Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 August 2020). «DNA methylation enables transposable element-driven genome expansion». Proceedings of the National Academy of Sciences of the United States of America 117 (32): 19359–19366. doi:. ISSN 1091-6490. PMID 32719115. PMC 7431005. https://pubmed.ncbi.nlm.nih.gov/32719115/.
- ↑ 29,0 29,1 29,2 Lewin, Benjamin (2004). Genes VIII (8th έκδοση). Upper Saddle River, NJ: Pearson/Prentice Hall. ISBN 978-0-13-143981-8.
- ↑ «FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences». BMC Bioinformatics 44 (9): e89. Apr 2021. doi:. PMID 33879057.
- ↑ Stojanovic, Nikola, επιμ. (2007). Computational genomics : current methods. Wymondham: Horizon Bioscience. ISBN 978-1-904933-30-4.
- ↑ 32,0 32,1 32,2 «Repeat DNA in genome organization and stability». Current Opinion in Genetics & Development 31: 12–19. April 2015. doi:. PMID 25917896.
- ↑ 33,0 33,1 «The biological effects of simple tandem repeats: lessons from the repeat expansion diseases». Genome Research 18 (7): 1011–19. July 2008. doi:. PMID 18593815.
- ↑ «Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review». Molecular Ecology 11 (12): 2453–65. December 2002. doi:. PMID 12453231.
- ↑ 35,0 35,1 «Transposable elements and the evolution of eukaryotic genomes». Proceedings of the National Academy of Sciences of the United States of America 103 (47): 17600–01. November 2006. doi:. PMID 17101965. Bibcode: 2006PNAS..10317600W.
- ↑ 36,0 36,1 36,2 36,3 36,4 «Mobile elements: drivers of genome evolution». Science 303 (5664): 1626–32. March 2004. doi:. PMID 15016989. Bibcode: 2004Sci...303.1626K.
- ↑ «Transposon | genetics». Encyclopedia Britannica (στα Αγγλικά). Ανακτήθηκε στις 5 Δεκεμβρίου 2020.
- ↑ Sanders, Mark Frederick (2019). Genetic Analysis: an integrated approach third edition. New York: Pearson, always learning, and mastering. σελ. 425. ISBN 9780134605173.
- ↑ «Mobile elements and mammalian genome evolution». Current Opinion in Genetics & Development 13 (6): 651–58. December 2003. doi:. PMID 14638329.
- ↑ «Transposable elements and host genome evolution». Trends in Ecology & Evolution 15 (3): 95–99. March 2000. doi:. PMID 10675923.
- ↑ «Comparative genomics and molecular dynamics of DNA repeats in eukaryotes». Microbiology and Molecular Biology Reviews 72 (4): 686–727. December 2008. doi:. PMID 19052325.
- ↑ «The impact of retrotransposons on human genome evolution». Nature Reviews. Genetics 10 (10): 691–703. October 2009. doi:. PMID 19763152.
- ↑ «LINE-1 retrotransposons: modulators of quantity and quality of mammalian gene expression?». BioEssays 27 (8): 775–84. August 2005. doi:. PMID 16015595.
- ↑ «Human genome». Ανακτήθηκε στις 19 Αυγούστου 2016.
- ↑ «Eukaryotic genome size databases». Nucleic Acids Research 35 (Database issue): D332–38. January 2007. doi:. PMID 17090588.
- ↑ «Essential genes of a minimal bacterium». Proceedings of the National Academy of Sciences of the United States of America 103 (2): 425–30. January 2006. doi:. PMID 16407165. Bibcode: 2006PNAS..103..425G.
- ↑ «Towards synthesis of a minimal cell». Molecular Systems Biology 2 (1): 45. 2006. doi:. PMID 16924266.
- ↑ Mankertz P (2008). «Molecular Biology of Porcine Circoviruses». Animal Viruses: Molecular Biology. Caister Academic Press. ISBN 978-1-904455-22-6.
- ↑ «Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene». Nature 260 (5551): 500–07. April 1976. doi:. PMID 1264203. Bibcode: 1976Natur.260..500F.
- ↑ «Complete nucleotide sequence of SV40 DNA». Nature 273 (5658): 113–20. May 1978. doi:. PMID 205802. Bibcode: 1978Natur.273..113F.
- ↑ «Nucleotide sequence of bacteriophage phi X174 DNA». Nature 265 (5596): 687–95. February 1977. doi:. PMID 870828. Bibcode: 1977Natur.265..687S.
- ↑ «Virology – Human Immunodeficiency Virus And Aids, Structure: The Genome And Proteins Of HIV». Pathmicro.med.sc.edu. 1 Ιουλίου 2010. Ανακτήθηκε στις 27 Ιανουαρίου 2011.
- ↑ «Recombineering: genetic engineering in bacteria using homologous recombination». Current Protocols in Molecular Biology Chapter 1: Unit 1.16. April 2007. doi:. ISBN 978-0-471-14272-0. PMID 18265390.
- ↑ «A new look at bacteriophage lambda genetic networks». Journal of Bacteriology 189 (2): 298–304. January 2007. doi:. PMID 17085553.
- ↑ «Nucleotide sequence of bacteriophage lambda DNA». Journal of Molecular Biology 162 (4): 729–73. December 1982. doi:. PMID 6221115.
- ↑ «Genomics of Megavirus and the elusive fourth domain of Life». Communicative & Integrative Biology 5 (1): 102–06. January 2012. doi:. PMID 22482024.
- ↑ «Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes». Science 341 (6143): 281–86. July 2013. doi:. PMID 23869018. Bibcode: 2013Sci...341..281P. https://hal-cea.archives-ouvertes.fr/cea-00862677/file/phi.pdf.
- ↑ «Sequence and organization of the human mitochondrial genome». Nature 290 (5806): 457–65. April 1981. doi:. PMID 7219534. Bibcode: 1981Natur.290..457A.
- ↑ «Small, smaller, smallest: the origins and evolution of ancient dual symbioses in a Phloem-feeding insect». Genome Biology and Evolution 5 (9): 1675–88. 5 August 2013. doi:. PMID 23918810.
- ↑ «Genome sequence of the endocellular bacterial symbiont of aphids Buchnera sp. APS». Nature 407 (6800): 81–86. September 2000. doi:. PMID 10993077. Bibcode: 2000Natur.407...81S.
- ↑ «Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation». Nature 424 (6952): 1042–47. August 2003. doi:. PMID 12917642. Bibcode: 2003Natur.424.1042R.
- ↑ «Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome». Proceedings of the National Academy of Sciences of the United States of America 100 (17): 10020–25. August 2003. doi:. PMID 12917486. Bibcode: 2003PNAS..10010020D.
- ↑ «Whole-genome random sequencing and assembly of Haemophilus influenzae Rd». Science 269 (5223): 496–512. July 1995. doi:. PMID 7542800. Bibcode: 1995Sci...269..496F.
- ↑ «The complete genome sequence of Escherichia coli K-12». Science 277 (5331): 1453–62. September 1997. doi:. PMID 9278503.
- ↑ «An overview of the genome of Nostoc punctiforme, a multicellular, symbiotic cyanobacterium». Photosynthesis Research 70 (1): 85–106. 2001. doi:. PMID 16228364.
- ↑ Steinke, Dirk, επιμ. (15 September 2011). «Biological consequences of ancient gene acquisition and duplication in the large genome of Candidatus Solibacter usitatus Ellin6076». PLOS ONE 6 (9): e24882. doi:. PMID 21949776. Bibcode: 2011PLoSO...624882C.
- ↑ «The dynamic nature of eukaryotic genomes». Molecular Biology and Evolution 25 (4): 787–94. April 2008. doi:. PMID 18258610.
- ↑ ScienceShot: Biggest Genome Ever Αρχειοθετήθηκε 11 October 2010 στο Wayback Machine., comments: "The measurement for Amoeba dubia and other protozoa which have been reported to have very large genomes were made in the 1960s using a rough biochemical approach which is now considered to be an unreliable method for accurate genome size determinations."
- ↑ «Evolution of genome size and chromosome number in the carnivorous plant genus Genlisea (Lentibulariaceae), with a new estimate of the minimum genome size in angiosperms». Annals of Botany 114 (8): 1651–63. December 2014. doi:. PMID 25274549.
- ↑ «Genome Assembly». The Arabidopsis Information Resource (TAIR).
- ↑ «Details - Arabidopsis thaliana - Ensembl Genomes 40». plants.ensembl.org.
- ↑ «Smallest angiosperm genomes found in lentibulariaceae, with chromosomes of bacterial size». Plant Biology 8 (6): 770–77. November 2006. doi:. PMID 17203433.
- ↑ «The genome of black cottonwood, Populus trichocarpa (Torr. & Gray)». Science 313 (5793): 1596–604. September 2006. doi:. PMID 16973872. Bibcode: 2006Sci...313.1596T. https://digital.library.unt.edu/ark:/67531/metadc883930/m2/1/high_res_d/901819.pdf.
- ↑ Pellicer, Jaume; Fay, Michael F.; Leitch, Ilia J. (15 September 2010). «The largest eukaryotic genome of them all?». Botanical Journal of the Linnean Society 164 (1): 10–15. doi:.
- ↑ «Exploring plant biodiversity: the Physcomitrella genome and beyond». Trends in Plant Science 13 (10): 542–49. October 2008. doi:. PMID 18762443.
- ↑ «Saccharomyces Genome Database». Yeastgenome.org. Ανακτήθηκε στις 27 Ιανουαρίου 2011.
- ↑ «Sequencing of Aspergillus nidulans and comparative analysis with A. fumigatus and A. oryzae». Nature 438 (7071): 1105–15. December 2005. doi:. PMID 16372000. Bibcode: 2005Natur.438.1105G.
- ↑ «Genome size of plant-parasitic nematodes». Nematology 9 (3): 449–50. 2007. doi:.
- ↑ Gregory TR (2005). «Animal Genome Size Database». Gregory, T.R. (2016). Animal Genome Size Database.
- ↑ The C. elegans Sequencing Consortium (December 1998). «Genome sequence of the nematode C. elegans: a platform for investigating biology». Science 282 (5396): 2012–18. doi:. PMID 9851916. Bibcode: 1998Sci...282.2012..
- ↑ «Intrapopulation genome size variation in D. melanogaster reflects life history variation and plasticity». PLOS Genetics 10 (7): e1004522. July 2014. doi:. PMID 25057905.
- ↑ Honeybee Genome Sequencing Consortium (October 2006). «Insights into social insects from the genome of the honeybee Apis mellifera». Nature 443 (7114): 931–49. doi:. PMID 17073008. Bibcode: 2006Natur.443..931T.
- ↑ The International Silkworm Genome (December 2008). «The genome of a lepidopteran model insect, the silkworm Bombyx mori». Insect Biochemistry and Molecular Biology 38 (12): 1036–45. doi:. PMID 19121390.
- ↑ «The genome of the fire ant Solenopsis invicta». Proceedings of the National Academy of Sciences of the United States of America 108 (14): 5679–84. April 2011. doi:. PMID 21282665. Bibcode: 2011PNAS..108.5679W.
- ↑ Roberts, Richard J, επιμ. (May 2009). «Lineage-specific biology revealed by a finished genome assembly of the mouse». PLOS Biology 7 (5): e1000112. doi:. PMID 19468303.
- ↑ «Pan paniscus (pygmy chimpanzee)». nih.gov. Ανακτήθηκε στις 30 Ιουνίου 2016.
- ↑ Eric Lander (Feb 15, 2001). «Initial sequencing and analysis of the human genome». Nature 409 (6822): 860–921. doi:. PMID 11237011. Table 8.
- ↑ «Functional and Comparative Genomics Fact Sheet». Ornl.gov. Αρχειοθετήθηκε από το πρωτότυπο στις 20 Σεπτεμβρίου 2008.
- ↑ «The sequence of the human genome». Science 291 (5507): 1304–51. February 2001. doi:. PMID 11181995. Bibcode: 2001Sci...291.1304V.
- ↑ International Chicken Genome Sequencing Consortium (December 2004). «Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution» (στα αγγλικά). Nature 432 (7018): 695–716. doi:. ISSN 0028-0836. PMID 15592404. Bibcode: 2004Natur.432..695C.
- ↑ «Characterization and repeat analysis of the compact genome of the freshwater pufferfish Tetraodon nigroviridis». Genome Research 10 (7): 939–49. July 2000. doi:. PMID 10899143.
- ↑ «Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype». Nature 431 (7011): 946–57. October 2004. doi:. PMID 15496914. Bibcode: 2004Natur.431..946J.
- ↑ «Tetraodon Project Information». Αρχειοθετήθηκε από το πρωτότυπο στις 26 Σεπτεμβρίου 2012. Ανακτήθηκε στις 17 Οκτωβρίου 2012.
- ↑ «Somatic mutation in cancer and normal cells». Science 349 (6255): 1483–89. September 2015. doi:. PMID 26404825. Bibcode: 2015Sci...349.1483M.
- ↑ «Genome-wide reprogramming in the mouse germ line entails the base excision repair pathway». Science 329 (5987): 78–82. July 2010. doi:. PMID 20595612. Bibcode: 2010Sci...329...78H.
- ↑ «Germline DNA demethylation dynamics and imprint erasure through 5-hydroxymethylcytosine». Science 339 (6118): 448–52. January 2013. doi:. PMID 23223451. Bibcode: 2013Sci...339..448H.
Παραπέρα μελέτη
- Benfey P, Protopapas AD (2004). Essentials of Genomics. Prentice Hall.
- Brown, Terence A. (2002). Genomes 2. Oxford: Bios Scientific Publishers. ISBN 978-1-85996-029-5.
- Gibson, Greg· Muse, Spencer V. (2004). A Primer of Genome Science
 (Second έκδοση). Sunderland, Mass: Sinauer Assoc. ISBN 978-0-87893-234-4.
(Second έκδοση). Sunderland, Mass: Sinauer Assoc. ISBN 978-0-87893-234-4. - Gregory, T. Ryan (2005). The Evolution of the Genome. Elsevier. ISBN 978-0-12-301463-4.
- Reece, Richard J. (2004). Analysis of Genes and Genomes. Chichester: John Wiley & Sons. ISBN 978-0-470-84379-6.
- Saccone, Cecilia· Pesole, Graziano (2003). Handbook of Comparative Genomics. Chichester: John Wiley & Sons. ISBN 978-0-471-39128-9.
- «In silico multicellular systems biology and minimal genomes». Drug Discovery Today 8 (24): 1121–27. December 2003. doi:. PMID 14678738.
Εξωτερικοί σύνδεσμοι
- UCSC Genome Browser – view the genome and annotations for more than 80 organisms.
- genomecenter.howard.edu
- Build a DNA Molecule
- Some comparative genome sizes
- DNA Interactive: The History of DNA Science
- DNA From The Beginning
- All About The Human Genome Project—from Genome.gov
- Animal genome size database
- Plant genome size database
- GOLD:Genomes OnLine Database
- The Genome News Network
- NCBI Entrez Genome Project database
- NCBI Genome Primer
- GeneCards—an integrated database of human genes
- BBC News – Final genome 'chapter' published
- IMG (The Integrated Microbial Genomes system)—for genome analysis by the DOE-JGI
- GeKnome Technologies Next-Gen Sequencing Data Analysis—next-generation sequencing data analysis for Illumina and 454 Service from GeKnome Technologies.
|

