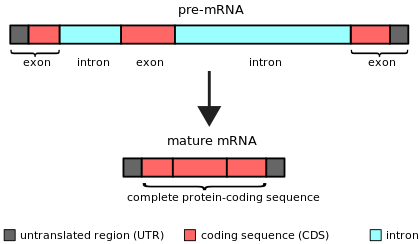
Ιντρόνιο
Ιντρόνιο (ή εσώνιο) ονομάζεται οποιαδήποτε νουκλεοτιδική αλληλουχία εντός ενός γονιδίου η οποία απομακρύνεται με το συναρμογή του RNA κατά την ωρίμανση του τελικού προϊόντος RNA.[1] [2] Με άλλα λόγια, τα ιντρόνια είναι μη κωδικοποιητικές περιοχές ενός μεταγράφου RNA, ή του DNA που το κωδικοποιεί, τα οποία απομακρύνονται με το μάτισμα πριν από τη μετάφραση.[3] [4] Η λέξη ιντρόνιο (intron) προέρχεται από τον όρο intragenic region, δηλαδή "περιοχή εντός ενός γονιδίου".[5] Η λέξη ιντρόνιο αναφέρεται τόσο στην αλληλουχία DNA εντός ενός γονιδίου όσο και στην αντίστοιχη αλληλουχία στα RNA μετάγραφα.[6] Οι αλληλουχίες που ενώνονται μαζί στο τελικό ώριμο RNA μετά το μάτισμα ονομάζονται εξώνια.
Τα ιντρόνια βρίσκονται στα γονίδια των περισσότερων οργανισμών και πολλών ιών και μπορούν να εντοπιστούν σε ένα ευρύ φάσμα γονιδίων, συμπεριλαμβανομένων εκείνων που παράγουν πρωτεΐνες, ριβοσωμικό RNA (rRNA) και μεταφορικό RNA (tRNA). Όταν οι πρωτεΐνες δημιουργούνται από γονίδια που περιέχουν ιντρόνια, το μάτισμα του RNA λαμβάνει χώρα ως μέρος της οδού επεξεργασίας RNA που ακολουθεί τη μεταγραφή και προηγείται της μετάφρασης.
Κατανομή
[Επεξεργασία | επεξεργασία κώδικα]Η συχνότητα των ιντρονίων εντός διαφορετικών γονιδιωμάτων έχει παρατηρηθεί ότι ποικίλλει ευρέως στο φάσμα των βιολογικών οργανισμών. Για παράδειγμα, τα ιντρόνια είναι εξαιρετικά κοινά στο πυρηνικό γονιδίωμα των σπονδυλωτών με γνάθους (π.χ. ανθρώπους και ποντίκια), όπου τα γονίδια που κωδικοποιούν πρωτεΐνες περιέχουν σχεδόν πάντα πολλαπλά ιντρόνια, ενώ τα ιντρόνια είναι σπάνια εντός των πυρηνικών γονιδίων ορισμένων ευκαρυωτικών μικροοργανισμών,[7] για παράδειγμα στη μαγιά αρτοποιίας/ζυθοποιίας (Saccharomyces cerevisiae). Αντιθέτως, τα μιτοχονδριακά γονιδιώματα των σπονδυλωτών στερούνται εντελώς ιντρονίων, ενώ αυτά των ευκαρυωτικών μικροοργανισμών μπορεί να περιέχουν πολλά ιντρόνια.[8]
Μια ιδιαίτερα ακραία περίπτωση είναι το γονίδιο dhc7 της Drosophila που περιέχει ένα ιντρόνιο περίπου 3,6 μεγαβάσεων (Mb), το οποίο χρειάζεται περίπου τρεις ημέρες για να μεταγραφεί.[9] [10] Στο άλλο άκρο, μια πρόσφατη μελέτη υποδεικνύει ότι το μικρότερο γνωστό ευκαρυωτικό ιντρόνιο έχει μήκος 30 ζευγών βάσεων (bp) και βρίσκεται στο ανθρώπινο γονίδιο MST1L. [11]
Βιολογικές λειτουργίες και εξέλιξη
[Επεξεργασία | επεξεργασία κώδικα]Παρόλο που τα ιντρόνια δεν κωδικοποιούν πρωτεϊνικά προϊόντα, είναι αναπόσπαστα στη ρύθμιση της γονιδιακής έκφρασης. Ορισμένα ιντρόνια από μόνα τους κωδικοποιούν λειτουργικά RNA μέσω περαιτέρω επεξεργασίας μετά από μάτισμα για τη δημιουργία μη κωδικοποιητικών RNA μορίων.[12] Το εναλλακτικό μάτισμα χρησιμοποιείται ευρέως για τη δημιουργία πολλαπλών πρωτεϊνών από ένα μόνο γονίδιο. Επιπλέον, ορισμένα ιντρόνια διαδραματίζουν βασικούς ρόλους σε ένα ευρύ φάσμα ρυθμιστικών λειτουργιών έκφρασης γονιδίων, όπως στη μη νοηματικά διαμεσολαβούμενη αποικοδόμηση[13] και την εξαγωγή του mRNA.[14]
Οι βιολογικές ρίζες των ιντρονίων είναι ασαφείς. Μετά την αρχική ανακάλυψη τους σε γονίδια του ευκαρυωτικού πυρήνα που κωδικοποιούν πρωτεΐνες, υπήρξε έντονη συζήτηση σχετικά με το εάν τα ιντρόνια στους σύγχρονους οργανισμούς κληρονομήθηκαν από έναν κοινό αρχαίο πρόγονο ή εμφανίστηκαν σε γονίδια σχετικά πρόσφατα στην εξελικτική διαδικασία. Μια άλλη θεωρία είναι ότι το σωμάτιο συναρμογής και η δομή ιντρονίων-εξωνίων των γονιδίων αποτελεί ένα απομεινάρι του κόσμου RNA.[15] Εξακολουθεί να υπάρχει σημαντική συζήτηση σχετικά με τον βαθμό στον οποίο αυτές οι υποθέσεις είναι πιο σωστές. Η δημοφιλής συναίνεση αυτή τη στιγμή είναι ότι τα ιντρόνια προέκυψαν εντός της ευκαρυωτικής γενεαλογίας ως εγωιστικά στοιχεία.[16]
Οι πρώτες μελέτες των γονιδιωματικών DNA αλληλουχιών από ένα ευρύ φάσμα οργανισμών δείχνουν ότι η δομή ιντρονίων-εξονίων ομόλογων γονιδίων σε διαφορετικούς οργανισμούς μπορεί να ποικίλει ευρέως.[17] Πιο πρόσφατες μελέτες για ολόκληρα ευκαρυωτικά γονιδιώματα έδειξαν πλέον ότι τα μήκη και η πυκνότητα (ιντρόνια/γονίδιο) των ιντρονίων ποικίλλει σημαντικά μεταξύ συγγενικών ειδών. Για παράδειγμα, ενώ το ανθρώπινο γονιδίωμα περιέχει κατά μέσο όρο 8,4 ιντρόνια / γονίδιο (139.418 στο γονιδίωμα), ο μονοκύτταρος μύκητας Encephalitozoon cuniculi περιέχει μόνο 0,0075 ιντρόνια / γονίδιο (15 ιντρόνια στο γονιδίωμα).[18] Δεδομένου ότι οι ευκαρυώτες προήλθαν από έναν κοινό πρόγονο (κοινή καταγωγή), πρέπει να έχει υπάρξει εκτεταμένο κέρδος ή απώλεια ιντρονίων κατά τη διάρκεια της εξελικτικής περιόδου.[19] [20] Αυτή η διαδικασία θεωρείται ότι υπόκειται σε επιλογή, με τάση αύξησης ιντρονίων σε μεγαλύτερα είδη λόγω του μικρότερου μεγέθους του πληθυσμού τους, και το αντίστροφο σε μικρότερα (ιδιαίτερα σε μονοκύτταρα) είδη. [21] Βιολογικοί παράγοντες επηρεάζουν επίσης το ποια γονίδια σε ένα γονιδίωμα χάνουν ή συσσωρεύουν ιντρόνια.[22] [23] [24]
Το εναλλακτικό μάτισμα εξωνίων εντός ενός γονιδίου δρα για να εισαγάγει μεγαλύτερη ποικιλία πρωτεϊνικών αλληλουχιών που μεταφράζονται από ένα μόνο γονίδιο, επιτρέποντας τη δημιουργία πολλαπλών συγγενικών πρωτεϊνών από ένα μόνο γονίδιο και ένα πρόδρομο μετάγραφο mRNA. Ο έλεγχος του εναλλακτικού ματίσματος RNA εκτελείται από ένα πολύπλοκο δίκτυο σηματοδοτικών μορίων που αποκρίνονται σε ένα ευρύ φάσμα ενδοκυτταρικών και εξωκυτταρικών σημάτων. Τα ιντρόνια περιέχουν αρκετές μικρές ακολουθίες που είναι σημαντικές για το αποτελεσματικό μάτισμα, όπως θέσεις δέκτη και δότη σε κάθε άκρο του ιντρονίου, καθώς και μια τοποθεσία σημείου διακλάδωσης, οι οποίες απαιτούνται για το ορθό μάτισμα από το σωμάτιο συναρμογής. Μερικά ιντρόνια είναι γνωστό ότι ενισχύουν την έκφραση του γονιδίου στο οποίο περιέχονται μέσω μιας διαδικασίας γνωστής ως ενίσχυσης μεσολαβούμενης από ιντρόνια (ΙΜΕ).
Οι ενεργά μεταγραφείσες περιοχές του DNA σχηματίζουν συχνά βρόχους R που είναι ευάλωτοι σε βλάβες του DNA. Σε γονίδια ζύμης με υψηλή έκφραση, τα ιντρόνια αναστέλλουν το σχηματισμό βρόχου R και την εμφάνιση βλάβης στο DNA.[25] Ανάλυση ολόκληρου του γονιδιώματος τόσο σε ζυμομύκητες όσο και σε ανθρώπους αποκάλυψε ότι τα γονίδια που περιέχουν ιντρόνια έχουν μειωμένα επίπεδα βρόχου R και μειωμένη βλάβη στο DNA σε σύγκριση με γονίδια παρόμοιας έκφρασης χωρίς ιντρόνια. Η εισαγωγή ενός ιντρονίου εντός ενός γονιδίου επιρρεπούς σε βρόχο R μπορεί επίσης να καταστείλει τον σχηματισμό και τον ανασυνδυασμό βρόχου R. Οι Bonnet et al. (2017) εικάζουν ότι η λειτουργία των ιντρονίων στη διατήρηση της γενετικής σταθερότητας μπορεί να εξηγήσει την εξελικτική τους διατήρηση σε ορισμένες τοποθεσίες, ιδιαίτερα σε γονίδια με υψηλή έκφραση.
Παραπομπές
[Επεξεργασία | επεξεργασία κώδικα]- ↑ Alberts, Bruce (2008). Molecular biology of the cell. New York: Garland Science. ISBN 978-0-8153-4105-5.
- ↑ Stryer, Lubert· Berg, Jeremy Mark (2007). Biochemistry. San Francisco: W.H. Freeman. ISBN 978-0-7167-6766-4.
- ↑ Ghosh, Shampa; Sinha, Jitendra Kumar (2017), Vonk, Jennifer; Shackelford, Todd, επιμ., «Intron», Encyclopedia of Animal Cognition and Behavior (Springer International Publishing): 1–5, doi:, ISBN 978-3-319-47829-6
- ↑ Editors, B. D. (6 Αυγούστου 2017). «Intron». Biology Dictionary (στα Αγγλικά). Ανακτήθηκε στις 1 Δεκεμβρίου 2019.
- ↑ Gilbert, Walter (1978). «Why genes in pieces». Nature 271 (5645): 501. doi:. PMID 622185.
- ↑ Kinniburgh, Alan; mertz, j; Ross, J. (July 1978). «The precursor of mouse β-globin messenger RNA contains two intervening RNA sequences». Cell 14 (3): 681–693. doi:. PMID 688388. http://www.cell.com/abstract/0092-8674(78)90251-9#.
- ↑ «Comparative genomic analysis of fungal genomes reveals intron-rich ancestors». Genome Biol. 8 (10): R223. 2007. doi:. PMID 17949488.
- ↑ Taanman, Jan-Willem (1999). «The mitochondrial genome: structure, transcription, translation and replication». Biochimica et Biophysica Acta (BBA) - Bioenergetics 1410 (2): 103–123. doi:. PMID 10076021.
- ↑ Tollervey, David; Caceres, Javier F (November 2000). «RNA Processing Marches on». Cell 103 (5): 703–709. doi:. PMID 11114327.
- ↑ Reugels, AM; Kurek, R; Lammermann, U; Bünemann, H (February 2000). «Mega-introns in the dynein gene DhDhc7(Y) on the heterochromatic Y chromosome give rise to the giant threads loops in primary spermatocytes of Drosophila hydei.». Genetics 154 (2): 759–69. PMID 10655227. PMC 1460963. http://www.genetics.org/content/154/2/759.long. Ανακτήθηκε στις 12 December 2014.
- ↑ Piovesan, Allison; Caracausi, Maria; Ricci, Marco; Strippoli, Pierluigi; Vitale, Lorenza; Pelleri, Maria Chiara (1 December 2015). «Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank». DNA Research 22 (6): 495–503. doi:. PMID 26581719.
- ↑ «Critical association of ncRNA with introns». Nucleic Acids Res. 39 (6): 2357–66. March 2011. doi:. PMID 21071396.
- ↑ «Introns in UTRs: why we should stop ignoring them.». BioEssays 34 (12): 1025–34. December 2012. doi:. PMID 23108796.
- ↑ Cenik, Can; Chua, Hon Nian; Zhang, Hui; Tarnawsky, Stefan P.; Akef, Abdalla; Derti, Adnan; Tasan, Murat; Moore, Melissa J. και άλλοι. (2011). «Genome Analysis Reveals Interplay between 5′UTR Introns and Nuclear mRNA Export for Secretory and Mitochondrial Genes». PLoS Genetics 7 (4): e1001366. doi:. ISSN 1553-7404. PMID 21533221.
- ↑ «An overview of the introns-first theory». Journal of Molecular Evolution 69 (5): 527–40. November 2009. doi:. PMID 19777149.
- ↑ Cavalier-Smith, T (1985). «Selfish DNA and the origin of introns.». Nature 315 (6017): 283–4. doi:. PMID 2987701.
- ↑ «Origins and evolution of spliceosomal introns». Annu. Rev. Genet. 40: 47–76. 2006. doi:. PMID 17094737.
- ↑ «Eukaryotic intron loss». Science 300 (5624): 1393. May 2003. doi:. PMID 12775832.
- ↑ «The evolution of spliceosomal introns: patterns, puzzles and progress». Nature Reviews Genetics 7 (3): 211–21. March 2006. doi:. PMID 16485020.
- ↑ de Souza SJ (July 2003). «The emergence of a synthetic theory of intron evolution». Genetica 118 (2–3): 117–21. doi:. PMID 12868602.
- ↑ Lynch M (April 2002). «Intron evolution as a population-genetic process». Proceedings of the National Academy of Sciences 99 (9): 6118–23. doi:. PMID 11983904.
- ↑ «The biology of intron gain and loss». Trends in Genetics 22 (1): 16–22. January 2006. doi:. PMID 16290250. https://archive.org/details/sim_trends-in-genetics_2006-01_22_1/page/16.
- ↑ «Rapidly regulated genes are intron poor». Trends in Genetics 24 (8): 375–8. August 2008. doi:. PMID 18586348. https://archive.org/details/sim_trends-in-genetics_2008-08_24_8/page/375.
- ↑ «Selection for short introns in highly expressed genes». Nature Genetics 31 (4): 415–8. August 2002. doi:. PMID 12134150. https://zenodo.org/record/1233421.
- ↑ «Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability». Mol. Cell 67 (4): 608–621.e6. 2017. doi:. PMID 28757210.
